
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 18,294,653 – 18,294,744 |
| Length | 91 |
| Max. P | 0.866557 |

| Location | 18,294,653 – 18,294,744 |
|---|---|
| Length | 91 |
| Sequences | 4 |
| Columns | 93 |
| Reading direction | forward |
| Mean pairwise identity | 95.11 |
| Mean single sequence MFE | -34.05 |
| Consensus MFE | -29.09 |
| Energy contribution | -28.77 |
| Covariance contribution | -0.31 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.85 |
| SVM RNA-class probability | 0.866557 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
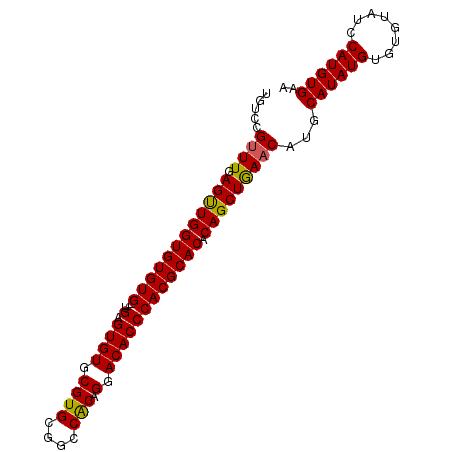
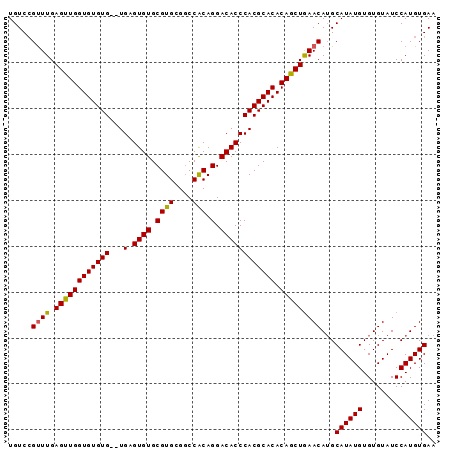
>3R_DroMel_CAF1 18294653 91 + 27905053 UGUCAGCUUGAGUUGGUGUGUG--UGAGUGUGCGUGCGGCCACAGGACACCCACGCACACAGCUGAACAUGCAUAUGUGUGUAUCCAUGUGAA ..((((((.......(((((((--((.((((.((((....))).).)))).)))))))))))))))(((((.((((....)))).)))))... ( -35.31) >DroSec_CAF1 7401 91 + 1 UGUCCGUUUGAGUUGGUGUGUG--UGAGUGUGCGUGCGGCCACAGGACACCCACGCACACAGCUGAACAUGCAUAUGUGUGUAUCCAUGUGAA .....((((.(((((.((((((--((.((((.((((....))).).)))).)))))))))))))))))...((((((........)))))).. ( -33.50) >DroSim_CAF1 6370 91 + 1 UGUCCGUUUGAGUUGGUGUGUG--UGAGUGUGCGUGCGGCCACAGGACACCCACGCACACAGCUGAACAUGCAUAUGUGUGUAUCCAUGUGAA .....((((.(((((.((((((--((.((((.((((....))).).)))).)))))))))))))))))...((((((........)))))).. ( -33.50) >DroEre_CAF1 6368 93 + 1 UGUCCGUUUGAGCUGGUGUGUGUUUGAGUGUGCGUGCGCCCGCAGGACACCCACGCACACAGCUAAACAAGCAUAUGUGUGUAUCCAUGUGAA .....((((.((((((((((((...(.((((.(.(((....))).))))))))))))).)))))))))...((((((........)))))).. ( -33.90) >consensus UGUCCGUUUGAGUUGGUGUGUG__UGAGUGUGCGUGCGGCCACAGGACACCCACGCACACAGCUGAACAUGCAUAUGUGUGUAUCCAUGUGAA .....((((.((((((((((((...(.((((.((((....))).).)))))))))))).)))))))))...((((((........)))))).. (-29.09 = -28.77 + -0.31)

| Location | 18,294,653 – 18,294,744 |
|---|---|
| Length | 91 |
| Sequences | 4 |
| Columns | 93 |
| Reading direction | reverse |
| Mean pairwise identity | 95.11 |
| Mean single sequence MFE | -29.38 |
| Consensus MFE | -23.76 |
| Energy contribution | -23.82 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.44 |
| Structure conservation index | 0.81 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.783059 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
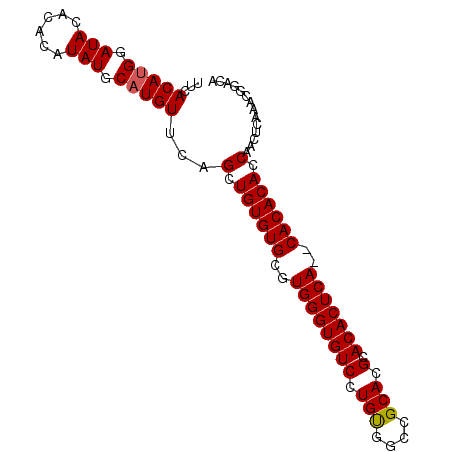
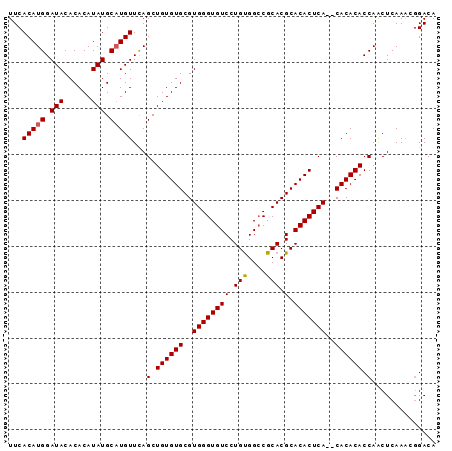
>3R_DroMel_CAF1 18294653 91 - 27905053 UUCACAUGGAUACACACAUAUGCAUGUUCAGCUGUGUGCGUGGGUGUCCUGUGGCCGCACGCACACUCA--CACACACCAACUCAAGCUGACA ...(((((.(((......))).)))))(((((((((((.(((((((((.(((....))).).)))))))--).))))).......)))))).. ( -33.81) >DroSec_CAF1 7401 91 - 1 UUCACAUGGAUACACACAUAUGCAUGUUCAGCUGUGUGCGUGGGUGUCCUGUGGCCGCACGCACACUCA--CACACACCAACUCAAACGGACA ((((((((.(((......))).)))))......(((((.(((((((((.(((....))).).)))))))--).)))))..........))).. ( -27.60) >DroSim_CAF1 6370 91 - 1 UUCACAUGGAUACACACAUAUGCAUGUUCAGCUGUGUGCGUGGGUGUCCUGUGGCCGCACGCACACUCA--CACACACCAACUCAAACGGACA ((((((((.(((......))).)))))......(((((.(((((((((.(((....))).).)))))))--).)))))..........))).. ( -27.60) >DroEre_CAF1 6368 93 - 1 UUCACAUGGAUACACACAUAUGCUUGUUUAGCUGUGUGCGUGGGUGUCCUGCGGGCGCACGCACACUCAAACACACACCAGCUCAAACGGACA .....(((........))).((..((((((((((.(((.(((..((...((((......))))....))..))).)))))))).)))))..)) ( -28.50) >consensus UUCACAUGGAUACACACAUAUGCAUGUUCAGCUGUGUGCGUGGGUGUCCUGUGGCCGCACGCACACUCA__CACACACCAACUCAAACGGACA ...(((((.(((......))).)))))...(.((((((..((((((((.(((....))).).)))))))..)))))).).............. (-23.76 = -23.82 + 0.06)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:33:43 2006