
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 18,284,036 – 18,284,143 |
| Length | 107 |
| Max. P | 0.570028 |

| Location | 18,284,036 – 18,284,143 |
|---|---|
| Length | 107 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.64 |
| Mean single sequence MFE | -45.05 |
| Consensus MFE | -27.28 |
| Energy contribution | -27.90 |
| Covariance contribution | 0.62 |
| Combinations/Pair | 1.26 |
| Mean z-score | -1.67 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.07 |
| SVM RNA-class probability | 0.570028 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
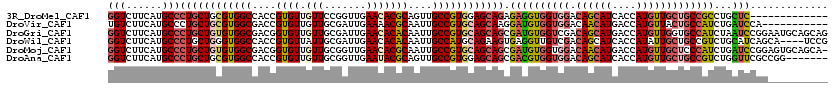
>3R_DroMel_CAF1 18284036 107 - 27905053 GGUCUUCAUGCCCUGCUGCGUGGCCACCGUGUUGUUCCGGUUGAACACGCAGUUGCCGUGGAGCAGAGAGGUGGUGGACAGCAUCACCAUGUUGCUGCCGCCUGCUC------------- .(.(((((((.(..((((((((..(((((........))).))..)))))))).).)))))))).(((((((((..(.((((((....)))))))..)))))).)))------------- ( -50.60) >DroVir_CAF1 19510 109 - 1 UGUCUUCAUGCCCUGCUGCGUGGCGACCGUGUUGUUGCGAUUGAAAACGCAAUUGCCGUGCAGCAAGGAUGUGGUGGACAACAUGACCAUGUUACUGCCAUCUGAUCCA----------- .............(((((((((((((..(((((.((......)).)))))..))))))))))))).((((..(((((..(((((....)))))....)))))..)))).----------- ( -39.00) >DroGri_CAF1 17200 120 - 1 GGUCUUCAUGCCCUGCUGUGUGGCGACGGUGUUGUUGCGAUUGAACACACAAUUGCCGUGCAGCAGCGAUGUGGUCGACAGCAUGACCAUGUUGGUGCCAUCUAAUCCGGAAUGCAGCAG ........(((.((((((..((((((..((((.(((.......)))))))..))))))..))))))(((((((((((......))))))))))).(((..(((.....)))..)))))). ( -45.80) >DroWil_CAF1 37610 116 - 1 GGUCUUCAUGCCCUGCUGGGUGGCCACCGUGUUAUUGCGAUUGAACACACAAUUGCCAUGCAGAAGUGAGGUUGUCGACAGCAUCACCAUAUUGCUGCCGUCUGCAUCAGCA----UCCG ((((.....((((....))))))))...(((((...(((((((......))))))).(((((((.((((.((((....)))).))))((......))...))))))).))))----)... ( -33.90) >DroMoj_CAF1 18666 119 - 1 GGUCUUCAUGCCCUGCUGUGUGGCGACGGUGUUGUUGCGGUUGAACACGCAAUUGCCGUGCAGCAGCGAUGUGGUGGACAACAUGACCAUGUUGCUCCCAUCUGAUCCGGAGUGCAGCA- ((((..(((..(((((((..((((((..((((.(((.......)))))))..))))))..)))))).)..)))(((.....))))))).((((((.(((.........)).).))))))- ( -47.50) >DroAna_CAF1 15358 113 - 1 GGUCUUCAUGCCCUGCUGCGUGGCCACCGUGUUGUUGCGGUUGAAUACGCAGUUGCCGUGGAGCAGCGACGUGGUGGACAGCAUCACCAUGUUGCUGCCGUCUGGUUCGCCGG------- ....((((((.(..((((((((..((((((......)))).))..)))))))).).))))))(((((((((((((((......)))))))))))))))...((((....))))------- ( -53.50) >consensus GGUCUUCAUGCCCUGCUGCGUGGCCACCGUGUUGUUGCGAUUGAACACGCAAUUGCCGUGCAGCAGCGAUGUGGUGGACAGCAUCACCAUGUUGCUGCCAUCUGAUCCGG__________ (((......)))((((((((((((....((((.(((.......)))))))....)))))))))))).((((((((((.((((((....)))))))))))))...)))............. (-27.28 = -27.90 + 0.62)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:33:38 2006