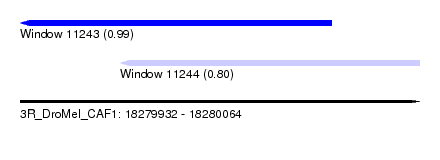
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 18,279,932 – 18,280,064 |
| Length | 132 |
| Max. P | 0.987003 |

| Location | 18,279,932 – 18,280,035 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 73.78 |
| Mean single sequence MFE | -28.75 |
| Consensus MFE | -18.05 |
| Energy contribution | -18.75 |
| Covariance contribution | 0.70 |
| Combinations/Pair | 1.26 |
| Mean z-score | -2.19 |
| Structure conservation index | 0.63 |
| SVM decision value | 2.06 |
| SVM RNA-class probability | 0.987003 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
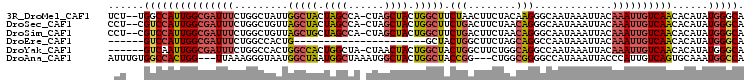
>3R_DroMel_CAF1 18279932 103 - 27905053 UCU--UGGCCAUUGGCGAUUUCUGGCUAUUGGCUACUAGCCA-CUAGCUACUGGCUUCUAACUUCUACAAGGGCAAUAAAUUACAAAUUGUCAACACAUAUGGGCA ...--..(((.((((((((((.((((((.((((.....))))-.))))))...(((..(.........)..)))..........))))))))))((....))))). ( -26.60) >DroSec_CAF1 5505 103 - 1 CCU--CGUCCAUUGGCGAUUUCUGGCUGUUAGCUACUAGCCA-CUAGCUACUGGCUUCUGACUUCUAACAGGGCAAUAAAUUACAAAUUGUCAACACAUAUGGGCA ...--.((((((((...........(((((((.....(((((-........)))))........)))))))((((((.........))))))....)).)))))). ( -26.22) >DroSim_CAF1 6255 103 - 1 CCU--CGUCCAUUGGCGAUUUCUGGCUGUUAGCUGCUAGCCA-CUAGCUACUGGCUUCUGACUUCUAACAGGGCAAUAAAUUACAAAUUGUCAACACAUAUGGGCA ...--.((((((((...........(((((((..((.(((((-........)))))...).)..)))))))((((((.........))))))....)).)))))). ( -27.80) >DroEre_CAF1 11540 78 - 1 ------GUCCAUUGGCGAUUUCUGGCCACUG----------------------GCUACUGGCUUCUAGCAGGCCAAUAAAUUACAAAUUGUCAACACAUAUGGGCA ------(((((((((((((((.(((((...)----------------------)))).((((((.....)))))).........)))))))))......)))))). ( -24.40) >DroYak_CAF1 5558 99 - 1 ------GUCAAUUGGCGAUUUCUGGCCACUGGCCACUGGCUA-CUAACUACUGGCUACUGGCUUCUGGCAGGCCAAUAAAUUACAAAUUGUCAACACAUAUGGGCA ------(((.(((((((....)..((((..(((((.((((((-........)))))).)))))..))))..))))))......((...(((....)))..))))). ( -31.20) >DroAna_CAF1 11008 100 - 1 AUUUGUGGCCACUGG---UUAAAGGGUAAUGGCUAAUGGCUAAAUGGCUACUGGCUACCGG---CUGGCGGGGCCAUAAAUUACCCAUUGUCAGUGCAAAUGGCCA .....((((((....---.....((((((((((((.(((((....))))).)))))...((---((.....))))....))))))).((((....)))).)))))) ( -36.30) >consensus _CU___GUCCAUUGGCGAUUUCUGGCUAUUGGCUACUAGCCA_CUAGCUACUGGCUACUGACUUCUAGCAGGGCAAUAAAUUACAAAUUGUCAACACAUAUGGGCA ......(((((((((((((((.........(((((.((((......)))).))))).(((........))).............))))))))))......))))). (-18.05 = -18.75 + 0.70)
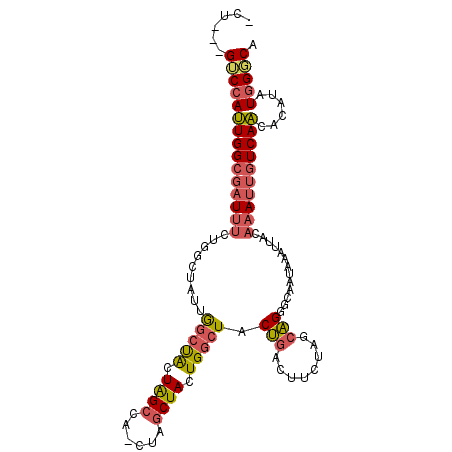
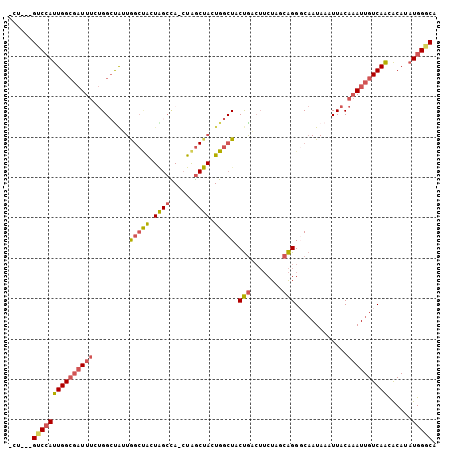
| Location | 18,279,965 – 18,280,064 |
|---|---|
| Length | 99 |
| Sequences | 5 |
| Columns | 106 |
| Reading direction | reverse |
| Mean pairwise identity | 76.92 |
| Mean single sequence MFE | -32.92 |
| Consensus MFE | -19.81 |
| Energy contribution | -19.89 |
| Covariance contribution | 0.08 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.83 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.61 |
| SVM RNA-class probability | 0.800214 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 18279965 99 - 27905053 GGAUUAGCCCAGCUUAGUCGAUUGCCAC----UUCU--UGGCCAUUGGCGAUUUCUGGCUAUUGGCUACUAGCCA-CUAGCUACUGGCUUCUAACUUCUACAAGGG (((((((.((((....((((((.((((.----....--)))).))))))......((((((.((((.....))))-.))))))))))...)))).)))........ ( -31.90) >DroSec_CAF1 5538 99 - 1 GGAUUAGCCCAGCUUAGUCGAUUGCCAC----UCCU--CGUCCAUUGGCGAUUUCUGGCUGUUAGCUACUAGCCA-CUAGCUACUGGCUUCUGACUUCUAACAGGG .......(((.....(((((((.((((.----...(--((((....)))))....)))).)))(((((.((((..-...)))).)))))...)))).......))) ( -27.00) >DroSim_CAF1 6288 99 - 1 GGAUUAGCCCAGCUUAGUCGAUUGCCAC----UCCU--CGUCCAUUGGCGAUUUCUGGCUGUUAGCUGCUAGCCA-CUAGCUACUGGCUUCUGACUUCUAACAGGG .......(((.....(((((...((((.----...(--((((....)))))....)))).(((((..(((((...-)))))..)))))...))))).......))) ( -27.50) >DroYak_CAF1 5591 94 - 1 GGAUUAGCCCAGCUUAGUCGAUUGCCAC-----------GUCAAUUGGCGAUUUCUGGCCACUGGCCACUGGCUA-CUAACUACUGGCUACUGGCUUCUGGCAGGC ......((((((....((((((((....-----------..)))))))).....)))((((..(((((.((((((-........)))))).)))))..)))).))) ( -35.50) >DroAna_CAF1 11041 100 - 1 GGAUUAGUCCAGCUUAGCCGAUAGCCAGCCACUAUUUGUGGCCACUGG---UUAAAGGGUAAUGGCUAAUGGCUAAAUGGCUACUGGCUACCGG---CUGGCGGGG (((....))).(((.(((((.(((((((...(((((..((((((.(((---(((........)))))).)))))))))))...))))))).)))---))))).... ( -42.70) >consensus GGAUUAGCCCAGCUUAGUCGAUUGCCAC____UCCU__CGUCCAUUGGCGAUUUCUGGCUAUUGGCUACUAGCCA_CUAGCUACUGGCUUCUGACUUCUAACAGGG .......(((....(((((((((((((..................))))))))...)))))..(((((.((((......)))).)))))..............))) (-19.81 = -19.89 + 0.08)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:33:32 2006