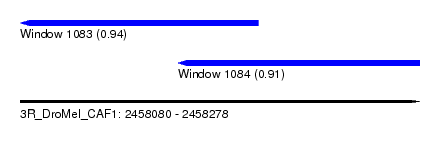
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 2,458,080 – 2,458,278 |
| Length | 198 |
| Max. P | 0.937144 |

| Location | 2,458,080 – 2,458,198 |
|---|---|
| Length | 118 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.82 |
| Mean single sequence MFE | -42.83 |
| Consensus MFE | -37.60 |
| Energy contribution | -37.73 |
| Covariance contribution | 0.13 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.32 |
| Structure conservation index | 0.88 |
| SVM decision value | 1.26 |
| SVM RNA-class probability | 0.937144 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 2458080 118 - 27905053 UGCCCAAUUUGUUGCUGCCCUUUUGCGUUUUUGCUCUGUGGCAUUGUUUUUUGGUGG--GAGCACACUCGAGCCACAAAAGCGAAGAAAAAAAGGAGCAGGGAUCGUUGGCCCUCGCAAU ((((((((..(((.((((((((((...((((((((.((((((.(((.......(((.--...)))...)))))))))..))))))))..)))))).)))).))).))))).....))).. ( -43.40) >DroSec_CAF1 17091 120 - 1 UGCCCAAUUUGUUGCUGCCCUUUUGCGUUUUUGCUCUGUGGCAUUGUUUUUUGGUGGGGGAGCACACUCGAGCCACAAAAGCGAAGGAAAAAAGGAGCAGGGAUCGUUGGCCCUCGCAAU ((((((((..(((.((((((((((...((((((((.((((((.(((......((((.(....).)))))))))))))..))))))))..)))))).)))).))).))))).....))).. ( -42.10) >DroSim_CAF1 13815 118 - 1 UGCCCAAUUUGUUGCUGCCCUUUUGCGUUUUUGCUCUGUGGCAUUGUUUUUUGGUGG--GAGCACACUCGAGCCACAAAAGCAAAGGAAAAAAGGAGCAGGGAUCGUUGGCCCUCGCAAU ((((((((..(((.((((((((((...((((((((.((((((.(((.......(((.--...)))...)))))))))..))))))))..)))))).)))).))).))))).....))).. ( -42.90) >DroEre_CAF1 18577 118 - 1 UGCCCAAUUUGUUGCUGCCCUUUUGCGUUUUUGCUCUGUGGCAUUGUUUUUCGGUGG--AAGCACAUUCCAGCCACAAAAGCAAAGGAAAAAAGGGCCAGGGAUCGGUGGCCCUCGGAAU .(((..((..(((.((((((((((...((((((((.((((((((((.....))))((--((.....)))).))))))..))))))))..))))))).))).)))..)))))......... ( -42.90) >consensus UGCCCAAUUUGUUGCUGCCCUUUUGCGUUUUUGCUCUGUGGCAUUGUUUUUUGGUGG__GAGCACACUCGAGCCACAAAAGCAAAGGAAAAAAGGAGCAGGGAUCGUUGGCCCUCGCAAU ...(((((..(((.((((((((((...((((((((.((((((.(((......((((........)))))))))))))..))))))))..)))))).)))).))).))))).......... (-37.60 = -37.73 + 0.13)

| Location | 2,458,158 – 2,458,278 |
|---|---|
| Length | 120 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 97.08 |
| Mean single sequence MFE | -36.53 |
| Consensus MFE | -35.00 |
| Energy contribution | -35.75 |
| Covariance contribution | 0.75 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.06 |
| Structure conservation index | 0.96 |
| SVM decision value | 1.05 |
| SVM RNA-class probability | 0.906458 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 2458158 120 - 27905053 AUAAAUGGACUGACGAACGCGAAAGGAUAACAGCAGGAUAUAUCGUUCAGGAAUACAAAUGUGGGGAGCAUGUUUGUUUUUGCCCAAUUUGUUGCUGCCCUUUUGCGUUUUUGCUCUGUG ....(((((..((.((((((((((((....(((((((((.....))))......((((((.(((((((((....)))))...))))))))))))))).))))))))))))))..))))). ( -36.60) >DroSec_CAF1 17171 120 - 1 AUAAAUGGACUGACGAACGCGAAAGGAUAACAGCAGGAUAUAUCGUUCAGGAAUACAAAUGUGGGUAGCAUGUUUGUUUUUGCCCAAUUUGUUGCUGCCCUUUUGCGUUUUUGCUCUGUG ....(((((..((.((((((((((((....(((((((((.....))))......((((((.(((((((((....)))...))))))))))))))))).))))))))))))))..))))). ( -37.70) >DroSim_CAF1 13893 114 - 1 ------GGACUGACGAACGCGAAAGGAUAACAGCAGGAUAUAUCGUUCAGGAAUACAAAUGUGGGGAGCAUGUUUGUUUUUGCCCAAUUUGUUGCUGCCCUUUUGCGUUUUUGCUCUGUG ------(((..((.((((((((((((....(((((((((.....))))......((((((.(((((((((....)))))...))))))))))))))).))))))))))))))..)))... ( -35.20) >DroEre_CAF1 18655 120 - 1 AUAAAUGGACUGACGAACGCGAAAGGAUAACAGCAGGAUAUAUCGUUCAGGAAUACAAAUGUGGGGAGCAUGUUUGUUUUUGCCCAAUUUGUUGCUGCCCUUUUGCGUUUUUGCUCUGUG ....(((((..((.((((((((((((....(((((((((.....))))......((((((.(((((((((....)))))...))))))))))))))).))))))))))))))..))))). ( -36.60) >consensus AUAAAUGGACUGACGAACGCGAAAGGAUAACAGCAGGAUAUAUCGUUCAGGAAUACAAAUGUGGGGAGCAUGUUUGUUUUUGCCCAAUUUGUUGCUGCCCUUUUGCGUUUUUGCUCUGUG ....(((((..((.((((((((((((....(((((((((.....))))......((((((.(((((((((....)))))...))))))))))))))).))))))))))))))..))))). (-35.00 = -35.75 + 0.75)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:49:17 2006