
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 17,938,473 – 17,938,647 |
| Length | 174 |
| Max. P | 0.980161 |

| Location | 17,938,473 – 17,938,577 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | forward |
| Mean pairwise identity | 90.56 |
| Mean single sequence MFE | -21.10 |
| Consensus MFE | -16.88 |
| Energy contribution | -17.24 |
| Covariance contribution | 0.36 |
| Combinations/Pair | 1.04 |
| Mean z-score | -3.83 |
| Structure conservation index | 0.80 |
| SVM decision value | 1.85 |
| SVM RNA-class probability | 0.980161 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
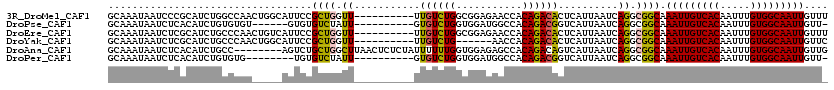
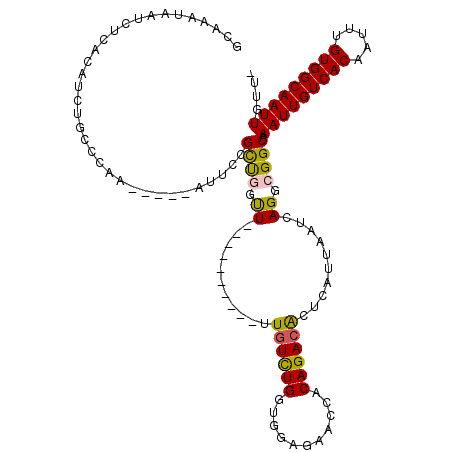
>3R_DroMel_CAF1 17938473 104 + 27905053 AUCAGCGACC-GCAACAAAAACAAUUAAGCAAAAUUGUCAUAAUUACAGCAGCACAAAUACAGGCAAACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUA (((((((...-((...............)).((((((((((((((...(.....).......(((((....)))))...))))))))))))))..)).))))).. ( -24.06) >DroPse_CAF1 16873 102 + 1 GACAACGAAAAA--AAAAAAACAAUUACGCAAAAUUGUCAUAAUUACAGCAUCCCAAACACAGAC-AACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUA ............--........(((((.((.((((((((((((((...(((..............-......)))....))))))))))))))...)).))))). ( -18.65) >DroEre_CAF1 17344 104 + 1 AUCAGCGACC-GCAACAAAAACAAUUAAGCAAAAUUGUCAUAAUUACAGUAGCACAAAUACAGGCAAACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUA (((((((...-((...............)).((((((((((((((...(((.......))).(((((....)))))...))))))))))))))..)).))))).. ( -27.26) >DroYak_CAF1 12535 104 + 1 AUCAGCGACC-GCAACAAAAACAAUUAAGCAAAAUUGUCAUAAUUACAGCAGCACAAAUACAGGCGAACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUA (((((((...-((...............)).((((((((((((((...(.....).......(((((....)))))...))))))))))))))..)).))))).. ( -23.76) >DroAna_CAF1 15123 104 + 1 AACGACAACC-GCAACAAAAACAAUUAAGCAAAAUGGUCAUAAUUACAGCAGCACAAACACAGACCAACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUA ..........-...........(((((.((.((((.(((((((((...((((...................))))....))))))))).))))...)).))))). ( -14.21) >DroPer_CAF1 20626 104 + 1 GACAACGAAAAACAAAAAAAACAAUUACGCAAAAUUGUCAUAAUUACAGCAUCACAAACACAGAC-AACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUA ......................(((((.((.((((((((((((((...(((..............-......)))....))))))))))))))...)).))))). ( -18.65) >consensus AACAACGACC_GCAACAAAAACAAUUAAGCAAAAUUGUCAUAAUUACAGCAGCACAAACACAGAC_AACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUA ......................(((((.((.((((((((((((((...((((...................))))....))))))))))))))...)).))))). (-16.88 = -17.24 + 0.36)


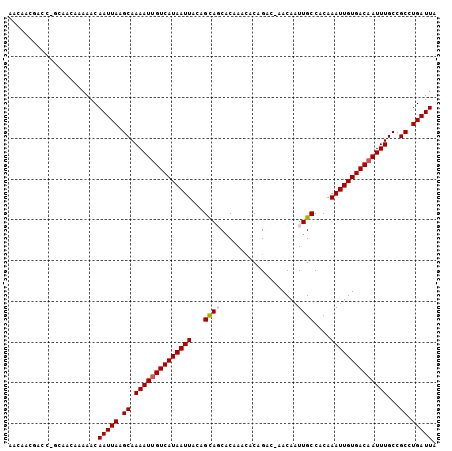
| Location | 17,938,473 – 17,938,577 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 105 |
| Reading direction | reverse |
| Mean pairwise identity | 90.56 |
| Mean single sequence MFE | -32.61 |
| Consensus MFE | -24.48 |
| Energy contribution | -25.45 |
| Covariance contribution | 0.97 |
| Combinations/Pair | 1.12 |
| Mean z-score | -4.29 |
| Structure conservation index | 0.75 |
| SVM decision value | 1.54 |
| SVM RNA-class probability | 0.962180 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 17938473 104 - 27905053 UAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUUGCCUGUAUUUGUGCUGCUGUAAUUAUGACAAUUUUGCUUAAUUGUUUUUGUUGC-GGUCGCUGAU ....(((((((..(((((((((.....)))))))))..))))))).......((.((((((((...(((((((......)))))))...)))))-))).)).... ( -37.80) >DroPse_CAF1 16873 102 - 1 UAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUU-GUCUGUGUUUGGGAUGCUGUAAUUAUGACAAUUUUGCGUAAUUGUUUUUUU--UUUUUCGUUGUC ....((((((((((.(((((((.....))))))))))))-))))).......((((..........(((((((......))))))).....--.....))))... ( -26.45) >DroEre_CAF1 17344 104 - 1 UAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUUGCCUGUAUUUGUGCUACUGUAAUUAUGACAAUUUUGCUUAAUUGUUUUUGUUGC-GGUCGCUGAU ....(((((((..(((((((((.....)))))))))..))))))).......((.((((((((...(((((((......)))))))...)))))-))).)).... ( -37.50) >DroYak_CAF1 12535 104 - 1 UAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUCGCCUGUAUUUGUGCUGCUGUAAUUAUGACAAUUUUGCUUAAUUGUUUUUGUUGC-GGUCGCUGAU ....(((((((..(((((((((.....)))))))))..))))))).......((.((((((((...(((((((......)))))))...)))))-))).)).... ( -39.80) >DroAna_CAF1 15123 104 - 1 UAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUGGUCUGUGUUUGUGCUGCUGUAAUUAUGACCAUUUUGCUUAAUUGUUUUUGUUGC-GGUUGUCGUU ....((((((((((.(((((((.....)))))))))))).))))).......((.((((((((...(((.(((......))).)))...)))))-))).)).... ( -28.10) >DroPer_CAF1 20626 104 - 1 UAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUU-GUCUGUGUUUGUGAUGCUGUAAUUAUGACAAUUUUGCGUAAUUGUUUUUUUUGUUUUUCGUUGUC ....((((((((((.(((((((.....))))))))))))-))))).....(((((......)))))(((((((......)))))))................... ( -26.00) >consensus UAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUU_GCCUGUAUUUGUGCUGCUGUAAUUAUGACAAUUUUGCUUAAUUGUUUUUGUUGC_GGUCGCUGAU ....((((((((((.(((((((.....)))))))))))).))))).......((.((((((((...(((((((......)))))))...))))).))).)).... (-24.48 = -25.45 + 0.97)

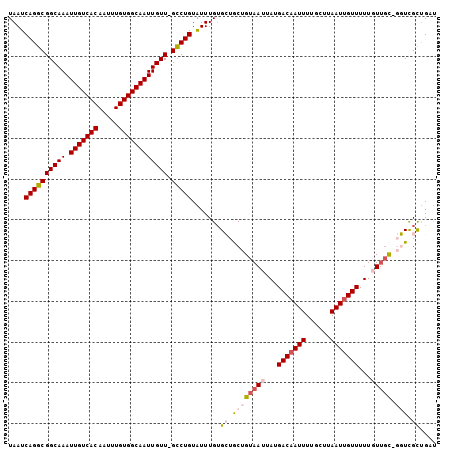
| Location | 17,938,497 – 17,938,607 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 87.88 |
| Mean single sequence MFE | -29.08 |
| Consensus MFE | -20.82 |
| Energy contribution | -20.93 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.08 |
| Mean z-score | -3.73 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.55 |
| SVM RNA-class probability | 0.779865 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
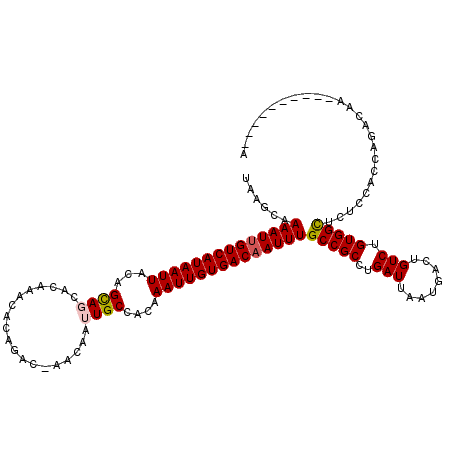
>3R_DroMel_CAF1 17938497 110 + 27905053 UAAGCAAAAUUGUCAUAAUUACAGCAGCACAAAUACAGGCAAACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUAAUGAGUGUCUGUGGUUCUCCGCCAGACAA----------A ...(((((.(((((((((((...(.....).......(((((....)))))...))))))))))))))))...............(((((((((....))).)))))).----------. ( -30.90) >DroPse_CAF1 16896 109 + 1 UACGCAAAAUUGUCAUAAUUACAGCAUCCCAAACACAGAC-AACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUAAUGACCGUCUGUGGCCAUCCACCAGACAC----------A ...(((((.(((((((((((...(((..............-......)))....))))))))))))))))................((((((((....))).)))))..----------. ( -26.15) >DroEre_CAF1 17368 110 + 1 UAAGCAAAAUUGUCAUAAUUACAGUAGCACAAAUACAGGCAAACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUAAUGAGUGUCUGUGGUUCUCCGCCAGACAA----------A ...(((((.(((((((((((...(((.......))).(((((....)))))...))))))))))))))))...............(((((((((....))).)))))).----------. ( -34.10) >DroYak_CAF1 12559 104 + 1 UAAGCAAAAUUGUCAUAAUUACAGCAGCACAAAUACAGGCGAACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUAAUGAGUGUCUGUGGUU------CAGACAA----------A .........(((((..((((((((((.((..(((.((((((...(((((.(((.....))).)))))...)))))))))..))..)).))))))))------..)))))----------. ( -32.10) >DroAna_CAF1 15147 120 + 1 UAAGCAAAAUGGUCAUAAUUACAGCAGCACAAACACAGACCAACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUAAUGACUGUCUGUGGCUCUCCACCAAAAAAUAGAGAGUUAA ...(((..(((((((((((((..((.(((...............(((((.(((.....))).)))))))).)).))))).)))))))).)))(((((((............))))))).. ( -25.06) >DroPer_CAF1 20651 109 + 1 UACGCAAAAUUGUCAUAAUUACAGCAUCACAAACACAGAC-AACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUAAUGACCGUCUGUGGCCAUCCACCAGACAC----------A ...(((((.(((((((((((...(((..............-......)))....))))))))))))))))................((((((((....))).)))))..----------. ( -26.15) >consensus UAAGCAAAAUUGUCAUAAUUACAGCAGCACAAACACAGAC_AACAAUUGCCACAAAUUGUGACAAUUUGCCGCCUGAUUAAUGACUGUCUGUGGCUCUCCACCAGACAA__________A ......((((((((((((((...((((...................))))....))))))))))))))(((((..(((........))).)))))......................... (-20.82 = -20.93 + 0.11)
| Location | 17,938,497 – 17,938,607 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.88 |
| Mean single sequence MFE | -35.65 |
| Consensus MFE | -26.47 |
| Energy contribution | -26.50 |
| Covariance contribution | 0.03 |
| Combinations/Pair | 1.09 |
| Mean z-score | -3.48 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.61 |
| SVM RNA-class probability | 0.799582 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 17938497 110 - 27905053 U----------UUGUCUGGCGGAGAACCACAGACACUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUUGCCUGUAUUUGUGCUGCUGUAAUUAUGACAAUUUUGCUUA .----------(((((....((....))((((.((..(((.((((((((((..(((((((((.....)))))))))..))))))).))).))).))))))......)))))......... ( -33.20) >DroPse_CAF1 16896 109 - 1 U----------GUGUCUGGUGGAUGGCCACAGACGGUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUU-GUCUGUGUUUGGGAUGCUGUAAUUAUGACAAUUUUGCGUA .----------.((((((((.(((((((......))))))).))))))))((((((((((((.....)))))))))(((-(((...((((((....))).)))...))))))..)))... ( -37.20) >DroEre_CAF1 17368 110 - 1 U----------UUGUCUGGCGGAGAACCACAGACACUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUUGCCUGUAUUUGUGCUACUGUAAUUAUGACAAUUUUGCUUA .----------.((((((..((....)).))))))..(((.((((((((((..(((((((((.....)))))))))..))))))).))).))).....((((...........))))... ( -32.20) >DroYak_CAF1 12559 104 - 1 U----------UUGUCUG------AACCACAGACACUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUCGCCUGUAUUUGUGCUGCUGUAAUUAUGACAAUUUUGCUUA .----------(((((..------....((((.((..(((.((((((((((..(((((((((.....)))))))))..))))))).))).))).))))))......)))))......... ( -34.60) >DroAna_CAF1 15147 120 - 1 UUAACUCUCUAUUUUUUGGUGGAGAGCCACAGACAGUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUGGUCUGUGUUUGUGCUGCUGUAAUUAUGACCAUUUUGCUUA ....(((((((((....)))))))))..((((.(((.(((.(((((((((((((.(((((((.....)))))))))))).))))).))).)))))))))).................... ( -38.80) >DroPer_CAF1 20651 109 - 1 U----------GUGUCUGGUGGAUGGCCACAGACGGUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUU-GUCUGUGUUUGUGAUGCUGUAAUUAUGACAAUUUUGCGUA .----------.((((((.(((....)))))))))(((((.(((((((((((((.(((((((.....))))))))))))-))))).))).)))))((...((((.....))))..))... ( -37.90) >consensus U__________UUGUCUGGUGGAGAACCACAGACACUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUU_GCCUGUAUUUGUGCUGCUGUAAUUAUGACAAUUUUGCUUA ............((((.((.......))((((.....(((.(((((((((((((.(((((((.....)))))))))))).))))).))).)))...))))......)))).......... (-26.47 = -26.50 + 0.03)

| Location | 17,938,537 – 17,938,647 |
|---|---|
| Length | 110 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.61 |
| Mean single sequence MFE | -35.02 |
| Consensus MFE | -19.66 |
| Energy contribution | -19.22 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.24 |
| Mean z-score | -3.14 |
| Structure conservation index | 0.56 |
| SVM decision value | 1.72 |
| SVM RNA-class probability | 0.973807 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
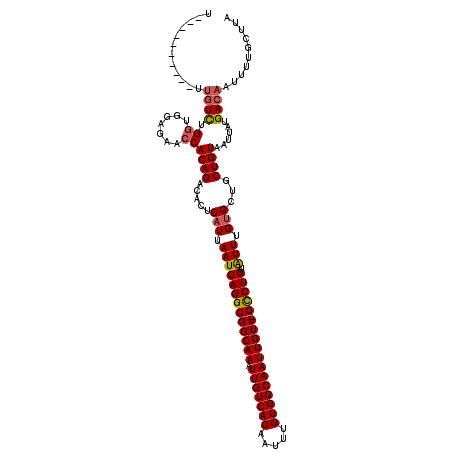
>3R_DroMel_CAF1 17938537 110 - 27905053 GCAAAUAAUCCCGCAUCUGGCCAACUGGCAUUCCGCUGGUU----------UUGUCUGGCGGAGAACCACAGACACUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUU ..........((((.(((((((....))).((((((..(..----------....)..)))))).....))))..((........))))))..(((((((((.....))))))))).... ( -37.00) >DroPse_CAF1 16936 103 - 1 GCAAAUAAUCUCACAUCUGUGUGU------GUGUGUCUAUU----------GUGUCUGGUGGAUGGCCACAGACGGUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUU- ((.((((..(.(((((......))------))).)..))))----------.((((((((.(((((((......))))))).)))))))))).(((((((((.....)))))))))...- ( -36.90) >DroEre_CAF1 17408 110 - 1 GCAAAUAAUCUCGCAUCUGCCCAACUGUCAUUCCGCUGGUU----------UUGUCUGGCGGAGAACCACAGACACUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUU ((..........))...(((((..((((..((((((..(..----------....)..))))))....))))...((........))).))))(((((((((.....))))))))).... ( -30.30) >DroYak_CAF1 12599 104 - 1 GCAAAUAAUCUCGCAUCUGCCCAACUGGCAUUCCGCUGGUU----------UUGUCUG------AACCACAGACACUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUC ((..........))..(((((.(((..((.....))..)))----------.((((((------.....))))))...........)))))..(((((((((.....))))))))).... ( -30.30) >DroAna_CAF1 15187 112 - 1 GCAAAUAAUCUCACAUCUGCC--------AGUCUGCUGGCUUAACUCUCUAUUUUUUGGUGGAGAGCCACAGACAGUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUUG .................((((--------.(((((.((((.....((((((((....))))))))))))))))).(((........)))))))(((((((((.....))))))))).... ( -38.90) >DroPer_CAF1 20691 101 - 1 GCAAAUAAUCUCACAUCUGUGUG--------UGUGUCUAUU----------GUGUCUGGUGGAUGGCCACAGACGGUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUU- ((.((((..(.(((((....)))--------)).)..))))----------.((((((((.(((((((......))))))).)))))))))).(((((((((.....)))))))))...- ( -36.70) >consensus GCAAAUAAUCUCACAUCUGCCCAA_____AUUCCGCUGGUU__________UUGUCUGGUGGAGAACCACAGACACUCAUUAAUCAGGCGGCAAAUUGUCACAAUUUGUGGCAAUUGUU_ ..................................((((.((...........((((((...........))))))..........)).)))).(((((((((.....))))))))).... (-19.66 = -19.22 + -0.44)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:30:02 2006