
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 17,623,947 – 17,624,075 |
| Length | 128 |
| Max. P | 0.980503 |

| Location | 17,623,947 – 17,624,046 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 81.70 |
| Mean single sequence MFE | -39.80 |
| Consensus MFE | -35.33 |
| Energy contribution | -36.00 |
| Covariance contribution | 0.67 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.84 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.86 |
| SVM RNA-class probability | 0.980503 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
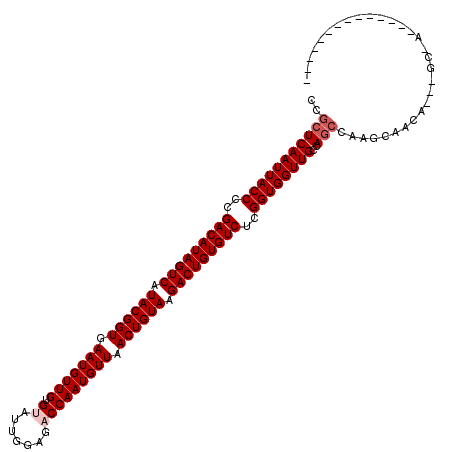
>3R_DroMel_CAF1 17623947 99 - 27905053 CCGCUCAAUUACCCCGACAUAGUCAUACGGUGAAUGUUGUGUAUUGGAGACCAAUGUUAACUGUAAGACUGUGUCUCGGUGGUUGCCAGCCCAGCCACA--------------------- ..(((((((((((..(((((((((.((((((.(((((((.((.......))))))))).)))))).)))))))))..))))))))..))).........--------------------- ( -38.20) >DroVir_CAF1 30589 102 - 1 CCGCUCAAUUACCGUGACAUAGUCGUACGGUGAAUGUUGUGUAU-GGAGUCCAAUGUUAACUGUAAGACUGUGUCUCGGUGGUUGUUAGCUAAUCAACA---GCGA-------------- ..((((((((((((.(((((((((.((((((.(((((((.(...-.....)))))))).)))))).))))))))).)))))))))..))).........---....-------------- ( -41.80) >DroGri_CAF1 20053 106 - 1 UCGCUCAAUUACCAUGACAUAGUCGUACGGUGAAUGUUGUGUUU-GGAGUCCAAUGUUAACUGUAAGACUGUGUCUUGGUGGUUGCCAGCCAAGCAACA--AGCGAUGA----------- (((((....(((((.(((((((((.((((((.(((((((..(..-...)..))))))).)))))).))))))))).)))))(((((.......))))).--)))))...----------- ( -43.20) >DroEre_CAF1 18652 118 - 1 CCGCUCAAUUACCCCGACAUAGUCAUACGGUGAAUGUUGUGUAUUGGAGACCAAUGUUAACUGUAAGACUGUGUCUCGGUGGUUGCCAGCCCAGCCACACCAGCCAC-ACCAGCCACAG- ..(((((((((((..(((((((((.((((((.(((((((.((.......))))))))).)))))).)))))))))..))))))))..))).................-...........- ( -38.20) >DroYak_CAF1 22455 114 - 1 CCGCUCAAUUACCCCGACAUAGUCAUACGGUGAAUGUUGUGUAUUGGAGACCAAUGUUAACUGUAAGACUGUGUCUCGGUGGUUGCCA-----GCCACACCAGCCAC-AUCAGCCACAUC ..(((((((((((..(((((((((.((((((.(((((((.((.......))))))))).)))))).)))))))))..))))))))..)-----))............-............ ( -38.20) >DroPer_CAF1 10264 97 - 1 CCGCUCAAUUACCAUGACAUAGUCGUACGGUGAAUGUUGUGUAUUGG--ACCAAUGUUAACUGUAAGACUGUGUCUCGGUGGUUGUCAGCCAAGCAGCA--------------------- ..(((((((((((..(((((((((.((((((.(((((((.((.....--))))))))).)))))).)))))))))..))))))))..))).........--------------------- ( -39.20) >consensus CCGCUCAAUUACCCCGACAUAGUCAUACGGUGAAUGUUGUGUAUUGGAGACCAAUGUUAACUGUAAGACUGUGUCUCGGUGGUUGCCAGCCAAGCAACA___GC_A______________ ..(((((((((((..(((((((((.((((((.(((((((.((.......))))))))).)))))).)))))))))..))))))))..))).............................. (-35.33 = -36.00 + 0.67)

| Location | 17,623,966 – 17,624,075 |
|---|---|
| Length | 109 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 78.90 |
| Mean single sequence MFE | -34.70 |
| Consensus MFE | -26.75 |
| Energy contribution | -26.75 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.55 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.63 |
| SVM RNA-class probability | 0.968994 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
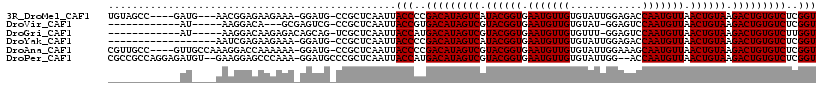
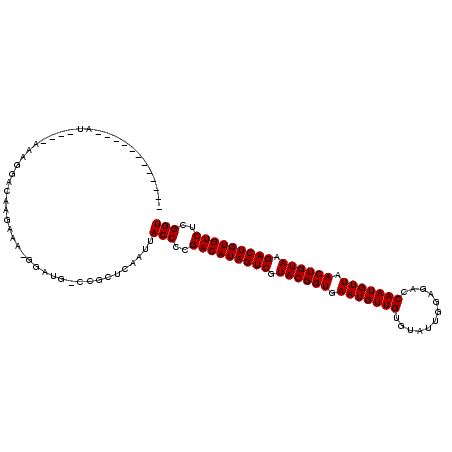
>3R_DroMel_CAF1 17623966 109 - 27905053 UGUAGCC----GAUG---AACGGAGAAGAAA-GGAUG-CCGCUCAAUUACCCCGACAUAGUCAUACGGUGAAUGUUGUGUAUUGGAGACCAAUGUUAACUGUAAGACUGUGUCUCGGU ....(((----((((---(.(((........-.....-))).)))........(((((((((.((((((.(((((((.((.......))))))))).)))))).)))))))))))))) ( -36.72) >DroVir_CAF1 30612 96 - 1 ------------AU-----AAGGACA---GCGAGUCG-CCGCUCAAUUACCGUGACAUAGUCGUACGGUGAAUGUUGUGUAU-GGAGUCCAAUGUUAACUGUAAGACUGUGUCUCGGU ------------..-----......(---(((.....-.)))).....((((.(((((((((.((((((.(((((((.(...-.....)))))))).)))))).))))))))).)))) ( -35.80) >DroGri_CAF1 20080 99 - 1 ------------AU-----AAGGACAAGAGACAGCAG-UCGCUCAAUUACCAUGACAUAGUCGUACGGUGAAUGUUGUGUUU-GGAGUCCAAUGUUAACUGUAAGACUGUGUCUUGGU ------------..-----.......((.(((....)-)).)).....((((.(((((((((.((((((.(((((((..(..-...)..))))))).)))))).))))))))).)))) ( -32.90) >DroYak_CAF1 22489 98 - 1 ------------------AAUCGAGAAGAAA-GGAUG-CCGCUCAAUUACCCCGACAUAGUCAUACGGUGAAUGUUGUGUAUUGGAGACCAAUGUUAACUGUAAGACUGUGUCUCGGU ------------------.(((((...((..-((...-))..)).........(((((((((.((((((.(((((((.((.......))))))))).)))))).)))))))))))))) ( -30.80) >DroAna_CAF1 27567 112 - 1 CGUUGCC----GUUGCCAAAGGACCAAAAAA-GGAUG-CCGCUCAAUUACCCCGACAUAGUCGUACGGUGAAUGUUGUGUAUUGGAAAGCAAUGUUAACUGUAAGACUGUGUCUCGGU ....(((----(........((.((......-))...-)).............(((((((((.((((((.((((((((..........)))))))).)))))).))))))))).)))) ( -33.90) >DroPer_CAF1 10283 113 - 1 CGCCGCCAGGAGAUGU--GAAGGAGCCCAAA-GGAUGCCCGCUCAAUUACCAUGACAUAGUCGUACGGUGAAUGUUGUGUAUUGG--ACCAAUGUUAACUGUAAGACUGUGUCUCGGU .((((...((.(((.(--((.((.((((...-))..))))..)))))).))..(((((((((.((((((.(((((((.((.....--))))))))).)))))).))))))))).)))) ( -38.10) >consensus ____________AU____AAAGGACAAGAAA_GGAUG_CCGCUCAAUUACCCCGACAUAGUCGUACGGUGAAUGUUGUGUAUUGGAGACCAAUGUUAACUGUAAGACUGUGUCUCGGU ................................................(((..(((((((((.((((((.(((((((............))))))).)))))).)))))))))..))) (-26.75 = -26.75 + -0.00)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:27:49 2006