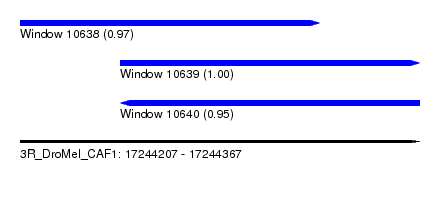
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 17,244,207 – 17,244,367 |
| Length | 160 |
| Max. P | 0.998926 |

| Location | 17,244,207 – 17,244,327 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.44 |
| Mean single sequence MFE | -38.43 |
| Consensus MFE | -36.51 |
| Energy contribution | -36.57 |
| Covariance contribution | 0.06 |
| Combinations/Pair | 1.06 |
| Mean z-score | -1.36 |
| Structure conservation index | 0.95 |
| SVM decision value | 1.65 |
| SVM RNA-class probability | 0.969854 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 17244207 120 + 27905053 CCGUUUAAAAAACUACACACUAAUUGAGUUAGAAGUUCGUUGGGGGCUUUGAACUGGCCGCCGGAUCAAAGGCGCCGCCGCUCUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUG (((((.....(((......(((((...)))))..((((...((.(.((((((.((((...)))).)))))).).))((((((.....))))))........)))))))...))))).... ( -37.70) >DroSec_CAF1 40619 120 + 1 CCGUUUAAAAAACUACACACUAAUUGAGUUAGAAGUUCGUUGGGGGCUUUGAACUGGCCGCCGGAUCAAAGGCGCCGCCGCUCUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUG (((((.....(((......(((((...)))))..((((...((.(.((((((.((((...)))).)))))).).))((((((.....))))))........)))))))...))))).... ( -37.70) >DroSim_CAF1 41345 120 + 1 CCGUUUAAAAAACUACACACUAAUUGAGUUAGAAGUUCGUUGGGGGCUUUGAACUGGCCGCCGGAUCAAAGGCGCCGCCGCUCUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUG (((((.....(((......(((((...)))))..((((...((.(.((((((.((((...)))).)))))).).))((((((.....))))))........)))))))...))))).... ( -37.70) >DroEre_CAF1 41442 118 + 1 CCGUUUAAAAAACUACACACUAAUUGAGUUAGAAGUUCGUCGGGGGCUUUGAACUGGCCGCCGGA--AAAGGCGCCGCCGCUCUAUUGGUGGCGCAACAAAGAGCGUUGACAACGGCAUG (((((.....(((......(((((...)))))..((((.((((.((((.......)))).)))).--....(((((((((......)))))))))......)))))))...))))).... ( -42.10) >DroYak_CAF1 63401 120 + 1 CCGUUUAAAAAACUACACACUAAUUGAGUUAGAAGUUCGUCGGGGGCUUUGAACUGGCCGCCGGAUCAAAGGCGCCGCCGCUCUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUG (((((.....(((......(((((...)))))..((((...((.(.((((((.((((...)))).)))))).).))((((((.....))))))........)))))))...))))).... ( -38.00) >DroAna_CAF1 32505 119 + 1 CCGUUUAAAAAACUACACACUAAUUGAGUUAGAAGUUCGU-GGGGGCUUUGAACUGGCCGACGGAUCAAAGGUGCCGCCACUUUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUG (((((.....(((......(((((...)))))..((((..-.(...((((((.(((.....))).))))))(((((((((......)))))))))..)...)))))))...))))).... ( -37.40) >consensus CCGUUUAAAAAACUACACACUAAUUGAGUUAGAAGUUCGUUGGGGGCUUUGAACUGGCCGCCGGAUCAAAGGCGCCGCCGCUCUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUG (((((.....(((......(((((...)))))..((((...((.(.((((((.(((.....))).)))))).).))((((((.....))))))........)))))))...))))).... (-36.51 = -36.57 + 0.06)



| Location | 17,244,247 – 17,244,367 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 96.89 |
| Mean single sequence MFE | -42.65 |
| Consensus MFE | -41.30 |
| Energy contribution | -41.05 |
| Covariance contribution | -0.25 |
| Combinations/Pair | 1.10 |
| Mean z-score | -1.85 |
| Structure conservation index | 0.97 |
| SVM decision value | 3.29 |
| SVM RNA-class probability | 0.998926 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 17244247 120 + 27905053 UGGGGGCUUUGAACUGGCCGCCGGAUCAAAGGCGCCGCCGCUCUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUGUAAAGCCAACAAUGCCCCAAGCAGCCACUAGAUGAAUGGA .((.(.((((((.((((...)))).)))))).).)).(((.((..((((((((.((((.......)))).....(((((((.......)).))))).......))))))))..)).))). ( -42.20) >DroSec_CAF1 40659 120 + 1 UGGGGGCUUUGAACUGGCCGCCGGAUCAAAGGCGCCGCCGCUCUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUGUAAAGCCAACAAUGCCCCAAGCAGCCUCUAGAUGAAUGGA ..(((((.(((...((((.(((........)))(((((((((.....)))))).((((.......)))).....))).......)))).))).)))))........((((......)))) ( -42.40) >DroSim_CAF1 41385 120 + 1 UGGGGGCUUUGAACUGGCCGCCGGAUCAAAGGCGCCGCCGCUCUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUGUAAAGCCAACAAUGCCCCAAGCAGCCACUAGAUGAAUGGA .((.(.((((((.((((...)))).)))))).).)).(((.((..((((((((.((((.......)))).....(((((((.......)).))))).......))))))))..)).))). ( -42.20) >DroEre_CAF1 41482 118 + 1 CGGGGGCUUUGAACUGGCCGCCGGA--AAAGGCGCCGCCGCUCUAUUGGUGGCGCAACAAAGAGCGUUGACAACGGCAUGUAAAGCCAACAAUGCCCCAAGCAGCCACUAGAUGAAUGGA ..(((((.(((...((((.(((...--...)))(((((((((.....))))))(((((.......)))).)...))).......)))).))).))))).........(((......))). ( -42.20) >DroYak_CAF1 63441 120 + 1 CGGGGGCUUUGAACUGGCCGCCGGAUCAAAGGCGCCGCCGCUCUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUGUAAAGCCAACAAUGCCCCAAGCAGCCACUAGAUGAAUGGA (((.(.((((((.((((...)))).)))))).).)))(((.((..((((((((.((((.......)))).....(((((((.......)).))))).......))))))))..)).))). ( -43.30) >DroAna_CAF1 32545 119 + 1 -GGGGGCUUUGAACUGGCCGACGGAUCAAAGGUGCCGCCACUUUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUGUAAAGCCAACAAUGCCCCAAGCAGCCACCAGAUGAAUGGA -.(((((.(((...((..(((((..((....(((((((((......)))))))))......)).))))).))..(((.......)))..))).))))).........(((......))). ( -43.60) >consensus UGGGGGCUUUGAACUGGCCGCCGGAUCAAAGGCGCCGCCGCUCUAUUGGUGGCACAACAAAGAGCGUUGACAACGGCAUGUAAAGCCAACAAUGCCCCAAGCAGCCACUAGAUGAAUGGA ..(((((.(((...((((.(((........)))(((((((((.....)))))).((((.......)))).....))).......)))).))).))))).........(((......))). (-41.30 = -41.05 + -0.25)

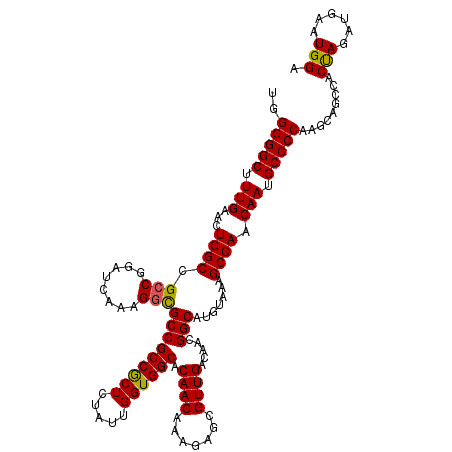
| Location | 17,244,247 – 17,244,367 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 96.89 |
| Mean single sequence MFE | -42.05 |
| Consensus MFE | -39.66 |
| Energy contribution | -40.05 |
| Covariance contribution | 0.39 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.33 |
| Structure conservation index | 0.94 |
| SVM decision value | 1.44 |
| SVM RNA-class probability | 0.954572 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 17244247 120 - 27905053 UCCAUUCAUCUAGUGGCUGCUUGGGGCAUUGUUGGCUUUACAUGCCGUUGUCAACGCUCUUUGUUGUGCCACCAAUAGAGCGGCGGCGCCUUUGAUCCGGCGGCCAGUUCAAAGCCCCCA .(((((.....)))))......(((((.(((((((((......(((((......((((((..((((......)))))))))))))))(((........)))))))))..))).))))).. ( -43.30) >DroSec_CAF1 40659 120 - 1 UCCAUUCAUCUAGAGGCUGCUUGGGGCAUUGUUGGCUUUACAUGCCGUUGUCAACGCUCUUUGUUGUGCCACCAAUAGAGCGGCGGCGCCUUUGAUCCGGCGGCCAGUUCAAAGCCCCCA ......................(((((.(((((((((......(((((......((((((..((((......)))))))))))))))(((........)))))))))..))).))))).. ( -41.00) >DroSim_CAF1 41385 120 - 1 UCCAUUCAUCUAGUGGCUGCUUGGGGCAUUGUUGGCUUUACAUGCCGUUGUCAACGCUCUUUGUUGUGCCACCAAUAGAGCGGCGGCGCCUUUGAUCCGGCGGCCAGUUCAAAGCCCCCA .(((((.....)))))......(((((.(((((((((......(((((......((((((..((((......)))))))))))))))(((........)))))))))..))).))))).. ( -43.30) >DroEre_CAF1 41482 118 - 1 UCCAUUCAUCUAGUGGCUGCUUGGGGCAUUGUUGGCUUUACAUGCCGUUGUCAACGCUCUUUGUUGCGCCACCAAUAGAGCGGCGGCGCCUUU--UCCGGCGGCCAGUUCAAAGCCCCCG .(((((.....)))))......(((((.(((((((((......(((((......((((((..((((......)))))))))))))))(((...--...)))))))))..))).))))).. ( -43.20) >DroYak_CAF1 63441 120 - 1 UCCAUUCAUCUAGUGGCUGCUUGGGGCAUUGUUGGCUUUACAUGCCGUUGUCAACGCUCUUUGUUGUGCCACCAAUAGAGCGGCGGCGCCUUUGAUCCGGCGGCCAGUUCAAAGCCCCCG .(((((.....)))))......(((((.(((((((((......(((((......((((((..((((......)))))))))))))))(((........)))))))))..))).))))).. ( -43.30) >DroAna_CAF1 32545 119 - 1 UCCAUUCAUCUGGUGGCUGCUUGGGGCAUUGUUGGCUUUACAUGCCGUUGUCAACGCUCUUUGUUGUGCCACCAAUAAAGUGGCGGCACCUUUGAUCCGUCGGCCAGUUCAAAGCCCCC- .(((((....(((((((.((..(((((.(((.((((.......))))....))).)))))..))...)))))))....))))).((...((((((.............))))))..)).- ( -38.22) >consensus UCCAUUCAUCUAGUGGCUGCUUGGGGCAUUGUUGGCUUUACAUGCCGUUGUCAACGCUCUUUGUUGUGCCACCAAUAGAGCGGCGGCGCCUUUGAUCCGGCGGCCAGUUCAAAGCCCCCA .(((((.....)))))......(((((.(((((((((......(((((......((((((..((((......)))))))))))))))(((........)))))))))..))).))))).. (-39.66 = -40.05 + 0.39)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:23:53 2006