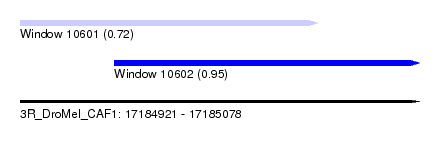
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 17,184,921 – 17,185,078 |
| Length | 157 |
| Max. P | 0.948493 |

| Location | 17,184,921 – 17,185,038 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 85.30 |
| Mean single sequence MFE | -25.32 |
| Consensus MFE | -16.59 |
| Energy contribution | -18.76 |
| Covariance contribution | 2.17 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.15 |
| Structure conservation index | 0.66 |
| SVM decision value | 0.39 |
| SVM RNA-class probability | 0.715836 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 17184921 117 + 27905053 ACACUCUUACUAUUUAUAGCCGCUUAUUUUUAUCAUUUGUAGAAGGCUGUAGCUAGUUUGCCUUUAACUGGCUACAAGUCGAUUGAUAGUCUAAUAUUCUAGUGUUUGGCUAGACAU ................((((((..(((............(((((((((((((((((((.......)))))))))).))))(((.....))).....))))))))..))))))..... ( -28.50) >DroSec_CAF1 6403 116 + 1 ACACUC-UACUAUUUAUAGCCGCUUAUUUAUAUCACUUGUAAAAGGUUGUAGCUAGUUUGCCUUUAACUGGCUACAAGUCGAUUGAUAGUCUAAUAUUCUAGUAUUUGGCUAGACAU .((.((-.(((......((((.....((((((.....)))))).))))((((((((((.......)))))))))).))).)).)).(((((.(((((....))))).)))))..... ( -28.20) >DroSim_CAF1 6808 116 + 1 ACACUC-UACUAUUUAUAGCCGCUUAUUUAUAUCACUUGUAAAAGGUUGUAGCUAGUUUGCCUUUAACUGGCUACAAGUCGAUUGAUAGUCUAAUAUUCUAGUAUUUGGCUAGAGAU ...(((-((.....................(((((.(((.....(.((((((((((((.......)))))))))))).)))).)))))(((.(((((....))))).)))))))).. ( -28.60) >DroEre_CAF1 8279 115 + 1 ACACUC-UACUAUUUAUAGCCGCUCAUAUAUAUCACUUGUAGGAGUUUGUAGCUAUUUUAUCU-UAAUUGGUUACUAGUCGAUUUAUAGUCUACUAUUUUAGUAAUUGGCUACACAU ......-.........(((((((((...((((.....)))).))))..(((((((.((.....-.)).)))))))((((.(((.....))).))))...........)))))..... ( -21.50) >DroYak_CAF1 8109 101 + 1 ACACUC-UACUAUUUAUAGCCGCUUAUUUAUAUCACUUGUGGGAGUUUGUAGCUACU---------------UACAAGUCGAUUUAUAGUCUACUAUUCUAGUAUUUCGCUACAGAU ......-(((((...((((..(((....((.(((((((((((((((.....))).))---------------))))))).))).)).)))...))))..)))))............. ( -19.80) >consensus ACACUC_UACUAUUUAUAGCCGCUUAUUUAUAUCACUUGUAGAAGGUUGUAGCUAGUUUGCCUUUAACUGGCUACAAGUCGAUUGAUAGUCUAAUAUUCUAGUAUUUGGCUAGACAU ........((((((.....(.((((.((((((.....)))))).....((((((((((.......)))))))))))))).)...)))))).......((((((.....))))))... (-16.59 = -18.76 + 2.17)


| Location | 17,184,958 – 17,185,078 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 82.49 |
| Mean single sequence MFE | -33.88 |
| Consensus MFE | -25.92 |
| Energy contribution | -28.28 |
| Covariance contribution | 2.36 |
| Combinations/Pair | 1.23 |
| Mean z-score | -2.24 |
| Structure conservation index | 0.77 |
| SVM decision value | 1.38 |
| SVM RNA-class probability | 0.948493 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 17184958 120 + 27905053 UGUAGAAGGCUGUAGCUAGUUUGCCUUUAACUGGCUACAAGUCGAUUGAUAGUCUAAUAUUCUAGUGUUUGGCUAGACAUCCCUUUUGUGGUCAAGAAGGGUUGAUUGGUAGCAAGGUGG ..(((((((((((((((((((.......)))))))))).))))(((.....))).....))))).(((...(((((.((.(((((((.......))))))).)).))))).)))...... ( -37.30) >DroSec_CAF1 6439 120 + 1 UGUAAAAGGUUGUAGCUAGUUUGCCUUUAACUGGCUACAAGUCGAUUGAUAGUCUAAUAUUCUAGUAUUUGGCUAGACAUUUCUUUUGUGGUCAAGAAGGGUUGAUUGGUAGCGAGGUGG .........((((.(((((((.((((((...((((((((((..((.((.(((((.(((((....))))).)))))..)).))..))))))))))..)))))).))))))).))))..... ( -37.70) >DroSim_CAF1 6844 120 + 1 UGUAAAAGGUUGUAGCUAGUUUGCCUUUAACUGGCUACAAGUCGAUUGAUAGUCUAAUAUUCUAGUAUUUGGCUAGAGAUUCCUUUUGUGGUCAAGAAGGGUUGAUUGGUAGCGAGGUGG .........((((.(((((((.((((((...((((((((((..((((..(((((.(((((....))))).)))))..))))...))))))))))..)))))).))))))).))))..... ( -39.20) >DroEre_CAF1 8315 119 + 1 UGUAGGAGUUUGUAGCUAUUUUAUCU-UAAUUGGUUACUAGUCGAUUUAUAGUCUACUAUUUUAGUAAUUGGCUACACAUCCUUUUUGUGGUCAAGAAGGGUUGAGUGGCAGCGAGGUGG ......(.(((((.(((((((.((((-(..((((....(((((((((.((((....))))......)))))))))((((.......)))).))))..))))).))))))).))))).).. ( -29.80) >DroYak_CAF1 8145 105 + 1 UGUGGGAGUUUGUAGCUACU---------------UACAAGUCGAUUUAUAGUCUACUAUUCUAGUAUUUCGCUACAGAUCCUUUUCGUGGACAAGGAGGGUUCCAUAGCAGCAAGGUGG ((((((((((((((((((((---------------....(((.(((.....))).))).....))))....)))))))))((((((.(....).)))))).)))))))............ ( -25.40) >consensus UGUAGAAGGUUGUAGCUAGUUUGCCUUUAACUGGCUACAAGUCGAUUGAUAGUCUAAUAUUCUAGUAUUUGGCUAGACAUCCCUUUUGUGGUCAAGAAGGGUUGAUUGGUAGCGAGGUGG .........((((.(((((((.((((((...((((((((((..(((...(((((.(((((....))))).)))))...)))...))))))))))..)))))).))))))).))))..... (-25.92 = -28.28 + 2.36)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:23:17 2006