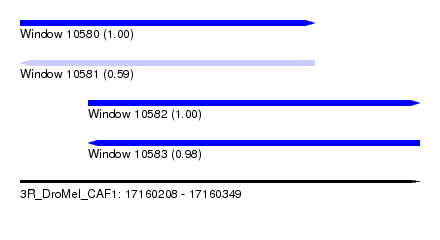
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 17,160,208 – 17,160,349 |
| Length | 141 |
| Max. P | 0.999993 |

| Location | 17,160,208 – 17,160,312 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 86.58 |
| Mean single sequence MFE | -26.05 |
| Consensus MFE | -23.90 |
| Energy contribution | -24.40 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.63 |
| Structure conservation index | 0.92 |
| SVM decision value | 3.03 |
| SVM RNA-class probability | 0.998175 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 17160208 104 + 27905053 GAG-----AUAGAGAUAU--AUAGAAAAAUAUAAAGUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAAGAUAGUCUUGAAUGCCC (((-----((.(((.(((--((......)))))((((....)))).......))).((((.((((((((((............)))))))))).)))))))))........ ( -23.00) >DroVir_CAF1 93180 94 + 1 -------------AUAA----UAAAAAAGUAUAAAGUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCC -------------....----.....((((((.......)))))).....(((((((((((((((((((((............)))))))))))))))....))))))... ( -27.60) >DroGri_CAF1 65266 94 + 1 -------------AGUAAU----AAAAAAUAUAAAGUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCC -------------......----..........((((....)))).....(((((((((((((((((((((............)))))))))))))))....))))))... ( -26.10) >DroWil_CAF1 88150 101 + 1 --------AUUGUAAUAA--UAAAAAAAGUAUAAAGUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCC --------..........--......((((((.......)))))).....(((((((((((((((((((((............)))))))))))))))....))))))... ( -27.60) >DroMoj_CAF1 76444 98 + 1 -------------AUUAAUUGGAAAAAAGUAUAAAGUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCC -------------.......((....((((((.......)))))).....(((((((((((((((((((((............)))))))))))))))....)))))))). ( -28.40) >DroAna_CAF1 62649 109 + 1 UAGAGGAUAGAGAGAUAU--AGAGAAAAAUAUAAAUUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAAGAUAGUCUUGAAUGCCC ..(((.(((((...((((--........))))...))).)).))).....((((((((((.((((((((((............)))))))))).))))....))))))... ( -23.60) >consensus _____________AAUAA___GAAAAAAAUAUAAAGUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCC .................................((((....)))).....(((((((((((((((((((((............)))))))))))))))....))))))... (-23.90 = -24.40 + 0.50)


| Location | 17,160,208 – 17,160,312 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | reverse |
| Mean pairwise identity | 86.58 |
| Mean single sequence MFE | -18.56 |
| Consensus MFE | -17.50 |
| Energy contribution | -17.50 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.20 |
| Structure conservation index | 0.94 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.588197 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 17160208 104 - 27905053 GGGCAUUCAAGACUAUCUUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAACUUUAUAUUUUUCUAU--AUAUCUCUAU-----CUC (((((((((....((((.((((.(((((............))))).)))).)))))))))).................(((((......))--))).)))...-----... ( -18.70) >DroVir_CAF1 93180 94 - 1 GGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAACUUUAUACUUUUUUA----UUAU------------- ...((((((....((((.((((.(((((............))))).)))).)))))))))).............................----....------------- ( -17.50) >DroGri_CAF1 65266 94 - 1 GGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAACUUUAUAUUUUUU----AUUACU------------- ...((((((....((((.((((.(((((............))))).)))).))))))))))...........................----......------------- ( -17.50) >DroWil_CAF1 88150 101 - 1 GGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAACUUUAUACUUUUUUUA--UUAUUACAAU-------- ...((((((....((((.((((.(((((............))))).)))).))))))))))(((((((.......................--...)))))))-------- ( -19.47) >DroMoj_CAF1 76444 98 - 1 GGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAACUUUAUACUUUUUUCCAAUUAAU------------- (((((((((....((((.((((.(((((............))))).)))).))))))))))..((((((.......))))))......))).......------------- ( -19.30) >DroAna_CAF1 62649 109 - 1 GGGCAUUCAAGACUAUCUUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAAAUUUAUAUUUUUCUCU--AUAUCUCUCUAUCCUCUA (((((((((....((((.((((.(((((............))))).)))).))))))))))..((((((.......)))))).........--...........))).... ( -18.90) >consensus GGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAACUUUAUACUUUUUUA___UUAUU_____________ ...((((((....((((.((((.(((((............))))).)))).)))))))))).................................................. (-17.50 = -17.50 + 0.00)



| Location | 17,160,232 – 17,160,349 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.08 |
| Mean single sequence MFE | -32.92 |
| Consensus MFE | -32.83 |
| Energy contribution | -33.17 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.39 |
| Structure conservation index | 1.00 |
| SVM decision value | 5.75 |
| SVM RNA-class probability | 0.999993 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 17160232 117 + 27905053 UAAAGUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAAGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACU---U ..........(((((....((((((((((.((((((((((............)))))))))).))))....))))))((((.......)))).)))))..................---. ( -30.20) >DroPse_CAF1 67863 117 + 1 UAAAAUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACU---U ..........(((((....(((((((((((((((((((((............)))))))))))))))....))))))((((.......)))).)))))..................---. ( -34.10) >DroWil_CAF1 88171 120 + 1 UAAAGUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACUUGUU ..((((.(((((..((...(((((((((((((((((((((............)))))))))))))))....))))))...))..)))))((((.....))))...........))))... ( -34.80) >DroMoj_CAF1 76462 117 + 1 UAAAGUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACU---U ..........(((((....(((((((((((((((((((((............)))))))))))))))....))))))((((.......)))).)))))..................---. ( -34.10) >DroAna_CAF1 62678 117 + 1 UAAAUUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAAGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACU---U ..........(((((....((((((((((.((((((((((............)))))))))).))))....))))))((((.......)))).)))))..................---. ( -30.20) >DroPer_CAF1 79800 117 + 1 UAAAAUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACU---U ..........(((((....(((((((((((((((((((((............)))))))))))))))....))))))((((.......)))).)))))..................---. ( -34.10) >consensus UAAAGUUAAUGCUUACAAUCAUUCAUAUCGUCAUUGGAUCUCAUUAUGUACUGAUCCGAUGAUGAUAGUCUUGAAUGCCCGUUCGUAUUGGGAUGAGCUCCCCCCCUAUGAUCACU___U ..........(((((....(((((((((((((((((((((............)))))))))))))))....))))))((((.......)))).)))))...................... (-32.83 = -33.17 + 0.33)


| Location | 17,160,232 – 17,160,349 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.08 |
| Mean single sequence MFE | -27.50 |
| Consensus MFE | -27.25 |
| Energy contribution | -27.25 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.17 |
| Structure conservation index | 0.99 |
| SVM decision value | 1.77 |
| SVM RNA-class probability | 0.976623 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 17160232 117 - 27905053 A---AGUGAUCAUAGGGGGGGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCUUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAACUUUA .---((((.((.((..((((.......))))..)).))((((.((((((....((((.((((.(((((............))))).)))).)))))))))).))))...))))....... ( -27.50) >DroPse_CAF1 67863 117 - 1 A---AGUGAUCAUAGGGGGGGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAAUUUUA .---((((.((.((..((((.......))))..)).))((((.((((((....((((.((((.(((((............))))).)))).)))))))))).))))...))))....... ( -27.50) >DroWil_CAF1 88171 120 - 1 AACAAGUGAUCAUAGGGGGGGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAACUUUA ....((((.((.((..((((.......))))..)).))((((.((((((....((((.((((.(((((............))))).)))).)))))))))).))))...))))....... ( -27.50) >DroMoj_CAF1 76462 117 - 1 A---AGUGAUCAUAGGGGGGGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAACUUUA .---((((.((.((..((((.......))))..)).))((((.((((((....((((.((((.(((((............))))).)))).)))))))))).))))...))))....... ( -27.50) >DroAna_CAF1 62678 117 - 1 A---AGUGAUCAUAGGGGGGGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCUUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAAAUUUA .---((((.((.((..((((.......))))..)).))((((.((((((....((((.((((.(((((............))))).)))).)))))))))).))))...))))....... ( -27.50) >DroPer_CAF1 79800 117 - 1 A---AGUGAUCAUAGGGGGGGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAAUUUUA .---((((.((.((..((((.......))))..)).))((((.((((((....((((.((((.(((((............))))).)))).)))))))))).))))...))))....... ( -27.50) >consensus A___AGUGAUCAUAGGGGGGGAGCUCAUCCCAAUACGAACGGGCAUUCAAGACUAUCAUCAUCGGAUCAGUACAUAAUGAGAUCCAAUGACGAUAUGAAUGAUUGUAAGCAUUAACUUUA ....((((.((.((..((((.......))))..)).))((((.((((((....((((.((((.(((((............))))).)))).)))))))))).))))...))))....... (-27.25 = -27.25 + 0.00)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:23:00 2006