
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 17,051,968 – 17,052,083 |
| Length | 115 |
| Max. P | 0.845074 |

| Location | 17,051,968 – 17,052,083 |
|---|---|
| Length | 115 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.84 |
| Mean single sequence MFE | -46.82 |
| Consensus MFE | -20.39 |
| Energy contribution | -19.95 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.50 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.44 |
| SVM decision value | 0.77 |
| SVM RNA-class probability | 0.845074 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
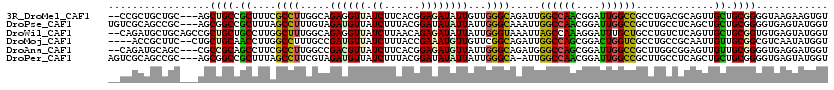
>3R_DroMel_CAF1 17051968 115 - 27905053 --CCGCUGCUGC---AGCUGCCGCUUUCGCCUUGGCAGAGGUUAUCUUCACGGAGAUAUUGUUGGGCAGAUUGGCCAACGGAUUGGCCGCCUGACGCAGUUGCUGCGGGGUAAGAAGUGU --((.((((.((---((((((.((((..((....))..))))((((((....))))))..((..(((.....((((((....)))))))))..)))))))))).)))))).......... ( -50.60) >DroPse_CAF1 2333 117 - 1 UGUCGCAGCCGC---AGCGGCCGCUUUAGCCUUUGUAGAUGUUAUCUUUACGGAUAUAUUAUUGGGCAAAUUGGCCAACGGAUUGGCCGCUUGCCUCAGCUGCUGCGGGGUGAGUAUGGU ..((((..((((---((((((.((....)).((((((((.......))))))))........(((((((..(((((((....))))))).))))).)))))))))))).))))....... ( -46.40) >DroWil_CAF1 9969 118 - 1 --CAGAUGCUGCAGCCGCUGCUGCCUUGGCUUUGGCAGAGGUUAUCUUAACAGAGAUAUUAUUGGGUAAAUUAGCCAAAGGAUUUGCUGCCUGUCUCAGUUGCUGCGGUGUGAGUAUGGU --...((((((((((.(((((((((........)))))((((((((((....))))))......((((((((........))))))))))))....)))).))))))))))......... ( -41.30) >DroMoj_CAF1 2643 114 - 1 ----ACCGCUUC--CUGCUGCAACCUUGGCCUUUGCCGAUGUUAUCUUUACCGAAAUGUUGUUCGGCAGAUUGGCCAGCGGACUGGUCGCCUGCCGCAAUUGUUGCGGCGUCAAUAUGGU ----((((....--.(((((((((..(((((((((((((....((.(((....))).))...))))))))..)))))((((...((...))..))))....)))))))))......)))) ( -41.70) >DroAna_CAF1 2153 115 - 1 --CAGAUGCAGC---CGCCGCAGCCUUCGCCUUGGCCGACGUUAUCUUCACGGAGAUGUUAUUGGGCAGAUGGGCCAGCGGAUUGGCCGCUUGGCGGAGUUGUUGCGGGGUGAGGAUGGU --((..(...((---(.(((((((((((((((((.((((...((((((....))))))...)))).)))...((((((....))))))....)))))))..))))))))))..)..)).. ( -50.40) >DroPer_CAF1 2324 116 - 1 AGUCGCAGCCGC---AGCGGCCGCUUUAGCCUUCGUAGAUGUUAUCUUUACGGAUAUAUUAUUGGGCA-AUUGGCCAACGGAUUGGCCGCUUGCCUCAGCUGCUGCGGGGUGAGUAUGGU (.((((..((((---((((((.((....)).((((((((.......))))))))........((((((-(.(((((((....))))))).))))).)))))))))))).)))).)..... ( -50.50) >consensus __CCGCUGCUGC___AGCUGCCGCCUUAGCCUUGGCAGAUGUUAUCUUUACGGAGAUAUUAUUGGGCAGAUUGGCCAACGGAUUGGCCGCCUGCCGCAGUUGCUGCGGGGUGAGUAUGGU .................((((.((....((((.....((((((.((......))))))))...)))).....((((((....)))))).............)).))))............ (-20.39 = -19.95 + -0.44)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:22:26 2006