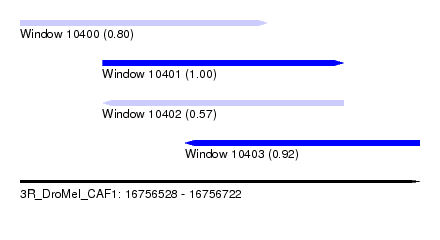
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 16,756,528 – 16,756,722 |
| Length | 194 |
| Max. P | 0.999444 |

| Location | 16,756,528 – 16,756,648 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.72 |
| Mean single sequence MFE | -45.43 |
| Consensus MFE | -43.29 |
| Energy contribution | -43.82 |
| Covariance contribution | 0.53 |
| Combinations/Pair | 1.02 |
| Mean z-score | -1.00 |
| Structure conservation index | 0.95 |
| SVM decision value | 0.61 |
| SVM RNA-class probability | 0.800096 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 16756528 120 + 27905053 CAGGCGUUGUUGAUGCUGCUGCUGCUGGCGGAGGAUGAUUUGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGG ...(((((((((........(((((..(((((......))))))))))((((((((((((((((((.....)))))....)))).)))))))))))))))))).(((((....).)))). ( -46.20) >DroPse_CAF1 37672 120 + 1 CAGGCGUUGUUGAUGCUGCUGCUGCUGGAGGAGGAUGAUUUGCGCAGUGACGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGACGG ....((((((..((.((.((.(....).)).)).))(((((((((.(.((((((((((((((((((.....)))))....)))).))))))))))(....))))))...)))))))))). ( -44.10) >DroSim_CAF1 52393 120 + 1 CAGGCGUUGUUGAUGCUGCUGCUGCUGGCGGAGGAUGAUUUGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGG ...(((((((((........(((((..(((((......))))))))))((((((((((((((((((.....)))))....)))).)))))))))))))))))).(((((....).)))). ( -46.20) >DroEre_CAF1 51204 120 + 1 CAGGCGUUGUUGAUGCUGCUGCUGCUGGCGGAGGAUGAUUUGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGG ...(((((((((........(((((..(((((......))))))))))((((((((((((((((((.....)))))....)))).)))))))))))))))))).(((((....).)))). ( -46.20) >DroYak_CAF1 34351 120 + 1 CAGGCGUUGUUGAUGCUGCUGCUGCUGGCGGAGGAUGAUUUGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGG ...(((((((((........(((((..(((((......))))))))))((((((((((((((((((.....)))))....)))).)))))))))))))))))).(((((....).)))). ( -46.20) >DroAna_CAF1 32169 120 + 1 CAGGCGUUGUUGAUGCUGCUGCUGCUGGAGGAGGAUGAUUUGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGG ...(((((((((.....(((((.((................)))))))((((((((((((((((((.....)))))....)))).)))))))))))))))))).(((((....).)))). ( -43.69) >consensus CAGGCGUUGUUGAUGCUGCUGCUGCUGGCGGAGGAUGAUUUGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGG ...(((((((((........(((((..(((((......))))))))))((((((((((((((((((.....)))))....)))).)))))))))))))))))).(((((....).)))). (-43.29 = -43.82 + 0.53)

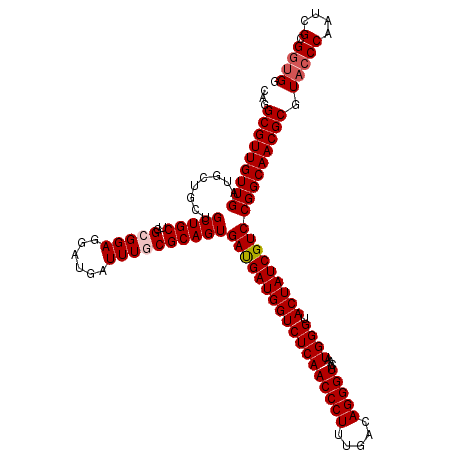

| Location | 16,756,568 – 16,756,685 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 94.64 |
| Mean single sequence MFE | -37.88 |
| Consensus MFE | -36.06 |
| Energy contribution | -35.92 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.95 |
| SVM decision value | 3.61 |
| SVM RNA-class probability | 0.999444 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 16756568 117 + 27905053 UGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGGUCACUGUUGUACCAAAUACCUGAAAAAUGUCAAUAUA ((((((((((((((((((((((((((.....)))))....)))).))))))))).(((..((((........))))...))))))).)))).......................... ( -35.80) >DroPse_CAF1 37712 115 + 1 UGCGCAGUGACGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGACGGUCACUGUUGUACCAGAUGCCUGCAAAAAAAGAGGA-- ...((((.((((((((((((((((((.....)))))....)))).))))))))))((((.((((........))))..(((.((....)))))...)))))))............-- ( -41.20) >DroSec_CAF1 59003 117 + 1 UGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGGUCACUGUUGUACCAAAUACCUGGAAAAUAUCAAAAUA ((((((((((((((((((((((((((.....)))))....)))).))))))))).(((..((((........))))...))))))).))))(((......))).............. ( -37.90) >DroSim_CAF1 52433 117 + 1 UGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGGUCACUGUUGUACCAAAUACCUGGAAAAUAUCAAAAUA ((((((((((((((((((((((((((.....)))))....)))).))))))))).(((..((((........))))...))))))).))))(((......))).............. ( -37.90) >DroEre_CAF1 51244 117 + 1 UGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGGUCACUGUUGUACCAAAUACCUGGAAUAUAUUAAUAUA ((((((((((((((((((((((((((.....)))))....)))).))))))))).(((..((((........))))...))))))).))))(((......))).............. ( -37.90) >DroYak_CAF1 34391 116 + 1 UGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGGUCACUGUUGUACCAAAUACCUGCAAAAUAU-AAUAUA (((((.(.((((((((((((((((((.....)))))....)))).))))))))))(....)))))).......((((((((.((....)))))).....)))).......-...... ( -36.60) >consensus UGCGCAGUGAUGAUGGUCUCAACCCUUUGACAGGGUACAAUGGGUACUAUCGUCCGGCAACGCGUACCCAAUCGCGGUGGUCACUGUUGUACCAAAUACCUGGAAAAUAUCAAUAUA ((((((((((((((((((((((((((.....)))))....)))).))))))))).(((..((((........))))...))))))).)))).......................... (-36.06 = -35.92 + -0.14)

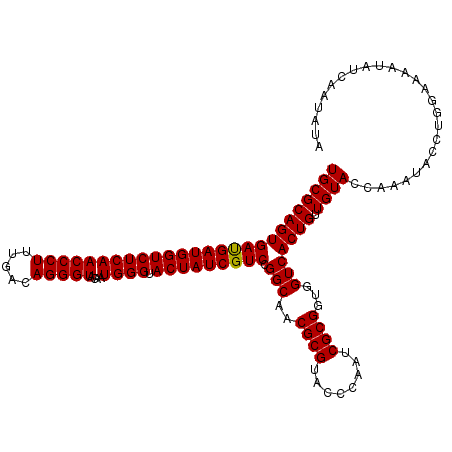

| Location | 16,756,568 – 16,756,685 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 94.64 |
| Mean single sequence MFE | -34.13 |
| Consensus MFE | -31.72 |
| Energy contribution | -31.72 |
| Covariance contribution | -0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -0.94 |
| Structure conservation index | 0.93 |
| SVM decision value | 0.08 |
| SVM RNA-class probability | 0.573615 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 16756568 117 - 27905053 UAUAUUGACAUUUUUCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCAUUGUACCCUGUCAAAGGGUUGAGACCAUCAUCACUGCGCA ....((((......))))((((...))))..((((((.....(((((.((((((.((((....))))...)))))))))))(((((.....)))))...........)))))).... ( -32.40) >DroPse_CAF1 37712 115 - 1 --UCCUCUUUUUUUGCAGGCAUCUGGUACAACAGUGACCGUCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCAUUGUACCCUGUCAAAGGGUUGAGACCAUCGUCACUGCGCA --...........(((..((.....))....(((((((.((((((((.((((((.((((....))))...)))))))))))(((((.....)))))...)))....))))))).))) ( -38.10) >DroSec_CAF1 59003 117 - 1 UAUUUUGAUAUUUUCCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCAUUGUACCCUGUCAAAGGGUUGAGACCAUCAUCACUGCGCA ..............((((....)))).....((((((.....(((((.((((((.((((....))))...)))))))))))(((((.....)))))...........)))))).... ( -32.60) >DroSim_CAF1 52433 117 - 1 UAUUUUGAUAUUUUCCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCAUUGUACCCUGUCAAAGGGUUGAGACCAUCAUCACUGCGCA ..............((((....)))).....((((((.....(((((.((((((.((((....))))...)))))))))))(((((.....)))))...........)))))).... ( -32.60) >DroEre_CAF1 51244 117 - 1 UAUAUUAAUAUAUUCCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCAUUGUACCCUGUCAAAGGGUUGAGACCAUCAUCACUGCGCA ..((((((.((((.....))))))))))...((((((.....(((((.((((((.((((....))))...)))))))))))(((((.....)))))...........)))))).... ( -32.80) >DroYak_CAF1 34391 116 - 1 UAUAUU-AUAUUUUGCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCAUUGUACCCUGUCAAAGGGUUGAGACCAUCAUCACUGCGCA ......-.......((((.....((((.((((..((((....(((((.((((((.((((....))))...))))))))))).....))))....))))..))))......))))... ( -36.30) >consensus UAUAUUGAUAUUUUCCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCAUUGUACCCUGUCAAAGGGUUGAGACCAUCAUCACUGCGCA ...............(((.....((((.((((..((((....(((((.((((((.((((....))))...))))))))))).....))))....))))..))))......))).... (-31.72 = -31.72 + -0.00)

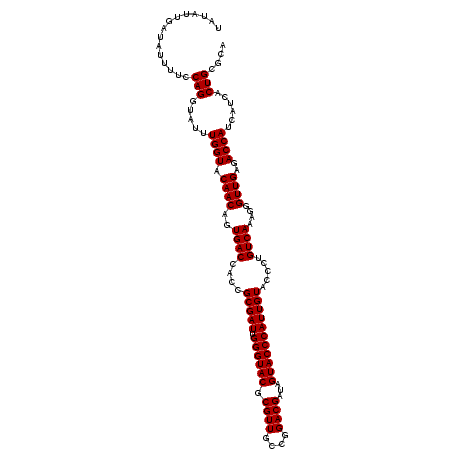

| Location | 16,756,608 – 16,756,722 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 82.17 |
| Mean single sequence MFE | -28.58 |
| Consensus MFE | -20.56 |
| Energy contribution | -21.48 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.72 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.920613 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 16756608 114 - 27905053 GCAACCAAAUUUCAACAUACAGAAUAA---UUGUAACAAAUAUAUUGACAUUUUUCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCA ((........(((........)))...---(((((.(((((((.((((......))))))))))).)))))...........))...(((((((.((((....))))...))))))) ( -26.20) >DroSec_CAF1 59043 114 - 1 ACAAUCAAAACUCAACAUACAGAAUAU---UUGUAGCAAAUAUUUUGAUAUUUUCCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCA ..((((.....................---(((((.(((((((((.((.....)).))))))))).)))))..(((.....)))))))((((((.((((....))))...)))))). ( -31.40) >DroSim_CAF1 52473 112 - 1 --AAUCAAAACUCAACAUGCAGAAUAA---UUGUAGCAAAUAUUUUGAUAUUUUCCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCA --((((.....................---(((((.(((((((((.((.....)).))))))))).)))))..(((.....)))))))((((((.((((....))))...)))))). ( -31.20) >DroEre_CAF1 51284 94 - 1 -----------------------ACCAACUAUAUACCAAAUAUAUUAAUAUAUUCCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCA -----------------------....((((.(((((.((((((....))))))...))))).))))......(((.....)))...(((((((.((((....))))...))))))) ( -25.10) >DroYak_CAF1 34431 113 - 1 GACACCAAAAGUCACUAUACGGAACCA---UUAAACCAAAUAUAUU-AUAUUUUGCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCA ..........((((((....(....).---....(((((((((..(-(.....))...))))))))).....)))))).........(((((((.((((....))))...))))))) ( -29.00) >consensus __AACCAAAACUCAACAUACAGAAUAA___UUGUACCAAAUAUAUUGAUAUUUUCCAGGUAUUUGGUACAACAGUGACCACCGCGAUUGGGUACGCGUUGCCGGACGAUAGUACCCA ...............................((((.(((((((...((.....))...))))))).))))...(((.....)))...(((((((.((((....))))...))))))) (-20.56 = -21.48 + 0.92)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:20:07 2006