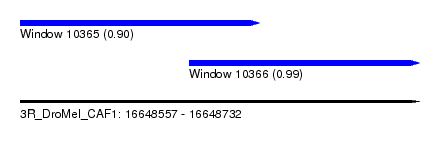
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 16,648,557 – 16,648,732 |
| Length | 175 |
| Max. P | 0.990636 |

| Location | 16,648,557 – 16,648,662 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 111 |
| Reading direction | forward |
| Mean pairwise identity | 77.27 |
| Mean single sequence MFE | -32.02 |
| Consensus MFE | -22.13 |
| Energy contribution | -21.63 |
| Covariance contribution | -0.50 |
| Combinations/Pair | 1.28 |
| Mean z-score | -1.63 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.03 |
| SVM RNA-class probability | 0.902803 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
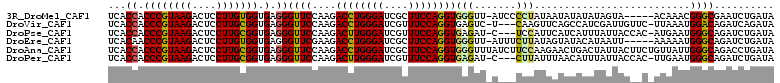
>3R_DroMel_CAF1 16648557 105 + 27905053 UCACCACCCGUAAGACUCCUUGUGGUGAGGGUUCCAAGACCUGGGAUCGCUUCCAGGUGGGUU-AUCCCCUAUAAUAUAUAUAGUA-----ACAAACGGGCGAAUCUGAUA (((((((....(((....))))))))))(((((((..(((((((((....))))))))(((..-..)))(((((.....)))))..-----.....)..).)))))).... ( -30.80) >DroVir_CAF1 1393 106 + 1 UCACCACCCGUAAGACUCCUUGCGGUGAGGGUUCCAAGACCUGGGAUCGUUUCCAGGUGAGUC-U---CAAGUUCAGCCAUCGAUUGUUC-UUAAAUGGACAGAUCAGAUA .......(((((((....)))))))(((((.((.....((((((((....)))))))))).))-)---)).........(((((((((((-......))))).))).))). ( -32.50) >DroPse_CAF1 1047 106 + 1 UCACCACCCGUAAGACUCCUUGCGGUGAGGGUUCCAAGACUUGGGAUCGUUUCCAGGUGAGAU-C---UCCAUUCAUCAUUUAUUACCAC-AUGAAUGGGCAGAUCUGAUA (((((..(((((((....))))))).((.(((((((.....))))))).))....)))))(((-(---(((((((((.............-))))))))..)))))..... ( -36.92) >DroEre_CAF1 1114 105 + 1 UCACAACCCGUAAGACUCCUUGUGGUGAGGGUUCGAAGACCUGGGAUCGCUUCCAGGUGGGUU-AUUUCUUAUAGUAUACAUAAUU-----AAAAAUGGGCAGAUCUGAUA (((.(((((....((..((((.....))))..))....((((((((....)))))))))))))-..........((...(((....-----....))).)).....))).. ( -25.80) >DroAna_CAF1 7769 111 + 1 UCACCACCCGUAAGACUCCUUGCGGUGAGGGUUCCAAGACCUGGGAUCGCUUCCAGGUGGGUUUAUCUUCCAAGAACUGACUAUUACUUCUGUUAUUGGGCAGACCUGAUA ...((.((((((((....))))))).).))((((....((((((((....))))))))(((.......)))..))))....(((((..((((((....))))))..))))) ( -34.90) >DroPer_CAF1 1064 106 + 1 UCACCACCCGUAAGACUCCUUGCGGUGAGGGUUCCAAGACUUGGGAUCGUUUCCAGGUGAGAU-C---CUUAUUUAACAUUUAUUACCAC-UUGAAUGGGCAGAUCUGAUA (((((..(((((((....))))))).((.(((((((.....))))))).))....)))))(((-(---(((((((((.............-)))))))))..))))..... ( -31.22) >consensus UCACCACCCGUAAGACUCCUUGCGGUGAGGGUUCCAAGACCUGGGAUCGCUUCCAGGUGAGUU_A___CCUAUUAAACAUUUAUUAC__C_AUAAAUGGGCAGAUCUGAUA ...((.((((((((....))))))).).))((((....((((((((....))))))))(((.......)))..........................)))).......... (-22.13 = -21.63 + -0.50)


| Location | 16,648,631 – 16,648,732 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 74.28 |
| Mean single sequence MFE | -29.18 |
| Consensus MFE | -22.53 |
| Energy contribution | -21.90 |
| Covariance contribution | -0.63 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.40 |
| Structure conservation index | 0.77 |
| SVM decision value | 2.22 |
| SVM RNA-class probability | 0.990636 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
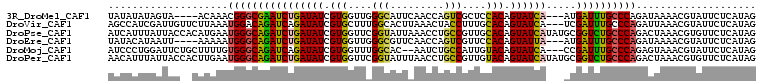
>3R_DroMel_CAF1 16648631 101 + 27905053 UAUAUAUAGUA----ACAAACGGGCGAAUCUGAUAUCGUGGUUGGGCAUUCAACCAGUCGCUCCACAGUAUCA---AUGAUUUGCCCAGAUAAAACGUAUUCUCAUAG ...........----......(((((((((((((((.((((((((........)))).....)))).))))))---..)))))))))..................... ( -28.90) >DroVir_CAF1 1464 105 + 1 AGCCAUCGAUUGUUCUUAAAUGGACAGAUCAGAUAUCGUGCUUUGGCACUUAAACUACCUUUGCACAGUAUCA---UCGAUUUGCCCAGAUUAAACGUAUUCUCAUAG .((.(((((((((((......))))))....(((((.((((...((...........))...)))).))))).---)))))..))....................... ( -22.30) >DroPse_CAF1 1118 108 + 1 AUCAUUUAUUACCACAUGAAUGGGCAGAUCUGAUAUCGUGGUUCGGUAUUAAACCUGCCGUUGCACAGUAUCAUAUGCGGUCUGCCCAGACUAAACGUGUUCUCAUAG .............(((((..((((((((((((((((.(((...(((((.......)))))...))).)))))).....)))))))))).......)))))........ ( -33.40) >DroEre_CAF1 1188 101 + 1 UAUACAUAAUU----AAAAAUGGGCAGAUCUGAUAUCGUGGUUGGGCGUUCAACCAGUCGUUCCACAGUAUUA---AUGAUUUGCCCAGAUAAAACGUAUUCUCAUAG ...........----.....((((((((((((((((.((((...(((.........)))...)))).))))))---..)))))))))).................... ( -28.00) >DroMoj_CAF1 1468 103 + 1 AUCCCUGGAUUCUGCUUUUGUGGGCAGAUCAGAUAUCGUGGUUUGGCAC--AAUCUGCCAUUGUACAGUAUCA---CCGAUUUGCCCAGAGUAAACGUAUUCUCAUAG (((....)))..(((((...((((((((((.(((((.(((...(((((.--....)))))...))).))))).---..)))))))))))))))............... ( -31.50) >DroPer_CAF1 1135 108 + 1 AACAUUUAUUACCACUUGAAUGGGCAGAUCUGAUAUCGUGGUUCGGUAUUUAACCUGCCGUUGUACAGUAUCAUAUGCGGUCUGCCCAGACUAAACGUGUUCUCAUAG ............(((((...((((((((((((((((.(((...(((((.......)))))...))).)))))).....)))))))))).....)).)))......... ( -31.00) >consensus AACAUUUAAUAC__CAUAAAUGGGCAGAUCUGAUAUCGUGGUUCGGCAUUCAACCUGCCGUUGCACAGUAUCA___ACGAUUUGCCCAGAUUAAACGUAUUCUCAUAG ....................((((((((((((((((.(((....(((.........)))....))).)))))).....)))))))))).................... (-22.53 = -21.90 + -0.63)
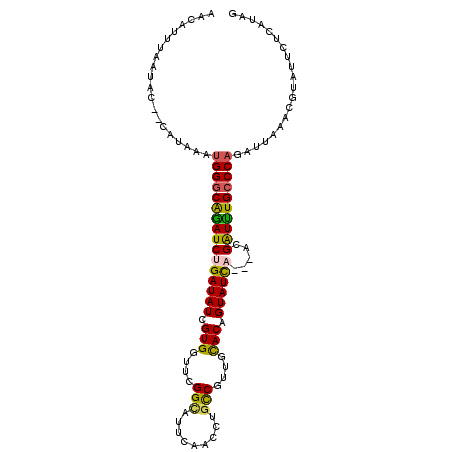
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:19:30 2006