| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 2,334,366 – 2,334,465 |
| Length | 99 |
| Max. P | 0.986500 |

| Location | 2,334,366 – 2,334,465 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.34 |
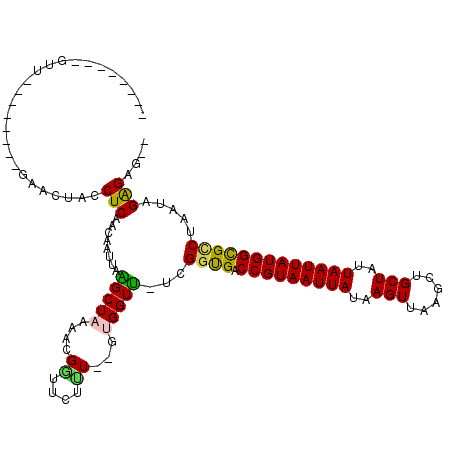
| Mean single sequence MFE | -31.57 |
| Consensus MFE | -18.76 |
| Energy contribution | -18.07 |
| Covariance contribution | -0.69 |
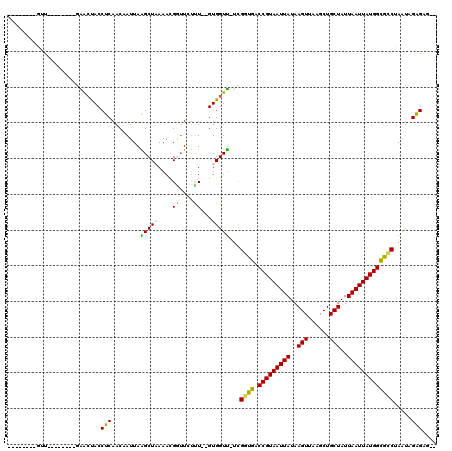
| Combinations/Pair | 1.31 |
| Mean z-score | -2.53 |
| Structure conservation index | 0.59 |
| SVM decision value | 2.04 |
| SVM RNA-class probability | 0.986500 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
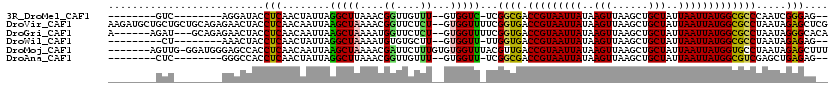
>3R_DroMel_CAF1 2334366 99 - 27905053 --------GUC--------AGGAUACCUCAACUAUUAGGCUUAAACGGUUGUUU--GUGGUC-UCGGCGACCGUAAUUAUAAGUUAAGCUGCUAUUAAUUAUGGCGCCCAAUCGGGAG-- --------...--------.......(((.......((((((((((....))))--).))))-).((((.(((((((((..(((......)))..))))))))))))).....)))..-- ( -26.30) >DroVir_CAF1 106934 118 - 1 AAGAUGCUGCUGCUGCAGAGAACUACCUCAACAAUUAAGCUAAAACGGUUCUCU--GUGGUUUUCGGUGACCGUAAUUAUAAGUUAAGCUGCUAUUAAUUAUGGCGCCUAAUAGAGCUCG ..((.(((.((((..(((((((((......................))))))))--)..))....((((.(((((((((..(((......)))..)))))))))))))....))))))). ( -37.75) >DroGri_CAF1 112745 109 - 1 A------AGAU---GCAGAGAACUACCUCAACAAUUAAGCUAAAAUGGUUCUCU--GUGGUUUUCGGUGACCGUAAUUAUAAGUUAAGCUGCUAUUAAUUAUGGCGCCUAAUAGGGCACA .------....---(((((((((((....................)))))))))--)).(((((.((((.(((((((((..(((......)))..)))))))))))))....)))))... ( -30.55) >DroWil_CAF1 106354 98 - 1 ---------CU--------AAACUACCUCAACUAUUAGGCUAAAAUGUGUGCUU--GUGGUU-UUGGUGACCGUAAUUAUAAGUUAAGCUGCUAUUAAUUAUGGCGCCUAAUAGAGAG-- ---------..--------.......(((..(((((((((......((..((((--((((((-..(....)...))))))))))...)).(((((.....)))))))))))))).)))-- ( -30.20) >DroMoj_CAF1 120104 112 - 1 -------AGUUG-GGAUGGGAGCCACCUCAACAAUUAAGCUAAAACGAUUCUUUGUGUGGUUUACGUUGACCGUAAUUAUAAGUUAAGCUGCUAUUAAUUAUGGUGCCUAAUAGAGCUUU -------.((((-((.((((......)))).(((((((((((..((((....)))).))))))).))))((((((((((..(((......)))..)))))))))).))))))........ ( -28.00) >DroAna_CAF1 41742 99 - 1 --------CUC--------GGGCCACCUCAACUAUUAGGCUUAAACGGUUGUUU--GUGGUU-UCGGCGACCGUAAUUAUAAGUUAAGCUGCUAUUAAUUAUGGCGUCGAGCUGAGAG-- --------(((--------(((((((..((((..((.......))..))))...--)))))(-((((((.(((((((((..(((......)))..)))))))))))))))))))))..-- ( -36.60) >consensus ________GUU________GAACUACCUCAACAAUUAAGCUAAAACGGUUCUUU__GUGGUU_UCGGUGACCGUAAUUAUAAGUUAAGCUGCUAUUAAUUAUGGCGCCUAAUAGAGAG__ ..........................(((........(((((....((....))...)))))...((((.(((((((((..(((......)))..))))))))))))).....))).... (-18.76 = -18.07 + -0.69)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:47:59 2006