
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 16,578,793 – 16,578,910 |
| Length | 117 |
| Max. P | 0.999964 |

| Location | 16,578,793 – 16,578,910 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.05 |
| Mean single sequence MFE | -44.03 |
| Consensus MFE | -37.32 |
| Energy contribution | -38.36 |
| Covariance contribution | 1.04 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.79 |
| Structure conservation index | 0.85 |
| SVM decision value | 4.05 |
| SVM RNA-class probability | 0.999775 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
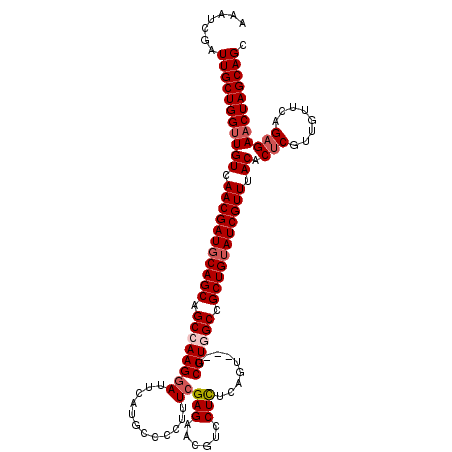
>3R_DroMel_CAF1 16578793 117 + 27905053 AAAUCGAUUGCUGGUUGUCAACGAUGCAGCAGCCAAGGAUUUAUGCCCCUUUCGAGAAAGUCCUUUCAGU---GCUUGGCCGCUGUAUCGUUUACACUCGUUGGUCAGAGAACUAGCAGC .......(((((((((((.(((((((((((.((((((.(((.......((((....)))).......)))---.)))))).))))))))))).)).(((........)))))))))))). ( -46.04) >DroSec_CAF1 135008 117 + 1 AAAUCGAUUGCUGGUUGUCAACGAUGCAGCAGCCAAGGAUUCAUGCCCCUUUCGAGAACGUCCUCUCAGU---GCUUGGCCGCUGUAUCGUUUACACUCGUUGUUCAGAGAACUAGCAGC .......(((((((((((.(((((((((((.((((((.(((............((((......)))))))---.)))))).))))))))))).)).(((........)))))))))))). ( -46.50) >DroSim_CAF1 140011 117 + 1 AAAUCGAUUGCUGGUUGUCAACGAUGCAGCAGCCAAGGAUUCAUGCCCCUUUCGAGAACGUCCUCUCAGU---GCUUGGCCGCUGUAUCGUUUACACUCGUUGUUCAGAGAACUAGCAGC .......(((((((((((.(((((((((((.((((((.(((............((((......)))))))---.)))))).))))))))))).)).(((........)))))))))))). ( -46.50) >DroEre_CAF1 139110 117 + 1 AAAUCGAUUGCUGGUUGUCAACGAUCCAGCGGCCAAGGACUCAUGCCCCUUUCGAGAACAUCCUCCCAAU---GCUUAGCCGCUGUAUCGUUUACACUCGUUGAUCAGAGAACUAGCAGC .......(((((((((((.((((((.(((((((.((((.........))))..(((......))).....---.....))))))).)))))).)).(((........)))))))))))). ( -39.60) >DroYak_CAF1 135126 120 + 1 AAAUCGAUUGCUGGUUGUCAACGAUGCAGCGGCCAAGCACUCACGCCCCUUUCGAGAACGUUCUCACAAAACAGCUUAGCCGCUGUAUCGUUUACACACGUUGUUCAGAGAUCUAGCAGC .......(((((((.(.((((((((((((((((.((((.(((...........)))...(((.......))).)))).)))))))))))))).(((.....)))...)).).))))))). ( -41.50) >consensus AAAUCGAUUGCUGGUUGUCAACGAUGCAGCAGCCAAGGAUUCAUGCCCCUUUCGAGAACGUCCUCUCAGU___GCUUGGCCGCUGUAUCGUUUACACUCGUUGUUCAGAGAACUAGCAGC .......(((((((((((.(((((((((((.((((((((............))(((......))).........)))))).))))))))))).)).(((........)))))))))))). (-37.32 = -38.36 + 1.04)

| Location | 16,578,793 – 16,578,910 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.05 |
| Mean single sequence MFE | -44.52 |
| Consensus MFE | -40.42 |
| Energy contribution | -39.74 |
| Covariance contribution | -0.68 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.27 |
| Structure conservation index | 0.91 |
| SVM decision value | 4.95 |
| SVM RNA-class probability | 0.999964 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
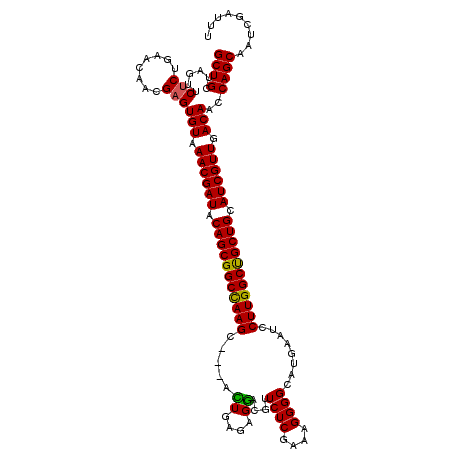
>3R_DroMel_CAF1 16578793 117 - 27905053 GCUGCUAGUUCUCUGACCAACGAGUGUAAACGAUACAGCGGCCAAGC---ACUGAAAGGACUUUCUCGAAAGGGGCAUAAAUCCUUGGCUGCUGCAUCGUUGACAACCAGCAAUCGAUUU ((((......(((........)))(((.((((((.(((((((((((.---.((....))....((((....))))........))))))))))).)))))).)))..))))......... ( -42.20) >DroSec_CAF1 135008 117 - 1 GCUGCUAGUUCUCUGAACAACGAGUGUAAACGAUACAGCGGCCAAGC---ACUGAGAGGACGUUCUCGAAAGGGGCAUGAAUCCUUGGCUGCUGCAUCGUUGACAACCAGCAAUCGAUUU ((((...((((...))))......(((.((((((.(((((((((((.---.((....)).(((((((....)))).)))....))))))))))).)))))).)))..))))......... ( -45.10) >DroSim_CAF1 140011 117 - 1 GCUGCUAGUUCUCUGAACAACGAGUGUAAACGAUACAGCGGCCAAGC---ACUGAGAGGACGUUCUCGAAAGGGGCAUGAAUCCUUGGCUGCUGCAUCGUUGACAACCAGCAAUCGAUUU ((((...((((...))))......(((.((((((.(((((((((((.---.((....)).(((((((....)))).)))....))))))))))).)))))).)))..))))......... ( -45.10) >DroEre_CAF1 139110 117 - 1 GCUGCUAGUUCUCUGAUCAACGAGUGUAAACGAUACAGCGGCUAAGC---AUUGGGAGGAUGUUCUCGAAAGGGGCAUGAGUCCUUGGCCGCUGGAUCGUUGACAACCAGCAAUCGAUUU ((((......(((........)))(((.((((((.((((((((....---.(((((((....))))))).((((((....)))))))))))))).)))))).)))..))))......... ( -43.00) >DroYak_CAF1 135126 120 - 1 GCUGCUAGAUCUCUGAACAACGUGUGUAAACGAUACAGCGGCUAAGCUGUUUUGUGAGAACGUUCUCGAAAGGGGCGUGAGUGCUUGGCCGCUGCAUCGUUGACAACCAGCAAUCGAUUU ((((.(((....))).........(((.((((((.((((((((((((.((((.....))))...(((....)))((....)))))))))))))).)))))).)))..))))......... ( -47.20) >consensus GCUGCUAGUUCUCUGAACAACGAGUGUAAACGAUACAGCGGCCAAGC___ACUGAGAGGACGUUCUCGAAAGGGGCAUGAAUCCUUGGCUGCUGCAUCGUUGACAACCAGCAAUCGAUUU ((((......(((........)))(((.((((((.(((((((((((.....((....))....((((....))))........))))))))))).)))))).)))..))))......... (-40.42 = -39.74 + -0.68)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:18:59 2006