
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 16,515,831 – 16,515,939 |
| Length | 108 |
| Max. P | 0.593949 |

| Location | 16,515,831 – 16,515,939 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.17 |
| Mean single sequence MFE | -44.00 |
| Consensus MFE | -31.48 |
| Energy contribution | -31.37 |
| Covariance contribution | -0.11 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.12 |
| SVM RNA-class probability | 0.593949 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 16515831 108 - 27905053 CUGCACCA------UUCUGAGAUGAGCAACGACCC------CAGGUGACGAUCGUUUGGGCCCAGCUCGAGCUCGCAAAGCAGCUUGGGCGGGUUACGCAAGACGAGCCUCAAGGUGCCC ..(((((.------...((((....((........------..(....)..((((((.(((((.(((((((((.((...)))))))))))))))).....)))))))))))).))))).. ( -41.00) >DroVir_CAF1 89473 114 - 1 CUACAUCAUUCCCACUCGGAACUGGGCAACGAGUC------CAGAUGACGUUCAUUUGGGCCCAGCUCAAGCUCGCAGAGCAGCUUGGGCGGGUUGCGUAAGACGAGCCUGAAGGUGCCC ...((((.(((...((((....((.(((((..(((------((((((.....)))))))))((.(((((((((.((...)))))))))))))))))).))...))))...)))))))... ( -46.90) >DroGri_CAF1 108841 120 - 1 CUGCAUCAUUCACACUCGGAUCUGGGGAAUGAGGCGAUGAGCAGAUGACGUUCGUUUGGGCCCAGCUCGAGCUCGCAGAGCAGCUUGGGCGGGUUGCGCAAGACAAGCCUCAAGGUGCCC ..((.((((((...((((....)))))))))).))(((((((.......))))))).((((((.(((((((((.((...)))))))))))((.((((....).))).))....)).)))) ( -48.60) >DroWil_CAF1 79722 108 - 1 CUGCAUCA------UUCGGAGAUGAGCAACGAUCC------CAGAUGACGUUCGUUUGGGCCCAGCUCAAGCUCGCAGAGCAGCUUGGGUGGGUUACGCAAGACGAGCCUUAAAGUGCCC ..((((((------(..((.(((........))))------)..)))).((((((((.((((((.((((((((.((...)))))))))))))))).....)))))))).......))).. ( -39.90) >DroMoj_CAF1 73926 114 - 1 CUGCAUCCGUCACAUUCUGAGCUGGGCAACGAGUC------CAGAUGACGCUCGUUUGGGCCCAGCUCAAGCUCACAGAGCAGCUUGGGCGGGUUGCGUAAGACGAGCCUGAAGGUGCCC ..(((((.((((...((((..((.(....).))..------))))))))((((((((.(((((.(((((((((........)))))))))))))).....)))))))).....))))).. ( -52.60) >DroAna_CAF1 69361 108 - 1 CUACACCA------CUCUGAGAUGAGCAACGAUCC------UAGGUGACGUUCGUUCGGGCCCAGCUCGAGCUCUCAGAGUAGCUUGGGUGGGUUACGGAAGACGAGUCUAAAGGUUCCC ((((.(((------((((((((.((((.((.....------...))...))))((((((((...))))))))))))))))).....))))))....(....)..((..(....)..)).. ( -35.00) >consensus CUGCAUCA______CUCGGAGAUGAGCAACGAGCC______CAGAUGACGUUCGUUUGGGCCCAGCUCAAGCUCGCAGAGCAGCUUGGGCGGGUUACGCAAGACGAGCCUCAAGGUGCCC ..(((((.......(((......))).......................((((((((.(((((.((((((((((.....).)))))))))))))).....)))))))).....))))).. (-31.48 = -31.37 + -0.11)

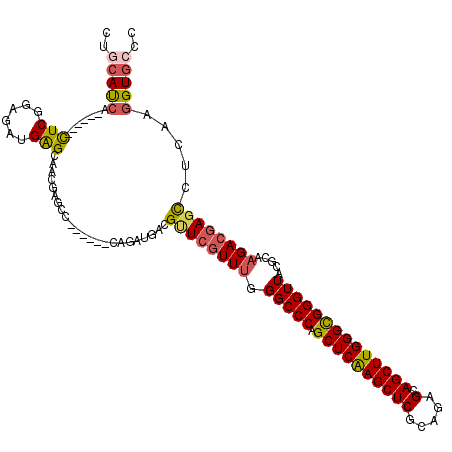
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:18:39 2006