
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
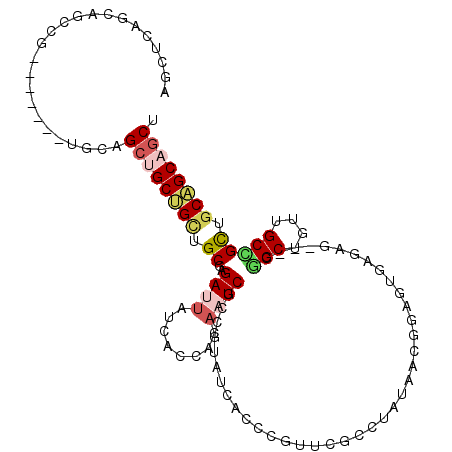
| Location | 16,451,912 – 16,452,023 |
| Length | 111 |
| Max. P | 0.998171 |

| Location | 16,451,912 – 16,452,023 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 67.62 |
| Mean single sequence MFE | -50.45 |
| Consensus MFE | -19.06 |
| Energy contribution | -19.70 |
| Covariance contribution | 0.64 |
| Combinations/Pair | 1.29 |
| Mean z-score | -3.11 |
| Structure conservation index | 0.38 |
| SVM decision value | 3.02 |
| SVM RNA-class probability | 0.998171 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 16451912 111 + 27905053 GGCUCAGCGGCCGCCGCUGCUGCAGCUGCUGCUGCGAGAUUAUCGCCCCAAUCGGCCUAUCACGCGUUCGCGUAUAACGGAGUGGGAG---CGGCUGUUGCCGCUGCAGCAGCU (((((((((((....))))))).))))((((((((...........((((.((.(..(((.((((....))))))).).)).))))((---((((....)))))))))))))). ( -58.70) >DroPse_CAF1 7550 99 + 1 --------AGCAG-------UUCGGCUGCAGCCGCGAGACUGUCACCUCCCUCGCCGUAUCAUCCGUUUGCCUAUAAUGGCGUCGGAGCAGCCGCUGUAGCGGCUGCUGCAGCU --------.....-------...(((((((((.(((((............))))).))....((((..((((......)))).))))((((((((....))))))))))))))) ( -47.10) >DroGri_CAF1 10232 104 + 1 AUUCGAGCAGCGG-------U---GGAGCCGUGGCUCGACUAUCACCACAAUCGCCGUAUCAUCCUUUUGGCUAUAAUGGUGUGAGUGCUGCGGCUGUUGCUGUUGCCGCGGCC ...(((((.((((-------.---....)))).)))))(((..(((((..((.((((...........)))).))..)))))..)))((((((((..........)))))))). ( -43.70) >DroSec_CAF1 7962 110 + 1 GGCUCAGCGGCCGCCGCUGCUGCAGCUGCUGCUGCGAGAUUAUCGCCACAAUCGGCGUAUCACGCGUUCACGUAUA-CGGAGUGAGAG---CGGCUGUUGCCGCUGCAGCAGCU .((.((((((...))))))..))(((((((((.(((.(((...((((......)))).))).))).(((((.....-....)))))((---((((....))))))))))))))) ( -56.90) >DroYak_CAF1 8620 111 + 1 AGUUCAGCGGCCGCCGCUGCUGCAGCUGCUGCUGCGAGAUUAUCGCCCCAAUCGGCCUAUCACGCAUUCGCGUAUAACGGAGUGGGGG---CGGCUGUUGCCGCAGCAGCAGCA ....(((((((....)))))))..((((((((((((.(((..(((((((..((.(..(((.((((....))))))).).))...))))---)))..)))..)))))))))))). ( -61.90) >DroMoj_CAF1 7947 101 + 1 AGACGAGUAG----------U---GCGGCUGGAGCACGAUUAUCACCACAAUCGCCGUAUCAUCCAUUUAGCUAUAAUGGUGUUAAUGCGGCCGCUGUGGCAGUCGCAGCGGCU .....(((..----------(---(((((((((.........)).(((((...(((((((((.(((((.......)))))))...)))))))...)))))))))))))...))) ( -34.40) >consensus AGCUCAGCAGCCG_______UGCAGCUGCUGCUGCGAGAUUAUCACCACAAUCGCCGUAUCACCCGUUCGCCUAUAACGGAGUGAGAG___CGGCUGUUGCCGCUGCAGCAGCU ........................((((((((.((..((((........)))).......................................(((....))))).)))))))). (-19.06 = -19.70 + 0.64)


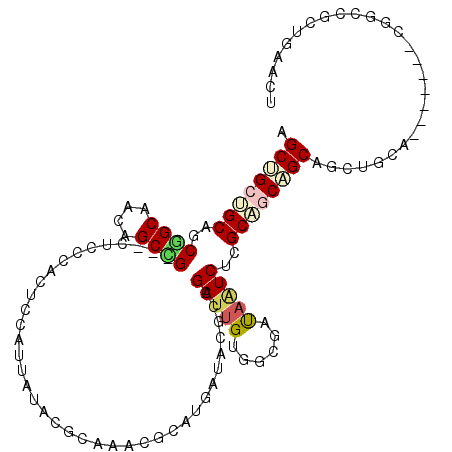
| Location | 16,451,912 – 16,452,023 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 67.62 |
| Mean single sequence MFE | -47.59 |
| Consensus MFE | -18.99 |
| Energy contribution | -19.55 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.94 |
| Structure conservation index | 0.40 |
| SVM decision value | 2.91 |
| SVM RNA-class probability | 0.997704 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 16451912 111 - 27905053 AGCUGCUGCAGCGGCAACAGCCG---CUCCCACUCCGUUAUACGCGAACGCGUGAUAGGCCGAUUGGGGCGAUAAUCUCGCAGCAGCAGCUGCAGCAGCGGCGGCCGCUGAGCC (((((((((((((((....))))---))((((.((.(((.(((((....)))))...))).)).))))((((.....)))).)))))))))((..((((((...)))))).)). ( -57.20) >DroPse_CAF1 7550 99 - 1 AGCUGCAGCAGCCGCUACAGCGGCUGCUCCGACGCCAUUAUAGGCAAACGGAUGAUACGGCGAGGGAGGUGACAGUCUCGCGGCUGCAGCCGAA-------CUGCU-------- .((((((((((((((....))))))))((((..(((......)))...)))).....(.((((((..(....)..)))))).).))))))....-------.....-------- ( -48.50) >DroGri_CAF1 10232 104 - 1 GGCCGCGGCAACAGCAACAGCCGCAGCACUCACACCAUUAUAGCCAAAAGGAUGAUACGGCGAUUGUGGUGAUAGUCGAGCCACGGCUCC---A-------CCGCUGCUCGAAU .((.(((((..........))))).)).....((((((....(((.............)))....))))))....((((((..(((....---.-------)))..)))))).. ( -38.32) >DroSec_CAF1 7962 110 - 1 AGCUGCUGCAGCGGCAACAGCCG---CUCUCACUCCG-UAUACGUGAACGCGUGAUACGCCGAUUGUGGCGAUAAUCUCGCAGCAGCAGCUGCAGCAGCGGCGGCCGCUGAGCC (((((((((((((((....))))---)).((((....-.....))))..(((.(((.(((((....)))))...))).))).)))))))))((..((((((...)))))).)). ( -53.40) >DroYak_CAF1 8620 111 - 1 UGCUGCUGCUGCGGCAACAGCCG---CCCCCACUCCGUUAUACGCGAAUGCGUGAUAGGCCGAUUGGGGCGAUAAUCUCGCAGCAGCAGCUGCAGCAGCGGCGGCCGCUGAACU .((((((((((((((....)))(---((((...((.(((.(((((....)))))...))).))..))))).........))))))))))).....((((((...)))))).... ( -56.00) >DroMoj_CAF1 7947 101 - 1 AGCCGCUGCGACUGCCACAGCGGCCGCAUUAACACCAUUAUAGCUAAAUGGAUGAUACGGCGAUUGUGGUGAUAAUCGUGCUCCAGCCGC---A----------CUACUCGUCU .(((((((.(.....).)))))))........(((((((.......))))).))....(((((..((((((.....((.((....)))))---)----------))))))))). ( -32.10) >consensus AGCUGCUGCAGCGGCAACAGCCG___CUCCCACUCCAUUAUACGCAAACGCAUGAUACGCCGAUUGUGGCGAUAAUCUCGCAGCAGCAGCUGCA_______CGGCCGCUGAACU .((((((((..((((....))))......................................(((((......)))))..))))))))........................... (-18.99 = -19.55 + 0.56)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:18:12 2006