
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 16,400,618 – 16,400,806 |
| Length | 188 |
| Max. P | 0.998512 |

| Location | 16,400,618 – 16,400,731 |
|---|---|
| Length | 113 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 82.31 |
| Mean single sequence MFE | -23.08 |
| Consensus MFE | -17.83 |
| Energy contribution | -17.75 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.19 |
| Mean z-score | -1.28 |
| Structure conservation index | 0.77 |
| SVM decision value | -0.03 |
| SVM RNA-class probability | 0.516929 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
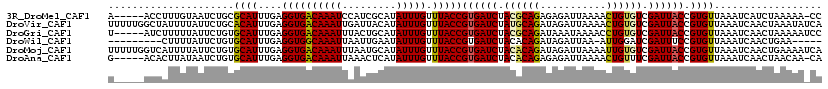
>3R_DroMel_CAF1 16400618 113 + 27905053 A-----ACCUUUGUAAUCUGCGCAUUUGAGGUGACAAAUCCAUCGCAUAUUUGUUUACCGUGAUCUACGCAGAGAGAUUAAAACUGUGUCGAUUACCGUGUUAAAUCAUCUAAAAA-CC .-----.....((......((((......((((((((((.........))))).)))))((((((.((((((...........)))))).)))))).)))).....))........-.. ( -23.30) >DroVir_CAF1 9652 119 + 1 UUUUUGGCUAUUUUAUUCUGCACAUUUGAGGUGACAAAUUGAUUACAUAUUUGUUUACCGUGAUCUAUGCAGAUAGAUUAAAACUGUGUCGAUUACCGUGUUAAAUCAACUAAAUAUCA ..(((((....((((....((((......((((((((((((....)).))))).)))))((((((.((((((...........)))))).)))))).))))))))....)))))..... ( -23.70) >DroGri_CAF1 6890 114 + 1 U-----AUCUUUUUAUUCUGUGCAUUUGAGGUGACAAAUUUACUGCAUAUUUGUUUACCGUGAUCUACGCAGAUAAAUAAAACCUGUGUCGAUUACCGUGUUAAAUCAACUAAAAAUCC .-----...((((((.....((.((((((((((((((((.........))))).)))))((((((.((((((...........)))))).))))))....))))))))..))))))... ( -23.60) >DroWil_CAF1 11459 104 + 1 ---------CUUUUAUUCUGUGCAUUUGAGGUGGCAAAUUAAUUGAAUAUUUGUUUACCGUGAUCUACACAGAUAGAUUAA-AUUGGAUCGAUUUCCGUGUUAAAUCAACUGAA----- ---------.......((((((.(((...((((((((((.........))))).)))))..)))...))))))..((((.(-((((((......)))).))).)))).......----- ( -20.50) >DroMoj_CAF1 8264 119 + 1 UUUUUGGUCAUUUUAUUCUGUGCAUUUGAGGUGACAAAUUUAAUGCAUAUUUGUUUACCGUGAUCUACACAGAUAGAUUAAAAUUGUGUCGAUUACCGUGUUAAAUCAACUGAAAAUCA ((((..((...((((...((((((((...(....)......))))))))......(((.((((((.((((((...........)))))).)))))).))).))))...))..))))... ( -25.10) >DroAna_CAF1 6691 113 + 1 G-----ACACUUAUAAUCUGUGCAUUUGAGGUGACAAAUUAAACUCAUAUUUGUUUACCGUGAUCUACACAGAGAGAUUAAAACUGUUUCGAUUACCGUGUUAAAUCAACUAACAA-CA (-----((((......((((((.(((...((((((((((.........))))).)))))..)))...))))))(((((.......))))).......)))))..............-.. ( -22.30) >consensus U_____ACCAUUUUAUUCUGUGCAUUUGAGGUGACAAAUUAAUUGCAUAUUUGUUUACCGUGAUCUACACAGAUAGAUUAAAACUGUGUCGAUUACCGUGUUAAAUCAACUAAAAA_CA .....................((((....((((((((((.........))))).)))))((((((.((((((...........)))))).)))))).)))).................. (-17.83 = -17.75 + -0.08)
| Location | 16,400,692 – 16,400,806 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 73.38 |
| Mean single sequence MFE | -29.33 |
| Consensus MFE | -14.97 |
| Energy contribution | -14.91 |
| Covariance contribution | -0.05 |
| Combinations/Pair | 1.21 |
| Mean z-score | -3.13 |
| Structure conservation index | 0.51 |
| SVM decision value | 3.13 |
| SVM RNA-class probability | 0.998512 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
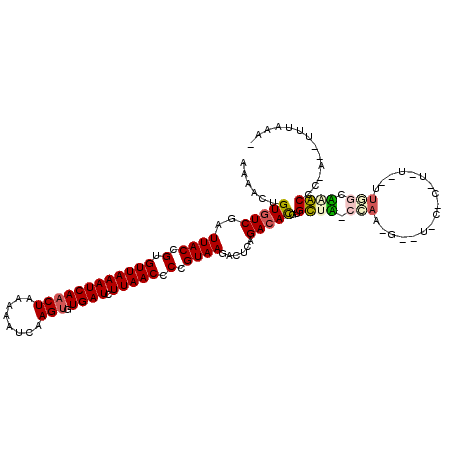
>3R_DroMel_CAF1 16400692 114 + 27905053 AAAACUGUGUCGAUUACCGUGUUAAAUCAUCUAAAAA-CCAGUGUGAUAUUAACCCCGUAAGACUCAGACAUGUGCUG-CCAAAGACUACACUU-U--UUGGCCAGCUC-GCUUUGAAGC ....(((.(((...(((.(.((((((((((((.....-..)).))))).))))).).))).))).)))....(.((((-(((((((......))-)--)))).)))).)-(((....))) ( -28.20) >DroVir_CAF1 9731 99 + 1 AAAACUGUGUCGAUUACCGUGUUAAAUCAACUAAAUAUCAAGUGUGAUCUUAACCCCGUAAGACUCAGACACGUGUUUGCU--------------U--UUAGCAGACGC-A--UUUCG-- ......(((((..((((.(.((((((((((((........))).)))).))))).).))))......)))))((((((((.--------------.--...))))))))-.--.....-- ( -27.20) >DroPse_CAF1 4015 117 + 1 AAAACUGUGUCGAUUACCGUGUUAAAUCAACUGAAUAUCAAGUGUGAUCUUAACCCCGUAAGACUCAGACACGUGCUA-CUAACGGAUUCCCAU-UCCGUUAGUCGCCC-GAUUCUAUAU ....(((.(((...(((.(.((((((((((((........))).)))).))))).).))).))).)))...((.((.(-((((((((.......-))))))))).)).)-)......... ( -34.00) >DroGri_CAF1 6964 100 + 1 AAACCUGUGUCGAUUACCGUGUUAAAUCAACUAAAAAUCCAGUGUGAUCUUAACCCCGUAAGACUCAGACACGUGUUUGCCA-------------U--UUGGCAAACGC-A--UUAGA-- ......(((((..((((.(.((((((((((((........))).)))).))))).).))))......)))))(((((((((.-------------.--..)))))))))-.--.....-- ( -30.30) >DroAna_CAF1 6765 111 + 1 AAAACUGUUUCGAUUACCGUGUUAAAUCAACUAACAA-CAAGUGUGAUUUUAACCCCGUAAGACUCAGACACGUGUUG-CCAAAGAUGAAAAUAUC--UUGGUCAACCACAC-----AAC ....(((..((...(((.(.((((((((((((.....-..))).))).)))))).).))).))..)))....((((((-((((.((((....))))--)))).))).)))..-----... ( -22.30) >DroPer_CAF1 4080 117 + 1 AAAACUGUGUCGAUUACCGUGUUAAAUCAACUGAAUAUCAAGUGUGAUCUUAACCCCGUAAGACUCAGACACGUGCUA-CUAACGGAUUCCCAU-UCCGUUAGUCGCCC-GAUUCUAUAU ....(((.(((...(((.(.((((((((((((........))).)))).))))).).))).))).)))...((.((.(-((((((((.......-))))))))).)).)-)......... ( -34.00) >consensus AAAACUGUGUCGAUUACCGUGUUAAAUCAACUAAAAAUCAAGUGUGAUCUUAACCCCGUAAGACUCAGACACGUGCUA_CCAA_G__U_C_C_U_U__UUGGCAAACCC_A__UUUAAA_ ......(((((..((((.(.((((((((((((........))).)))).))))).).))))......)))))..((((.(((.................))).))))............. (-14.97 = -14.91 + -0.05)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:17:42 2006