
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 2,317,507 – 2,317,648 |
| Length | 141 |
| Max. P | 0.995192 |

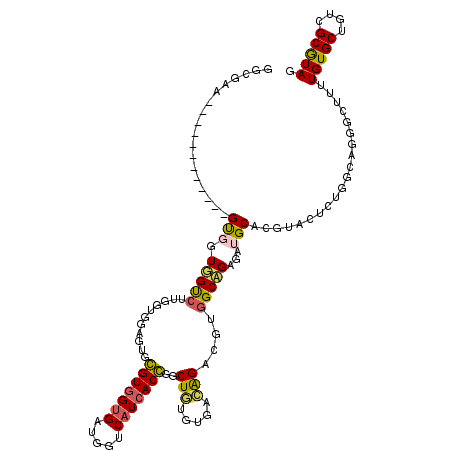
| Location | 2,317,507 – 2,317,627 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 74.60 |
| Mean single sequence MFE | -46.65 |
| Consensus MFE | -17.77 |
| Energy contribution | -17.05 |
| Covariance contribution | -0.72 |
| Combinations/Pair | 1.30 |
| Mean z-score | -2.39 |
| Structure conservation index | 0.38 |
| SVM decision value | 1.06 |
| SVM RNA-class probability | 0.907821 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
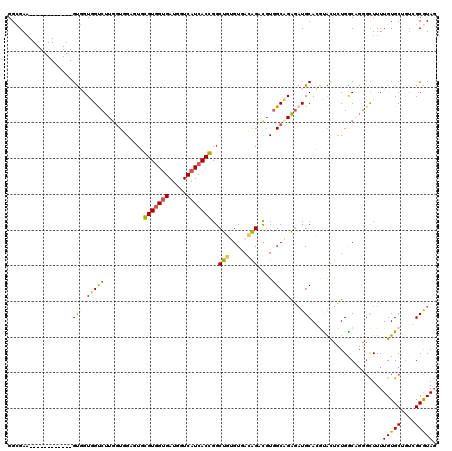
>3R_DroMel_CAF1 2317507 120 - 27905053 GGCGAAGGCCACCUAGUGGUGGUGGUCUUGUUGGAUUGGGUGGUGAUGGUCAUCACCGGCUGUGUGACAGACGUGGCCGAGAUGCACAUAUUUUGACACGGCUUUUGUGCUGUGGCGUAG (((.(((((((((.......))))))))))))......(((((((.....)))))))(((..(((.....)))..)))...((((.((.....)).((((((......)))))))))).. ( -49.50) >DroPse_CAF1 27823 108 - 1 GGGGAA------------GUUGUGGUCUUGGUUGUGUUCGUGGUGAUGGUCAUCACGGGCUUGGUCAGGGCUGUGGCCGAGAUGCAGGUACUCUGGCAGGGCUUUUGUGCAGUCGCAUAG ..((((------------((((..(((((((..(.((((((((((.....)))))))))).)..)))))))..).((((((.((....))))).)))..)))))))((((....)))).. ( -41.00) >DroGri_CAF1 94685 108 - 1 GGCGAU------------GCGGUUGUCUUUGUGGUGUUCGUCGUGACGGUCAUCACGGGCUGUGUGAGAGCUGUCGCAGAUUUGCAGCUGCUAUGGCAGCCUUUUUGUGCGGUCGCGUAC .(((((------------(((((((((...(..((((..(((((((((((..(((((.....)))))..)))))))).)))..))).)..)...))))))).......)).))))).... ( -43.71) >DroEre_CAF1 28793 120 - 1 GGCGAAGGCCACCUAGUGGUGGUGGCCUUGGUGGAUUGGGUGGUGAUGGUCAUCACCGGCUGUGUGACAGACCUGGCAGAGAUGCACGUAUUCUGACCGGGCUUUUGUGCUGUAGCGUAG .((.(((((((((.......))))))))).))......(((((((.....))))))).((((((..(.((.((((((((((((....)).))))).))))))).)..)))...))).... ( -51.20) >DroYak_CAF1 22891 120 - 1 GGCGAAGGCCACCUAGUGGUGGUUGUCUUGGUGGAUUGGGUGGCGAUGGUCAUCACCGGCUGUGUGACAGACGUGGCAGGGAUGCACGCAUUCUGGCCGGGCUUUUGUGCUUGGGCGUAG .((((..((((((....))))))..))...(((.(((.((((((....)))))).((.((..(((.....)))..)).))))).))))).....(.((((((......)))))).).... ( -48.80) >DroPer_CAF1 27656 108 - 1 GGGGAA------------GUUGUGGUCUUGGUUGUGUUCGUGGUGAUGGUCAUCACGGGCUUGGUCAGGGCUGUGGCCGAGAUGCAGGUGCUCUGGCAGGGCUUUUGUGCGGUCGCAUAG ......------------.....((((((((..(.((((((((((.....)))))))))).)..))))))))(((((((....(((((.((((.....)))).))))).))))))).... ( -45.70) >consensus GGCGAA____________GUGGUGGUCUUGGUGGAGUGCGUGGUGAUGGUCAUCACCGGCUGUGUGACAGACGUGGCAGAGAUGCACGUACUCUGGCAGGGCUUUUGUGCUGUCGCGUAG ..................(((.(((((...........(((((((.....)))))))..(((.....)))....)))))...)))....................(((((....))))). (-17.77 = -17.05 + -0.72)

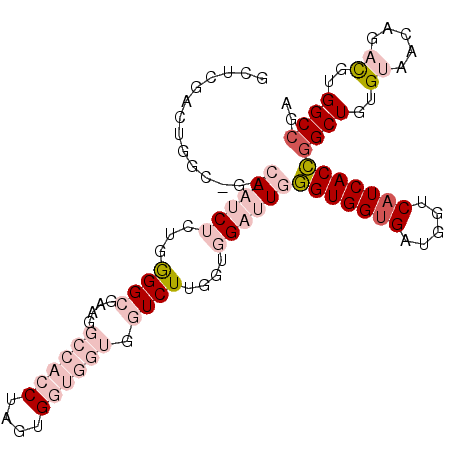
| Location | 2,317,547 – 2,317,648 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 104 |
| Reading direction | reverse |
| Mean pairwise identity | 75.21 |
| Mean single sequence MFE | -42.22 |
| Consensus MFE | -21.35 |
| Energy contribution | -24.55 |
| Covariance contribution | 3.20 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.89 |
| Structure conservation index | 0.51 |
| SVM decision value | 2.55 |
| SVM RNA-class probability | 0.995192 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
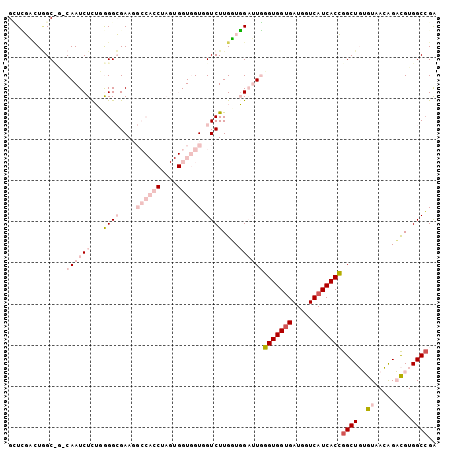
>3R_DroMel_CAF1 2317547 101 - 27905053 GCUCGACUGGC---CAAUCUUUUGGGCGAAGGCCACCUAGUGGUGGUGGUCUUGUUGGAUUGGGUGGUGAUGGUCAUCACCGGCUGUGUGACAGACGUGGCCGA ..(((((((.(---(((....)))).))(((((((((.......))))))))))))))....(((((((.....)))))))(((..(((.....)))..))).. ( -45.00) >DroPse_CAF1 27863 92 - 1 GAUACUUUAGCUGGCAAUCUCAUAGGGGAA------------GUUGUGGUCUUGGUUGUGUUCGUGGUGAUGGUCAUCACGGGCUUGGUCAGGGCUGUGGCCGA ...........((((..((((....)))).------------...(..(((((((..(.((((((((((.....)))))))))).)..)))))))..).)))). ( -34.60) >DroSec_CAF1 23275 104 - 1 GCUCGACUGGCCGCCAAUCUCUGGGGCGAAGGCCACCUAGUGGUGGUGGUCUUGUUGGAUUGGGUGGUGAUGGUCAUCACCGGCUGUGUAACAGACGUGGCCGA ..(((((.((((((((.((.(((((((....))).)).)).))))))))))..)))))....(((((((.....)))))))(((..(((.....)))..))).. ( -51.20) >DroSim_CAF1 23451 104 - 1 GCUCGACUGGCCACCAAUCUUUGGGGCGAAGGCCACCUAGUGGUGGUAGUCUUGUUGGAUUGGGUGGUGAUGGUCAUCACCGGCUGUGUAACAGACGUGGCCGA ((..(((((.((((((......(((((....))).))...)))))))))))..)).......(((((((.....)))))))(((..(((.....)))..))).. ( -47.40) >DroYak_CAF1 22931 101 - 1 GUUUGCAUGGC---CAAUCUUUGGGGCGAAGGCCACCUAGUGGUGGUUGUCUUGGUGGAUUGGGUGGCGAUGGUCAUCACCGGCUGUGUGACAGACGUGGCAGG (((((((((((---(((((((..(((((...((((((....)))))))))))..).))))).((((((....))))))...)))))))...)))))........ ( -40.50) >DroPer_CAF1 27696 92 - 1 GAUACUUUCGCUGGCAAUCUCAUAGGGGAA------------GUUGUGGUCUUGGUUGUGUUCGUGGUGAUGGUCAUCACGGGCUUGGUCAGGGCUGUGGCCGA ...........((((..((((....)))).------------...(..(((((((..(.((((((((((.....)))))))))).)..)))))))..).)))). ( -34.60) >consensus GCUCGACUGGC_G_CAAUCUCUGGGGCGAAGGCCACCUAGUGGUGGUGGUCUUGGUGGAUUGGGUGGUGAUGGUCAUCACCGGCUGUGUAACAGACGUGGCCGA ..............((((((...((((....((((((....)))))).))))....))))))(((((((.....)))))))((((..((.....))..)))).. (-21.35 = -24.55 + 3.20)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:47:50 2006