
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 2,265,588 – 2,265,718 |
| Length | 130 |
| Max. P | 0.942769 |

| Location | 2,265,588 – 2,265,691 |
|---|---|
| Length | 103 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | reverse |
| Mean pairwise identity | 75.00 |
| Mean single sequence MFE | -40.53 |
| Consensus MFE | -18.97 |
| Energy contribution | -21.08 |
| Covariance contribution | 2.11 |
| Combinations/Pair | 1.30 |
| Mean z-score | -2.29 |
| Structure conservation index | 0.47 |
| SVM decision value | 1.33 |
| SVM RNA-class probability | 0.942769 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
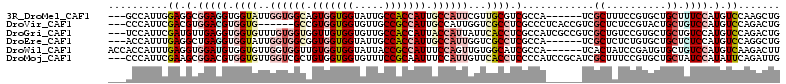
>3R_DroMel_CAF1 2265588 103 - 27905053 ---GCCAUUGGAGGCGGAGGUGGUAUUGGUGGCAGUGGUGGUAUUGCCACCAUUGCCAUUCGUUGCGUCGCCA------UCGCUUUCCGUGCUGCUUUCCAUGUCCAAGCUG ---((.((.(((((((..(((((((..((((((((((((((.....))))))))))))))...)))..)))).------.))))))).)))).((((.........)))).. ( -45.60) >DroVir_CAF1 2771 103 - 1 ---CCCAUUCGACGUGGACGUGGUG------GCCGUGGUGGUGUUGCCGCCAUUGCCAUUGGUCGCCUCGCCCUCACCGUCGCUCUCCGUACUGCUGUCCAUGUCCAGACUG ---.......((((((((((..(((------((.(((((((.....))))))).)))...((..((..((.......))..))...))..))..).)))))))))....... ( -41.50) >DroGri_CAF1 2321 109 - 1 ---UCCAUUCGAUGUUGAGGUGGUGUUUGUGGUGGUUGUGGUGUUGCCACCAUUACCAUUAUUCACCUCGCCAUCGCCGUCGCUGUCCGUGCUGCUGUCCAUGUCCAGACUG ---......(((((.((((((((.....((((((((.((((((....)))))).)))))))))))))))).)))))......(((..((((........))))..))).... ( -40.60) >DroEre_CAF1 2243 103 - 1 ---ACCAUUUGAGGCUGAGGUGGUAUUGGUGGCGGUGGUGGUAUUGCCACCAUUGCCAUUGGUCGCCUCGCCA------UCGCUCUCUGUGCUGCUCUCCAUGUCCAGGCUG ---.......(((((.((((((..((..(((((((((((((.....)))))))))))))..))))))))))).------))((...(((..(..........)..))))).. ( -46.80) >DroWil_CAF1 1806 106 - 1 ACCACCAUUUGAGGUGGAUGUGGUGUUGGUGGUUGUGGUGGUAUUACCGCCAUUUCCAGUUGUGGCAUCGCCA------UCACUAUCCGAUGUGCUGUCCAUGUCAAGACUU .......(((((.(((((((..(..((((.((..(((((((.....)))))))..))(((.(((((...))))------).)))..))))..)..))))))).))))).... ( -41.70) >DroMoj_CAF1 2545 109 - 1 ---CCCAUUCGAAGCGGACGUGGUGUUGGUCGCUGUGGUGGUGUUUCCGCAAUUUCCAUUGUUCACCUCCCCAUCCGCAUCGCUUUCCGUGCUGCUAUCCAUAUUCAGAUUG ---..........(((((.(((((((.((....((.((.((((.....(((((....))))).)))).)).)).)))))))))..)))))...................... ( -27.00) >consensus ___ACCAUUCGAGGCGGAGGUGGUGUUGGUGGCGGUGGUGGUAUUGCCACCAUUGCCAUUGGUCGCCUCGCCA______UCGCUCUCCGUGCUGCUGUCCAUGUCCAGACUG ..........((.(.(((((.((((..((((((.(((((((.....))))))).))))))...)))).)............((..........)).)))).).))....... (-18.97 = -21.08 + 2.11)
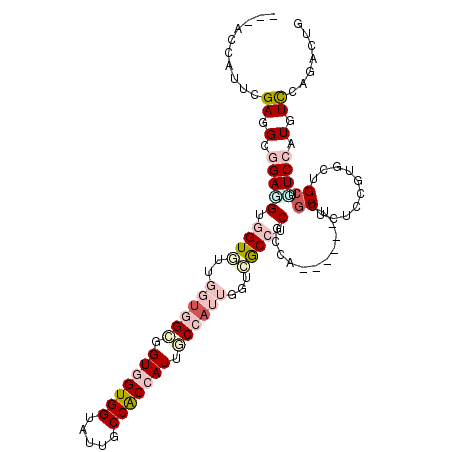
| Location | 2,265,622 – 2,265,718 |
|---|---|
| Length | 96 |
| Sequences | 6 |
| Columns | 102 |
| Reading direction | reverse |
| Mean pairwise identity | 76.53 |
| Mean single sequence MFE | -38.15 |
| Consensus MFE | -17.84 |
| Energy contribution | -19.53 |
| Covariance contribution | 1.70 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.39 |
| Structure conservation index | 0.47 |
| SVM decision value | 1.20 |
| SVM RNA-class probability | 0.929615 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 2265622 96 - 27905053 GUCGGACCAAACUUUCCUAGUCAUCGG------GCCAUUGGAGGCGGAGGUGGUAUUGGUGGCAGUGGUGGUAUUGCCACCAUUGCCAUUCGUUGCGUCGCC (.((.((((((((..(((.(((..(((------....)))..)))..))).))).)))))(((((((((((.....)))))))))))........)).)... ( -38.20) >DroVir_CAF1 2811 90 - 1 GUCCGACCACACCUUGCGCGUCAUUGG------CCCAUUCGACGUGGACGUGGUG------GCCGUGGUGGUGUUGCCGCCAUUGCCAUUGGUCGCCUCGCC ...(((((((((..(.((((((.....------.......)))))).).)))(((------((.(((((((.....))))))).)))))))))))....... ( -38.20) >DroGri_CAF1 2361 96 - 1 GUCCGACCACACCUUACGCGUCAUUGG------UCCAUUCGAUGUUGAGGUGGUGUUUGUGGUGGUUGUGGUGUUGCCACCAUUACCAUUAUUCACCUCGCC ...(((....((((((.(((((.....------.......)))))))))))((((...((((((((.((((((....)))))).)))))))).))))))).. ( -34.20) >DroEre_CAF1 2277 96 - 1 GUCGGACCAAACUUUCCUAGUCAUAGG------ACCAUUUGAGGCUGAGGUGGUAUUGGUGGCGGUGGUGGUAUUGCCACCAUUGCCAUUGGUCGCCUCGCC .......((((...(((((....))))------)...)))).(((.((((((..((..(((((((((((((.....)))))))))))))..))))))))))) ( -47.80) >DroWil_CAF1 1840 102 - 1 AUCGGACCAUACUUUUCGUGUCAUGGGUCCACCACCAUUUGAGGUGGAUGUGGUGUUGGUGGUUGUGGUGGUAUUACCGCCAUUUCCAGUUGUGGCAUCGCC ...(((((((((.....))))....))))).(((((......)))))..(((((((((.(((..(((((((.....)))))))..))).)...)))))))). ( -42.40) >DroMoj_CAF1 2585 96 - 1 AUCCGACCACACUUUGCGCGUCAUUGG------CCCAUUCGAAGCGGACGUGGUGUUGGUCGCUGUGGUGGUGUUUCCGCAAUUUCCAUUGUUCACCUCCCC ...(((((((((....((((((.((((------.....))))....))))))))).))))))....((.((((.....(((((....))))).)))).)).. ( -28.10) >consensus GUCCGACCACACUUUCCGCGUCAUUGG______ACCAUUCGAGGCGGAGGUGGUGUUGGUGGCGGUGGUGGUAUUGCCACCAUUGCCAUUGGUCGCCUCGCC ....(((............)))...........................(.((((..((((((.(((((((.....))))))).))))))...)))).)... (-17.84 = -19.53 + 1.70)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:47:23 2006