
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 121,085 – 121,199 |
| Length | 114 |
| Max. P | 0.977575 |

| Location | 121,085 – 121,176 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 98 |
| Reading direction | forward |
| Mean pairwise identity | 82.04 |
| Mean single sequence MFE | -43.05 |
| Consensus MFE | -40.58 |
| Energy contribution | -40.30 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.12 |
| Mean z-score | -6.56 |
| Structure conservation index | 0.94 |
| SVM decision value | 1.50 |
| SVM RNA-class probability | 0.959581 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 121085 91 + 27905053 GA-AC-----CAGUCCUGGUGGAGCUCAAAUUGACUCUAGUAGGGAGUCCUU-UAAUGAGCGACUCCCUAACGGAGUCAGAUUGAGCUGCAAAGGAGC ..-..-----...((((..((.(((((((.((((((((..((((((((((((-....))).)))))))))..)))))))).))))))).)).)))).. ( -50.40) >DroVir_CAF1 31194 96 + 1 CG-ACGCGUGAAUUCCUGCCGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUC-ACGAGCGACUCCCUAACGGAGUCAGAUUGAAUUACAAAGGACC ..-..........((((...(.(((((((.((((((((..((((((((((((..-..))).)))))))))..)))))))).))))))).)..)))).. ( -40.50) >DroGri_CAF1 31374 97 + 1 UG-CCCCCGAACUUCCUGCUGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUUAACGAGCGACUCCCUAACGGAGUCAGAUUGAGUUACAAAGGACC ..-..........((((..((.(..((((.((((((((..((((((((((((.....))).)))))))))..)))))))).))))..).)).)))).. ( -39.60) >DroWil_CAF1 37116 93 + 1 UGAGA-----ACUUCCUGCGGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUAUAUAAGCGACUCCCUAACGGAGUCAGAUUGAAUUACAAAGGAAC .....-----..(((((...(.(((((((.((((((((..((((((((((((.....))).)))))))))..)))))))).))))))).)..))))). ( -42.70) >DroMoj_CAF1 26299 94 + 1 CU-ACGAGU---UUCCUCAUGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUUUUUUAACGACUCCCUAACGGAGUCAGAUUGAAUUUCAGAGGACC ..-......---.(((((.((.(((((((.((((((((..(((((((((.((......)).)))))))))..)))))))).))))))).))))))).. ( -43.00) >DroPer_CAF1 23437 92 + 1 CG-GG-----AUUUCCUGCAGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUUAUUGAGCGACUCCCUAACGGAGUCAGAUUGAAUUACCAAGGAAC ..-..-----..(((((...(.(((((((.((((((((..((((((((((((.....))).)))))))))..)))))))).))))))).)..))))). ( -42.10) >consensus CG_AC_____AAUUCCUGCUGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUUAAUGAGCGACUCCCUAACGGAGUCAGAUUGAAUUACAAAGGACC .............((((...(.(((((((.((((((((..((((((((((((.....))).)))))))))..)))))))).))))))).)..)))).. (-40.58 = -40.30 + -0.28)


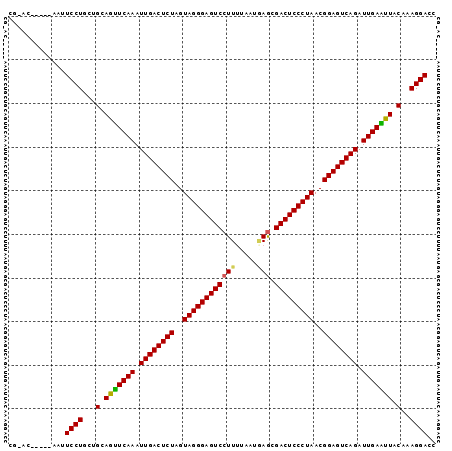
| Location | 121,085 – 121,176 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 98 |
| Reading direction | reverse |
| Mean pairwise identity | 82.04 |
| Mean single sequence MFE | -38.70 |
| Consensus MFE | -35.43 |
| Energy contribution | -35.77 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.13 |
| Mean z-score | -5.27 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.79 |
| SVM RNA-class probability | 0.977575 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 121085 91 - 27905053 GCUCCUUUGCAGCUCAAUCUGACUCCGUUAGGGAGUCGCUCAUUA-AAGGACUCCCUACUAGAGUCAAUUUGAGCUCCACCAGGACUG-----GU-UC ..((((.((.(((((((..((((((...(((((((((.((.....-.)))))))))))...))))))..))))))).))..))))...-----..-.. ( -40.70) >DroVir_CAF1 31194 96 - 1 GGUCCUUUGUAAUUCAAUCUGACUCCGUUAGGGAGUCGCUCGU-GAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCGGCAGGAAUUCACGCGU-CG ..(((((((((.(((((..((((((...(((((((((.((...-...)))))))))))...))))))..))))).))))).))))..........-.. ( -38.30) >DroGri_CAF1 31374 97 - 1 GGUCCUUUGUAACUCAAUCUGACUCCGUUAGGGAGUCGCUCGUUAAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCAGCAGGAAGUUCGGGGG-CA ..(((((((((..((((..((((((...(((((((((.((.......)))))))))))...))))))..))))..))))).)))).(((....))-). ( -37.90) >DroWil_CAF1 37116 93 - 1 GUUCCUUUGUAAUUCAAUCUGACUCCGUUAGGGAGUCGCUUAUAUAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCCGCAGGAAGU-----UCUCA .(((((..(((.(((((..((((((...(((((((((.(((.....))))))))))))...))))))..))))).)))...)))))..-----..... ( -37.80) >DroMoj_CAF1 26299 94 - 1 GGUCCUCUGAAAUUCAAUCUGACUCCGUUAGGGAGUCGUUAAAAAAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCAUGAGGAA---ACUCGU-AG ..(((((((...(((((..((((((...(((((((((.((......)).)))))))))...))))))..)))))...)).))))).---......-.. ( -38.90) >DroPer_CAF1 23437 92 - 1 GUUCCUUGGUAAUUCAAUCUGACUCCGUUAGGGAGUCGCUCAAUAAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCUGCAGGAAAU-----CC-CG .((((((((((.(((((..((((((...(((((((((.((.......)))))))))))...))))))..))))).))))).)))))..-----..-.. ( -38.60) >consensus GGUCCUUUGUAAUUCAAUCUGACUCCGUUAGGGAGUCGCUCAUUAAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCAGCAGGAAGU_____GU_CG ..(((((((((.(((((..((((((...(((((((((.((.......)))))))))))...))))))..))))).))))).))))............. (-35.43 = -35.77 + 0.33)


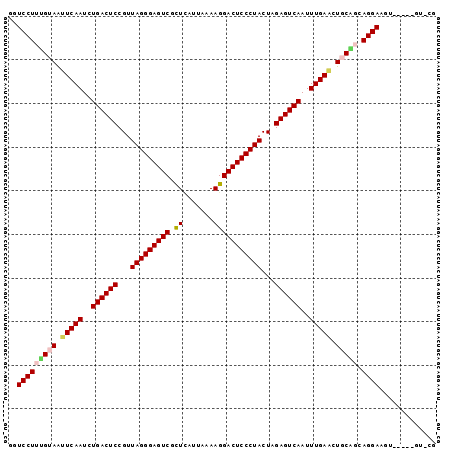
| Location | 121,097 – 121,199 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 74.97 |
| Mean single sequence MFE | -41.27 |
| Consensus MFE | -36.01 |
| Energy contribution | -35.73 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.14 |
| Mean z-score | -5.44 |
| Structure conservation index | 0.87 |
| SVM decision value | 1.07 |
| SVM RNA-class probability | 0.909776 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 121097 102 + 27905053 GUGGAGCUCAAAUUGACUCUAGUAGGGAGUCCUU-UAAUGAGCGACUCCCUAACGGAGUCAGAUUGAGCUGCAAAGGAGCGAC-ACAACA-----GAGGAGCA---------CUU-AAA .((.(((((((.((((((((..((((((((((((-....))).)))))))))..)))))))).))))))).)).(((.((..(-......-----..)..)).---------)))-... ( -48.30) >DroVir_CAF1 31211 110 + 1 CCGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUC-ACGAGCGACUCCCUAACGGAGUCAGAUUGAAUUACAAAGGACCCAG-ACAACG-----AGUAAAAAUGUGC-CAAGUG-ACA .((((((((((.((((((((..((((((((((((..-..))).)))))))))..)))))))).))))))).....((.(.((.-((....-----.)).....)).))-)..)))-... ( -39.90) >DroGri_CAF1 31391 103 + 1 CUGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUUAACGAGCGACUCCCUAACGGAGUCAGAUUGAGUUACAAAGGACCAAG-UCAACA-----AU-------GCC--CCAGUG-AUA ..(((.(((((.((((((((..((((((((((((.....))).)))))))))..)))))))).)))))........(((...)-))....-----.)-------)).--......-... ( -36.40) >DroWil_CAF1 37129 107 + 1 CGGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUAUAUAAGCGACUCCCUAACGGAGUCAGAUUGAAUUACAAAGGAACAACAACAACA-----AAAAGCCACGA------CCC-ACC .((((((((((.((((((((..((((((((((((.....))).)))))))))..)))))))).))))))).....(......).......-----....)))....------...-... ( -41.20) >DroMoj_CAF1 26313 113 + 1 AUGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUUUUUUAACGACUCCCUAACGGAGUCAGAUUGAAUUUCAGAGGACCCAG-ACAACG-----AAGAAGCGCGAGCACAAAUGCAAA .((((((((((.((((((((..(((((((((.((......)).)))))))))..)))))))).))))))..............-......-----.....((....)).....)))).. ( -41.20) >DroAna_CAF1 20275 106 + 1 GCGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUUAACGAGAGACUCCCUAACGGAGUCAGAUUGAAUUACAAAGGAGCGAG-ACAACGACUACGUGGUGGC---------CU---AA ..(.(((((((.((((((((..((((((((((((.....))).)))))))))..)))))))).))))))).)..(((.(((((-.......)).))).....)---------))---.. ( -40.60) >consensus CUGCAGUUCAAAUUGACUCUAGUAGGGAGUCCUUUUAACGAGCGACUCCCUAACGGAGUCAGAUUGAAUUACAAAGGACCAAG_ACAACA_____AAGAAGCA_G_______CUG_AAA ..(.(((((((.((((((((..((((((((((((.....))).)))))))))..)))))))).))))))).)............................................... (-36.01 = -35.73 + -0.28)

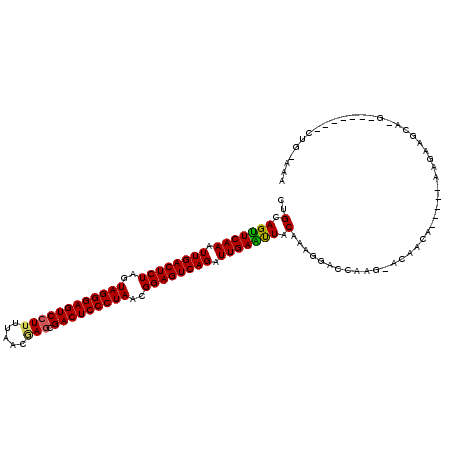
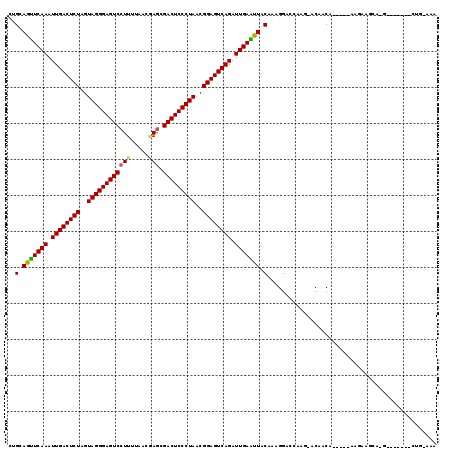
| Location | 121,097 – 121,199 |
|---|---|
| Length | 102 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 74.97 |
| Mean single sequence MFE | -37.17 |
| Consensus MFE | -30.26 |
| Energy contribution | -30.40 |
| Covariance contribution | 0.14 |
| Combinations/Pair | 1.12 |
| Mean z-score | -3.75 |
| Structure conservation index | 0.81 |
| SVM decision value | 1.70 |
| SVM RNA-class probability | 0.972654 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 121097 102 - 27905053 UUU-AAG---------UGCUCCUC-----UGUUGU-GUCGCUCCUUUGCAGCUCAAUCUGACUCCGUUAGGGAGUCGCUCAUUA-AAGGACUCCCUACUAGAGUCAAUUUGAGCUCCAC ...-...---------........-----....((-(..((......))(((((((..((((((...(((((((((.((.....-.)))))))))))...))))))..))))))).))) ( -38.00) >DroVir_CAF1 31211 110 - 1 UGU-CACUUG-GCACAUUUUUACU-----CGUUGU-CUGGGUCCUUUGUAAUUCAAUCUGACUCCGUUAGGGAGUCGCUCGU-GAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCGG ...-.(((..-(.(((........-----...)))-)..)))....((((.(((((..((((((...(((((((((.((...-...)))))))))))...))))))..))))).)))). ( -37.60) >DroGri_CAF1 31391 103 - 1 UAU-CACUGG--GGC-------AU-----UGUUGA-CUUGGUCCUUUGUAACUCAAUCUGACUCCGUUAGGGAGUCGCUCGUUAAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCAG ...-....((--(((-------..-----.(....-)...))))).((((..((((..((((((...(((((((((.((.......)))))))))))...))))))..))))..)))). ( -36.30) >DroWil_CAF1 37129 107 - 1 GGU-GGG------UCGUGGCUUUU-----UGUUGUUGUUGUUCCUUUGUAAUUCAAUCUGACUCCGUUAGGGAGUCGCUUAUAUAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCCG ((.-.(.------..(..((....-----.))..)................(((((..((((((...(((((((((.(((.....))))))))))))...))))))..))))))..)). ( -38.00) >DroMoj_CAF1 26313 113 - 1 UUUGCAUUUGUGCUCGCGCUUCUU-----CGUUGU-CUGGGUCCUCUGAAAUUCAAUCUGACUCCGUUAGGGAGUCGUUAAAAAAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCAU ..((((...(..((((.((.....-----....))-.))))..).......(((((..((((((...(((((((((.((......)).)))))))))...))))))..))))).)))). ( -37.70) >DroAna_CAF1 20275 106 - 1 UU---AG---------GCCACCACGUAGUCGUUGU-CUCGCUCCUUUGUAAUUCAAUCUGACUCCGUUAGGGAGUCUCUCGUUAAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCGC ..---.(---------((.((.(((....))).))-...))).....(((.(((((..((((((...(((((((((.((.......)))))))))))...))))))..))))).))).. ( -35.40) >consensus UUU_CAG_______C_UGCUUCUU_____CGUUGU_CUCGGUCCUUUGUAAUUCAAUCUGACUCCGUUAGGGAGUCGCUCAUUAAAAGGACUCCCUACUAGAGUCAAUUUGAACUGCAG ..............................................((((.(((((..((((((...(((((((((.((.......)))))))))))...))))))..))))).)))). (-30.26 = -30.40 + 0.14)

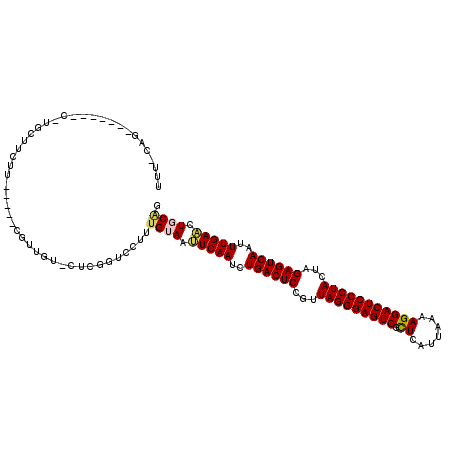
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:29:55 2006