
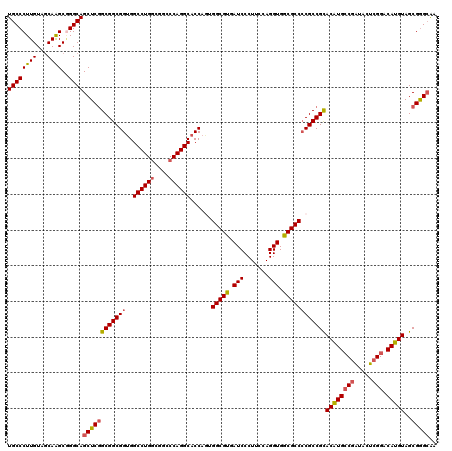
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 15,763,435 – 15,763,555 |
| Length | 120 |
| Max. P | 0.972863 |

| Location | 15,763,435 – 15,763,555 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.42 |
| Mean single sequence MFE | -60.08 |
| Consensus MFE | -55.26 |
| Energy contribution | -55.58 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.57 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.70 |
| SVM RNA-class probability | 0.972863 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
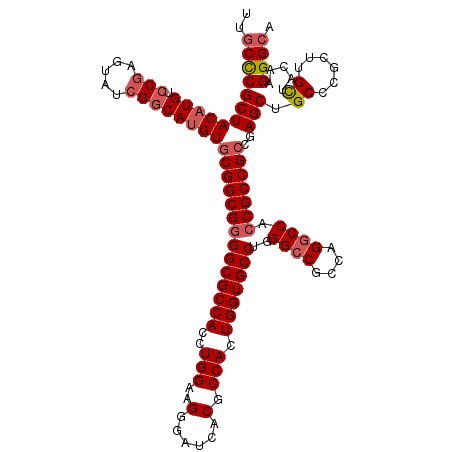
>3R_DroMel_CAF1 15763435 120 + 27905053 UGCCCUUGUAGCAAGCGGGCAGCUCGGCGGCGGUGGCCUGGCGGCCCAGGCACCAAUGGCGUGAUCCCUUCCAGGUGGCGCCCCGCCGCACAUGCCGAUACUCGGACAUGUAGCGAGCAA (((((((......)).)))))((((((((((((..((((((....))))))......(((((.(((.......))).))))))))))))((((((((.....))).)))))..))))).. ( -64.20) >DroSec_CAF1 20634 120 + 1 UGCCCUUGUAGCAAGCGGGCAGCUCGGCGGCGGUGGCCUGGCGGCCCAGGCACCAGUGGCGUGAUCCCUUCCAGGUGGCGCCCCGCCGUACAUGCCGAUACUCGGACAUGUAGCGGGCAA (((((((......)).)))))((((((((((((((.(((((....))))))))).(.(((((.(((.......))).))))).))))))((((((((.....))).)))))..))))).. ( -61.30) >DroSim_CAF1 20613 120 + 1 UGCCCUUGUAACAAGCAGGCAGCUCGGCGGCGGUGGCCUGGCGGCCCAGGCACCAAUGGCGUGAUCCCUUCCAGGUGGCGCCCCGCCGCACAUGCCUAUACUCGGACAUGUAGCGGGCAA ((((((((...))))..))))((((((((((((..((((((....))))))......(((((.(((.......))).))))))))))))(((((((.......)).)))))..))))).. ( -58.30) >DroEre_CAF1 22819 120 + 1 UGCCCUUGUAGCAGGCGGGCAGCUCGGCGGCGGUGGCCUGGCGGCCCAGGCACCAGUGGCGUGAUCCCUUCCAGGUGGCGCCCCGCCGCACGUGCCGAUACUCGGACAUGUAGCGGGCAA (((((...........)))))((((((((((((((.(((((....))))))))).(.(((((.(((.......))).))))).))))))((((((((.....))).)))))..))))).. ( -62.90) >DroPer_CAF1 21106 120 + 1 UGCCCUUGUAGCAGGCUGGCAGCUCAGCGGCGGAGGCCUGUCGCCCCAGGCACCAGUGGCGCGAUCCCUUCCAGGUGGCGCCGCGCCGCACAUGGCGGUACUCGGACAUGUAGCGGGAGA ..(((.....((......)).(((..(((((((..(((((......))))).)).(((((((.(((.......))).))))))))))))(((((.((.....))..)))))))))))... ( -53.70) >consensus UGCCCUUGUAGCAAGCGGGCAGCUCGGCGGCGGUGGCCUGGCGGCCCAGGCACCAGUGGCGUGAUCCCUUCCAGGUGGCGCCCCGCCGCACAUGCCGAUACUCGGACAUGUAGCGGGCAA ((((((((...))))..))))((((((((((((..((((((....))))))......(((((.(((.......))).))))))))))))((((((((.....))).)))))..))))).. (-55.26 = -55.58 + 0.32)


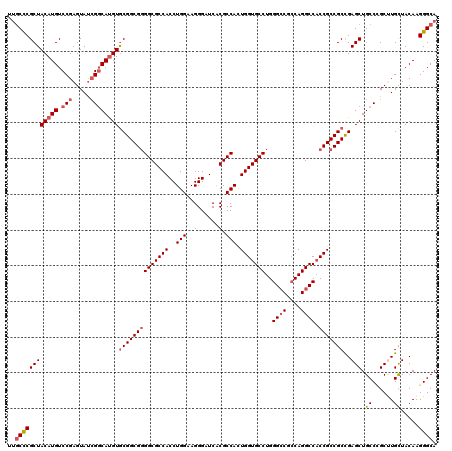
| Location | 15,763,435 – 15,763,555 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.42 |
| Mean single sequence MFE | -57.96 |
| Consensus MFE | -50.40 |
| Energy contribution | -51.48 |
| Covariance contribution | 1.08 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.30 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.707119 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 15763435 120 - 27905053 UUGCUCGCUACAUGUCCGAGUAUCGGCAUGUGCGGCGGGGCGCCACCUGGAAGGGAUCACGCCAUUGGUGCCUGGGCCGCCAGGCCACCGCCGCCGAGCUGCCCGCUUGCUACAAGGGCA ..(((((..(((((.(((.....))))))))((((((((((((((..(((..(......).))).)))))))..((((....)))).)))))))))))).((((...........)))). ( -63.30) >DroSec_CAF1 20634 120 - 1 UUGCCCGCUACAUGUCCGAGUAUCGGCAUGUACGGCGGGGCGCCACCUGGAAGGGAUCACGCCACUGGUGCCUGGGCCGCCAGGCCACCGCCGCCGAGCUGCCCGCUUGCUACAAGGGCA .(((((((((((((.(((.....))))))))).((((((((((((..(((..(......).))).)))))))..((((....)))).)))))))(((((.....)))))......))))) ( -59.70) >DroSim_CAF1 20613 120 - 1 UUGCCCGCUACAUGUCCGAGUAUAGGCAUGUGCGGCGGGGCGCCACCUGGAAGGGAUCACGCCAUUGGUGCCUGGGCCGCCAGGCCACCGCCGCCGAGCUGCCUGCUUGUUACAAGGGCA .(((((...(((((((........)))))))((((((((((((((..(((..(......).))).)))))))..((((....)))).)))))))(((((.....)))))......))))) ( -60.20) >DroEre_CAF1 22819 120 - 1 UUGCCCGCUACAUGUCCGAGUAUCGGCACGUGCGGCGGGGCGCCACCUGGAAGGGAUCACGCCACUGGUGCCUGGGCCGCCAGGCCACCGCCGCCGAGCUGCCCGCCUGCUACAAGGGCA .((((((((.((((((((.....))).)))))((((((((((...(((....)))....))))...(((((((((....))))).)))).))))))))).((......)).....))))) ( -58.10) >DroPer_CAF1 21106 120 - 1 UCUCCCGCUACAUGUCCGAGUACCGCCAUGUGCGGCGCGGCGCCACCUGGAAGGGAUCGCGCCACUGGUGCCUGGGGCGACAGGCCUCCGCCGCUGAGCUGCCAGCCUGCUACAAGGGCA ......((((((((..((.....)).)))))(((((..(((((..(((....)))...)))))...((.(((((......)))))..)))))))..))).....((((.......)))). ( -48.50) >consensus UUGCCCGCUACAUGUCCGAGUAUCGGCAUGUGCGGCGGGGCGCCACCUGGAAGGGAUCACGCCACUGGUGCCUGGGCCGCCAGGCCACCGCCGCCGAGCUGCCCGCUUGCUACAAGGGCA ..((((((((((((.(((.....))))))))((((((((((((((..(((..(......).))).)))))))..((((....)))).)))))))..))).((......)).....)))). (-50.40 = -51.48 + 1.08)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:09:29 2006