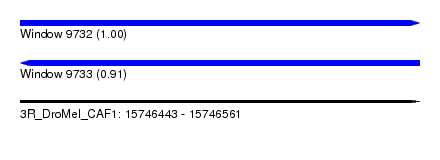
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 15,746,443 – 15,746,561 |
| Length | 118 |
| Max. P | 0.995898 |

| Location | 15,746,443 – 15,746,561 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 87.46 |
| Mean single sequence MFE | -55.62 |
| Consensus MFE | -47.82 |
| Energy contribution | -48.70 |
| Covariance contribution | 0.88 |
| Combinations/Pair | 1.10 |
| Mean z-score | -6.37 |
| Structure conservation index | 0.86 |
| SVM decision value | 2.63 |
| SVM RNA-class probability | 0.995898 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 15746443 118 + 27905053 UGUAUAAAUCGUUCCCUGACUGGUGGCUUCUCCGACACCCGCUAACACAAAGUAUACAC--CAUAGGCAGUGUAUAAUCUGUUCUAGCGGGAGUCGGUGAAGGCACCUGUCAGGGAAACG .........(((((((((((.((((.((((.(((((.(((((((..(((...(((((((--........)))))))...)))..))))))).))))).)))).)))).))))))).)))) ( -65.30) >DroSec_CAF1 3632 118 + 1 UGUAGAAAUCGUUCCCUGACUGGUGGCUUCUCCGAGACCCGCUAACACAAAGUAUACAC--CAUAGGCUGUGUAUAAUCUGUUCUAGCGGGAGUCGGUGAAGGCACCUGUCAGGGAAACG .........(((((((((((.((((.((((.((((..(((((((..(((...(((((((--........)))))))...)))..)))))))..)))).)))).)))).))))))).)))) ( -61.90) >DroSim_CAF1 3635 120 + 1 UGUAGAAAUCGUUCCCUGACUGGUGGCUUCUCCGACAACCGCUAACACAAAGUAUACACCCCAUAGGAUGUGCAUAAUCUGUUCUAGCGGGAGUCGGUGAAGGCACCUGUCAGGGAAACG .........(((((((((((.((((.((((.(((((..((((((..(((...(((.(((((....))..))).)))...)))..))))))..))))).)))).)))).))))))).)))) ( -58.60) >DroEre_CAF1 4724 118 + 1 UGUACAAAUCGUUCCCUGACUGGUGACUUCUCCGACACCCACAAAAACAAAGUAUACAC--CAUGGGCUGUGCAUAAUCCGUUCUUGUGGGAGUCGUGGAAGGCACCCGCCUAGGAAACG .........((((.((((.(.((((.((((..((((.(((((((.(((....(((.(((--........))).)))....))).))))))).))))..)))).)))).)..)))).)))) ( -43.00) >DroYak_CAF1 3623 118 + 1 UGUACAAAUCGUUCCCUGGCUGAAGUCUUCUCCGACACCCACAAAAACAAAGUAUCCAC--CCUAGGCUGUGCAUAAUCCGUUUUUGUGGGAGUGGGUGAAGGCAUCCGCCUAGGAAACG .........((((.((((((.((.((((((.((.((.(((((((((((....(((.(((--........))).)))....))))))))))).)).)).)))))).)).))).))).)))) ( -49.30) >consensus UGUACAAAUCGUUCCCUGACUGGUGGCUUCUCCGACACCCGCUAACACAAAGUAUACAC__CAUAGGCUGUGCAUAAUCUGUUCUAGCGGGAGUCGGUGAAGGCACCUGUCAGGGAAACG .........(((((((((((.((((.((((.(((((.(((((((..(((...(((.(((..........))).)))...)))..))))))).))))).)))).)))).))))))).)))) (-47.82 = -48.70 + 0.88)

| Location | 15,746,443 – 15,746,561 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 87.46 |
| Mean single sequence MFE | -63.24 |
| Consensus MFE | -59.32 |
| Energy contribution | -58.44 |
| Covariance contribution | -0.88 |
| Combinations/Pair | 1.20 |
| Mean z-score | -8.24 |
| Structure conservation index | 0.94 |
| SVM decision value | 1.07 |
| SVM RNA-class probability | 0.910195 |
| Prediction | RNA |
| WARNING | Mean z-score out of range. |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 15746443 118 - 27905053 CGUUUCCCUGACAGGUGCCUUCACCGACUCCCGCUAGAACAGAUUAUACACUGCCUAUG--GUGUAUACUUUGUGUUAGCGGGUGUCGGAGAAGCCACCAGUCAGGGAACGAUUUAUACA ((((.(((((((.((((.((((.(((((.((((((((.(((((.((((((((......)--))))))).))))).)))))))).))))).)))).)))).)))))))))))......... ( -69.10) >DroSec_CAF1 3632 118 - 1 CGUUUCCCUGACAGGUGCCUUCACCGACUCCCGCUAGAACAGAUUAUACACAGCCUAUG--GUGUAUACUUUGUGUUAGCGGGUCUCGGAGAAGCCACCAGUCAGGGAACGAUUUCUACA ((((.(((((((.((((.((((.((((..((((((((.(((((.(((((((........--))))))).))))).))))))))..)))).)))).)))).)))))))))))......... ( -64.20) >DroSim_CAF1 3635 120 - 1 CGUUUCCCUGACAGGUGCCUUCACCGACUCCCGCUAGAACAGAUUAUGCACAUCCUAUGGGGUGUAUACUUUGUGUUAGCGGUUGUCGGAGAAGCCACCAGUCAGGGAACGAUUUCUACA ((((.(((((((.((((.((((.(((((..(((((((.(((((.(((((((.(((...)))))))))).))))).)))))))..))))).)))).)))).)))))))))))......... ( -65.40) >DroEre_CAF1 4724 118 - 1 CGUUUCCUAGGCGGGUGCCUUCCACGACUCCCACAAGAACGGAUUAUGCACAGCCCAUG--GUGUAUACUUUGUUUUUGUGGGUGUCGGAGAAGUCACCAGUCAGGGAACGAUUUGUACA ((((((((.(((.((((.((((..((((.((((((((((((((.(((((((........--))))))).)))))))))))))).))))..)))).)))).)))))))))))......... ( -63.70) >DroYak_CAF1 3623 118 - 1 CGUUUCCUAGGCGGAUGCCUUCACCCACUCCCACAAAAACGGAUUAUGCACAGCCUAGG--GUGGAUACUUUGUUUUUGUGGGUGUCGGAGAAGACUUCAGCCAGGGAACGAUUUGUACA ((((((((.(((.((.(.((((.((.((.((((((((((((((.(((.(((........--))).))).)))))))))))))).)).)).)))).).)).)))))))))))......... ( -53.80) >consensus CGUUUCCCUGACAGGUGCCUUCACCGACUCCCGCUAGAACAGAUUAUGCACAGCCUAUG__GUGUAUACUUUGUGUUAGCGGGUGUCGGAGAAGCCACCAGUCAGGGAACGAUUUCUACA ((((.(((((((.((((.((((.(((((.((((((((.(((((.(((((((..........))))))).))))).)))))))).))))).)))).)))).)))))))))))......... (-59.32 = -58.44 + -0.88)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:09:10 2006