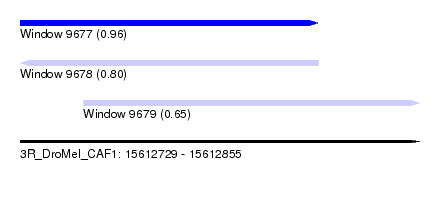
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 15,612,729 – 15,612,855 |
| Length | 126 |
| Max. P | 0.955131 |

| Location | 15,612,729 – 15,612,823 |
|---|---|
| Length | 94 |
| Sequences | 4 |
| Columns | 94 |
| Reading direction | forward |
| Mean pairwise identity | 87.06 |
| Mean single sequence MFE | -29.93 |
| Consensus MFE | -20.66 |
| Energy contribution | -22.10 |
| Covariance contribution | 1.44 |
| Combinations/Pair | 1.10 |
| Mean z-score | -3.35 |
| Structure conservation index | 0.69 |
| SVM decision value | 1.45 |
| SVM RNA-class probability | 0.955131 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 15612729 94 + 27905053 CGAUCGGUGAUCGGUGAUCGGAGAUCGCAGAUCUCAGUUCCAGAAUCGCAAAUCUGCGAUCUAAUCUGCACGCUGAUUAAUUCAUUAGCAACUG .((((.((((((..........)))))).)))).(((((.((((((((((....))))))....))))...((((((......))))))))))) ( -29.80) >DroSim_CAF1 122747 94 + 1 CGAUCGGUGAUCUGAGAUCGCAGAGCUCAGAUCACAGUUCCAGAAUCGCAAAUCUGCGAUCUAAUCUGCACGCUGAUUAAUUCAUUAGCAACUG ......((((((((((.((...)).))))))))))((((.((((((((((....))))))....))))...((((((......)))))))))). ( -31.80) >DroEre_CAF1 118215 94 + 1 CGAUCGGCGAUUGGAGAUUGGAGAUCACAGAUCUCAGAUCCAGAAUCGCAAAUCUGCGAUCUAAUCCGCACGCUGAUUAAUUCAUUAGCAACUG .((((((((..((((((((.((((((...)))))).)))).(((.(((((....))))))))..))))..))))))))................ ( -32.20) >DroYak_CAF1 131222 80 + 1 CGAUCG--------------GAGAUCGCAGAUCUCAGAUCCAGAAUCGCAAAUCUGCGAUCUAAUCUGCACGCUGAUUAAUUCAUUAGCAACUG .((((.--------------((((((...)))))).))))((((((((((....))))))....))))...((((((......))))))..... ( -25.90) >consensus CGAUCGGUGAUCGGAGAUCGGAGAUCGCAGAUCUCAGAUCCAGAAUCGCAAAUCUGCGAUCUAAUCUGCACGCUGAUUAAUUCAUUAGCAACUG .((((..(((((...)))))..))))((((((...(((((((((........)))).))))).))))))..((((((......))))))..... (-20.66 = -22.10 + 1.44)



| Location | 15,612,729 – 15,612,823 |
|---|---|
| Length | 94 |
| Sequences | 4 |
| Columns | 94 |
| Reading direction | reverse |
| Mean pairwise identity | 87.06 |
| Mean single sequence MFE | -28.20 |
| Consensus MFE | -19.19 |
| Energy contribution | -19.88 |
| Covariance contribution | 0.69 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.21 |
| Structure conservation index | 0.68 |
| SVM decision value | 0.61 |
| SVM RNA-class probability | 0.799368 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 15612729 94 - 27905053 CAGUUGCUAAUGAAUUAAUCAGCGUGCAGAUUAGAUCGCAGAUUUGCGAUUCUGGAACUGAGAUCUGCGAUCUCCGAUCACCGAUCACCGAUCG ....(((...(((.....)))....)))(((((((((((((((((.((.((....)).)))))))))))))))..))))..(((((...))))) ( -27.60) >DroSim_CAF1 122747 94 - 1 CAGUUGCUAAUGAAUUAAUCAGCGUGCAGAUUAGAUCGCAGAUUUGCGAUUCUGGAACUGUGAUCUGAGCUCUGCGAUCUCAGAUCACCGAUCG (((((((...(((.....)))....))))....((((((......)))))))))((.(.((((((((((.((...)).)))))))))).).)). ( -29.80) >DroEre_CAF1 118215 94 - 1 CAGUUGCUAAUGAAUUAAUCAGCGUGCGGAUUAGAUCGCAGAUUUGCGAUUCUGGAUCUGAGAUCUGUGAUCUCCAAUCUCCAAUCGCCGAUCG .....(((.((......)).))).(((((......)))))((((.((((((..((((..((((((...))))))..))))..)))))).)))). ( -28.90) >DroYak_CAF1 131222 80 - 1 CAGUUGCUAAUGAAUUAAUCAGCGUGCAGAUUAGAUCGCAGAUUUGCGAUUCUGGAUCUGAGAUCUGCGAUCUC--------------CGAUCG ....(((...(((.....)))....)))(((((((((((((((((..((((...))))..))))))))))))).--------------.)))). ( -26.50) >consensus CAGUUGCUAAUGAAUUAAUCAGCGUGCAGAUUAGAUCGCAGAUUUGCGAUUCUGGAACUGAGAUCUGCGAUCUCCGAUCUCCGAUCACCGAUCG .....(((.((......)).)))..((((((((((((.((((........)))))))))..)))))))((((.................)))). (-19.19 = -19.88 + 0.69)



| Location | 15,612,749 – 15,612,855 |
|---|---|
| Length | 106 |
| Sequences | 6 |
| Columns | 107 |
| Reading direction | forward |
| Mean pairwise identity | 82.84 |
| Mean single sequence MFE | -27.47 |
| Consensus MFE | -16.75 |
| Energy contribution | -16.53 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.45 |
| Structure conservation index | 0.61 |
| SVM decision value | 0.24 |
| SVM RNA-class probability | 0.649492 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 15612749 106 + 27905053 GAGAUCGCAGAUCUCAGUUCCAGAAUCGCAAAUCUGCGAUCUAAUCUGCACGCUGAUUAAUUCAUUAGCAACUGUCGACCAGUUUGUCAGUUA-CAUGGCAAUAACU .(((((((((((..(.(((....))).)...)))))))))))....(((..((((((......))))))(((((.(((.....))).))))).-....)))...... ( -30.30) >DroSim_CAF1 122767 106 + 1 CAGAGCUCAGAUCACAGUUCCAGAAUCGCAAAUCUGCGAUCUAAUCUGCACGCUGAUUAAUUCAUUAGCAACUGUCGACCAGUUUGUCAGUUA-CAUGGCAAUAACU ..(((((..(.((((((((.((((((((((....))))))....))))...((((((......)))))))))))).)).))))))((((....-..))))....... ( -26.20) >DroEre_CAF1 118235 106 + 1 GAGAUCACAGAUCUCAGAUCCAGAAUCGCAAAUCUGCGAUCUAAUCCGCACGCUGAUUAAUUCAUUAGCAACUGUCGACCAGUUUGUCAGUUA-CAUGGCAAUAACU ((((((...))))))(((((((((........)))).))))).....((..((((((......))))))(((((.(((.....))).))))).-....))....... ( -29.60) >DroWil_CAF1 127386 94 + 1 GAAAUCAC-----U-CGAUCAUGGAUUGUUGAUCUGCGAUCUU----UUGCGCUGAUUAAUUCAUUAGCAACUGUCGACCAGUUUGUCAGUUAACAAGUCGA--AC- ........-----(-((((...(((((((......))))))).----(((.((((((......))))))(((((.(((.....))).)))))..))))))))--..- ( -21.50) >DroYak_CAF1 131228 106 + 1 GAGAUCGCAGAUCUCAGAUCCAGAAUCGCAAAUCUGCGAUCUAAUCUGCACGCUGAUUAAUUCAUUAGCAACUGUCGACCAGUUUGUCAGUUA-CAUGGCAAUAACU .(((((((((((....(((.....)))....)))))))))))....(((..((((((......))))))(((((.(((.....))).))))).-....)))...... ( -32.70) >DroAna_CAF1 134829 94 + 1 ------------CACAGAUCUGCGAUCGCGAAUCUGCGAUCUAAUCUCCGUGCUGAUUAAUUCAUUAGCAACUGUCGACCAGUUUGUCAGUUA-CAUGGCAAUGACU ------------.((((......(((((((....))))))).........(((((((......))))))).)))).....(((((((((....-..)))))..)))) ( -24.50) >consensus GAGAUCACAGAUCUCAGAUCCAGAAUCGCAAAUCUGCGAUCUAAUCUGCACGCUGAUUAAUUCAUUAGCAACUGUCGACCAGUUUGUCAGUUA_CAUGGCAAUAACU ........................((((((....))))))...........((((((......))))))(((((.(((.....))).)))))............... (-16.75 = -16.53 + -0.22)

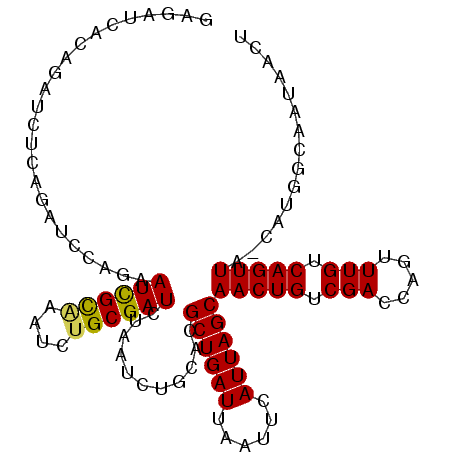

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:08:04 2006