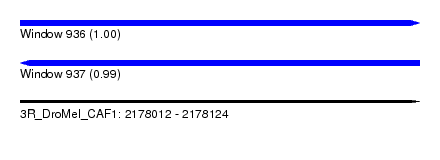
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 2,178,012 – 2,178,124 |
| Length | 112 |
| Max. P | 0.999660 |

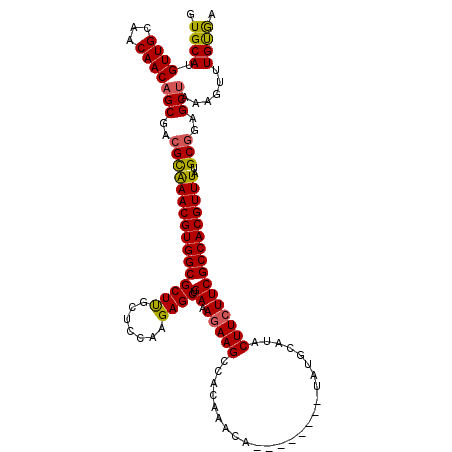
| Location | 2,178,012 – 2,178,124 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 90.97 |
| Mean single sequence MFE | -42.16 |
| Consensus MFE | -35.36 |
| Energy contribution | -35.68 |
| Covariance contribution | 0.32 |
| Combinations/Pair | 1.11 |
| Mean z-score | -3.52 |
| Structure conservation index | 0.84 |
| SVM decision value | 3.85 |
| SVM RNA-class probability | 0.999660 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 2178012 112 + 27905053 UCACAAACUUUGGCUCCGCGUAAACGUGGCGAAGAAGUAUGCAUA--------UGUUUGUGGCUUCUUUCAGCUCUUGGAGCGAGCGCCACGUUUGCGUCGCUGUUGUUGCAACAUGCAC .....(((..((((...(((((((((((((((((((((.((((..--------....))))))))))))..((((.......))))))))))))))))).))))..)))((.....)).. ( -43.90) >DroSec_CAF1 3113 112 + 1 UCACAAACUUUAGCUCCGCAUAAACGUGGCGAAGAAGUAUGCAUA--------UGUUUGUGGCUUCUUUCAGCUCUUGGAGCAAGCGCCACGUUUGCGUCGCUGUUGUUGCAACAUGCAC .....(((..((((..((((..((((((((((((((((.((((..--------....)))))))))))...((((...))))...)))))))))))))..))))..)))((.....)).. ( -42.50) >DroSim_CAF1 3218 112 + 1 UCACAAACUUUAGCUCCGCAUAAACGUGGCGAAGAAGUAUGCAUA--------UGUUUGUGGCUUCUUUCAGCUCUUGGAGCAAGCGCCACGUUUGCGUCGCUGUUGUUGCAACAUGCAC .....(((..((((..((((..((((((((((((((((.((((..--------....)))))))))))...((((...))))...)))))))))))))..))))..)))((.....)).. ( -42.50) >DroEre_CAF1 4977 120 + 1 UUGCAAGCUUUAGCUCCGCAUAAACGUGGCGAAGAAGUAUACAUACAUAUGUUUGUUUGUGGCUUCUAUCAGCUCUUGGAGCAAGCGCCACGUUUGCUUCGCUGUUGUUGCAACAUGCAC ((((((.(..((((...(((..(((((((((.((((((.((((.(((......))).))))))))))....((((...))))...))))))))))))...))))..)))))))....... ( -44.60) >DroYak_CAF1 3482 110 + 1 UCGCAAAC--UAGCUCCGCAUAAACGUGGCGAACAAGUAUACAUA--------UGCUUGUGGCUUCUUUCAGCUCUUGGAGCAAGCGCCACGUUCACGUCGCUGUUGUUACAACGUGCAC ..(((...--.(((..((....(((((((((.((((((((....)--------))))))).(((((...........)))))...)))))))))..))..)))((((...)))).))).. ( -37.30) >consensus UCACAAACUUUAGCUCCGCAUAAACGUGGCGAAGAAGUAUGCAUA________UGUUUGUGGCUUCUUUCAGCUCUUGGAGCAAGCGCCACGUUUGCGUCGCUGUUGUUGCAACAUGCAC .....(((..((((..((((..((((((((((((((((.((((..............)))))))))))...((((...))))...)))))))))))))..))))..)))........... (-35.36 = -35.68 + 0.32)

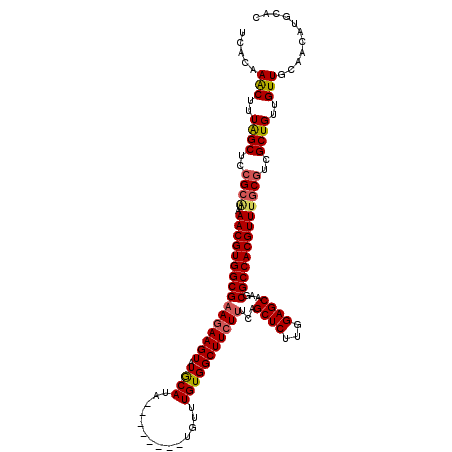
| Location | 2,178,012 – 2,178,124 |
|---|---|
| Length | 112 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 90.97 |
| Mean single sequence MFE | -35.74 |
| Consensus MFE | -30.99 |
| Energy contribution | -30.71 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.15 |
| Mean z-score | -2.08 |
| Structure conservation index | 0.87 |
| SVM decision value | 2.35 |
| SVM RNA-class probability | 0.992793 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 2178012 112 - 27905053 GUGCAUGUUGCAACAACAGCGACGCAAACGUGGCGCUCGCUCCAAGAGCUGAAAGAAGCCACAAACA--------UAUGCAUACUUCUUCGCCACGUUUACGCGGAGCCAAAGUUUGUGA ..((((((((...)))))((..((((((((((((((((.......)))).(((.((((...((....--------..))....))))))))))))))))..)))..)).......))).. ( -35.00) >DroSec_CAF1 3113 112 - 1 GUGCAUGUUGCAACAACAGCGACGCAAACGUGGCGCUUGCUCCAAGAGCUGAAAGAAGCCACAAACA--------UAUGCAUACUUCUUCGCCACGUUUAUGCGGAGCUAAAGUUUGUGA .(((.....))).....(((..(((((((((((((...((((...))))...((((((...((....--------..))....)))))))))))))))..))))..)))........... ( -34.80) >DroSim_CAF1 3218 112 - 1 GUGCAUGUUGCAACAACAGCGACGCAAACGUGGCGCUUGCUCCAAGAGCUGAAAGAAGCCACAAACA--------UAUGCAUACUUCUUCGCCACGUUUAUGCGGAGCUAAAGUUUGUGA .(((.....))).....(((..(((((((((((((...((((...))))...((((((...((....--------..))....)))))))))))))))..))))..)))........... ( -34.80) >DroEre_CAF1 4977 120 - 1 GUGCAUGUUGCAACAACAGCGAAGCAAACGUGGCGCUUGCUCCAAGAGCUGAUAGAAGCCACAAACAAACAUAUGUAUGUAUACUUCUUCGCCACGUUUAUGCGGAGCUAAAGCUUGCAA .(((.(((((...))))))))....(((((((((....((((...)))).((.(((((..(((.(((......))).)))...)))))))))))))))).((((.(((....))))))). ( -38.00) >DroYak_CAF1 3482 110 - 1 GUGCACGUUGUAACAACAGCGACGUGAACGUGGCGCUUGCUCCAAGAGCUGAAAGAAGCCACAAGCA--------UAUGUAUACUUGUUCGCCACGUUUAUGCGGAGCUA--GUUUGCGA .((((((((((....)))))).((((((((((((((((((((...))))......)))).(((((.(--------(....)).)))))..))))))))))))........--...)))). ( -36.10) >consensus GUGCAUGUUGCAACAACAGCGACGCAAACGUGGCGCUUGCUCCAAGAGCUGAAAGAAGCCACAAACA________UAUGCAUACUUCUUCGCCACGUUUAUGCGGAGCUAAAGUUUGUGA .((((.((((...))))(((..((((((((((((((((.......)))).((.(((((.........................))))))))))))))))..)))..)))......)))). (-30.99 = -30.71 + -0.28)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:46:49 2006