
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 15,201,165 – 15,201,316 |
| Length | 151 |
| Max. P | 0.997916 |

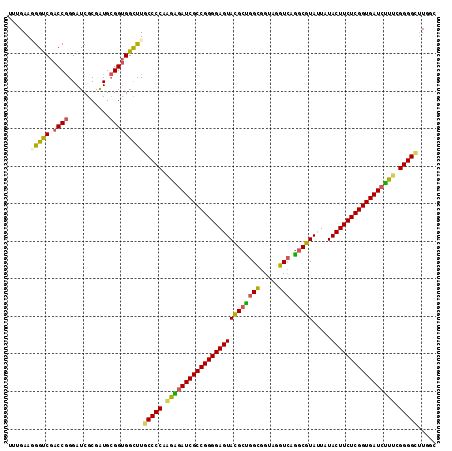
| Location | 15,201,165 – 15,201,279 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 86.67 |
| Mean single sequence MFE | -54.58 |
| Consensus MFE | -50.48 |
| Energy contribution | -49.77 |
| Covariance contribution | -0.71 |
| Combinations/Pair | 1.33 |
| Mean z-score | -3.21 |
| Structure conservation index | 0.92 |
| SVM decision value | 2.96 |
| SVM RNA-class probability | 0.997916 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 15201165 114 + 27905053 UUUGAAGGGUCGACCGGGAUCGCGAUGCGGUGGCUUGCCCCAAGAGAUCGCCGGGGAGUACGCUGGCGGUAGGUCAGGCGUAUUAUACUUCUCGGUGAUCUUUCGGGGCUUUGC ......(((((.((((.(.......).)))))))))(((((.(((((((((((((((((((((((((.....)))).)))).....))))))))))))))))).)))))..... ( -55.20) >DroSec_CAF1 124478 114 + 1 UUUGAAUGGUCGACCGGGAUCGCGAUGCGGUGGCUUGCCCCAAGAGAUCGCCGGGGAGUACGCUGGUGGUAGGUCAGGCGUAUUAUACUUCUCGGUGAUCUUUCGGGGCUUUGC .......((((.((((.(.......).)))))))).(((((.((((((((((((((((((((((.(........).))))).....))))))))))))))))).)))))..... ( -50.50) >DroSim_CAF1 127159 114 + 1 UUUGAAUGGUCGACCGGGAUCGCGAUGCGGUGGCUUGCCCCAAGAGAUCGCCGGGGAGUACGCUGGCGGUAGGUCAGGCGUAUUAUACUUCUCGGUGAUCUUUCGGGGCUUUGC .......((((.((((.(.......).)))))))).(((((.(((((((((((((((((((((((((.....)))).)))).....))))))))))))))))).)))))..... ( -54.00) >DroEre_CAF1 122656 114 + 1 CUGGAAGGGUCGACCGGGAUCGCGAUGCGGUGGCCUGCCCCAAAAGAUCGCCGGGGAGUACGCCGGCGGUCGGUCCGGCGUAUUGUACUUCUCGGUGAUCUUUCGGGGUUUGGC ((((.........)))).(((((...))))).(((.(((((.((((((((((((((((((((.((.(((.....))).))...)))))))))))))))))))).)))))..))) ( -60.10) >DroYak_CAF1 130386 114 + 1 AUUGAAGGGUCGACCGGGAUCGCGAUGCGGUGGCUUGCCCCAAGGGAUCGCCGGGGAGUACGCUGGCGGUUGGUCUGGCGUAUUAUACUUCUCGGUGAUCUUCCGGGGCUUGGC ......(((((.((((.(.......).)))))))))(((((..((((((((((((((((((((..((.....).)..)))).....))))))))))))))))..)))))..... ( -52.40) >DroAna_CAF1 120324 114 + 1 CUUGAAAGGCCUUCCAGGAUCGCGGUGAGGCGGUCUACCCCACACAAUCGCCGGGGAGUAGGCUGGCGGAUCGUAGAUCUUAGUAUACUUCUCGGUGAUAGUGCGGGGCAUGGA ............(((((((((((......))))))).((((.(((.((((((((((((((.((((..(((((...))))))))).)))))))))))))).))).))))..)))) ( -55.30) >consensus UUUGAAGGGUCGACCGGGAUCGCGAUGCGGUGGCUUGCCCCAAGAGAUCGCCGGGGAGUACGCUGGCGGUAGGUCAGGCGUAUUAUACUUCUCGGUGAUCUUUCGGGGCUUGGC ......(((((.((((...........)))))))))(((((.(((((((((((((((((((((((((.....))).))))).....))))))))))))))))).)))))..... (-50.48 = -49.77 + -0.71)


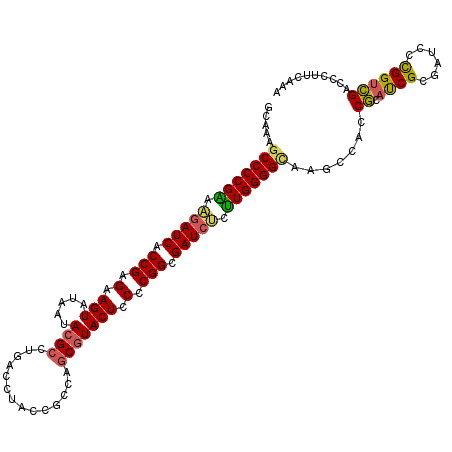
| Location | 15,201,165 – 15,201,279 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 86.67 |
| Mean single sequence MFE | -38.22 |
| Consensus MFE | -29.65 |
| Energy contribution | -29.37 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.24 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.78 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.513047 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 15201165 114 - 27905053 GCAAAGCCCCGAAAGAUCACCGAGAAGUAUAAUACGCCUGACCUACCGCCAGCGUACUCCCCGGCGAUCUCUUGGGGCAAGCCACCGCAUCGCGAUCCCGGUCGACCCUUCAAA ((...(((((((.(((((.(((.(.(((.....((((.((.........))))))))).).))).))))).)))))))..((....))...))(((....)))........... ( -36.70) >DroSec_CAF1 124478 114 - 1 GCAAAGCCCCGAAAGAUCACCGAGAAGUAUAAUACGCCUGACCUACCACCAGCGUACUCCCCGGCGAUCUCUUGGGGCAAGCCACCGCAUCGCGAUCCCGGUCGACCAUUCAAA ((...(((((((.(((((.(((.(.(((.....((((.((.........))))))))).).))).))))).)))))))..((....))...))(((....)))........... ( -36.70) >DroSim_CAF1 127159 114 - 1 GCAAAGCCCCGAAAGAUCACCGAGAAGUAUAAUACGCCUGACCUACCGCCAGCGUACUCCCCGGCGAUCUCUUGGGGCAAGCCACCGCAUCGCGAUCCCGGUCGACCAUUCAAA ((...(((((((.(((((.(((.(.(((.....((((.((.........))))))))).).))).))))).)))))))..((....))...))(((....)))........... ( -36.70) >DroEre_CAF1 122656 114 - 1 GCCAAACCCCGAAAGAUCACCGAGAAGUACAAUACGCCGGACCGACCGCCGGCGUACUCCCCGGCGAUCUUUUGGGGCAGGCCACCGCAUCGCGAUCCCGGUCGACCCUUCCAG (((...((((((((((((.(((.(.(((.....(((((((........)))))))))).).))).))))))))))))..)))..........((((....)))).......... ( -47.90) >DroYak_CAF1 130386 114 - 1 GCCAAGCCCCGGAAGAUCACCGAGAAGUAUAAUACGCCAGACCAACCGCCAGCGUACUCCCCGGCGAUCCCUUGGGGCAAGCCACCGCAUCGCGAUCCCGGUCGACCCUUCAAU (((..(((((((..((((.(((.(.(((.....((((..(........)..))))))).).))).))))..))))))).......(((...))).....)))............ ( -32.10) >DroAna_CAF1 120324 114 - 1 UCCAUGCCCCGCACUAUCACCGAGAAGUAUACUAAGAUCUACGAUCCGCCAGCCUACUCCCCGGCGAUUGUGUGGGGUAGACCGCCUCACCGCGAUCCUGGAAGGCCUUUCAAG ((((((((((((((.(((.(((.(.((((..((..((((...))))....))..)))).).))).))).))))))))))((.(((......))).)).))))............ ( -39.20) >consensus GCAAAGCCCCGAAAGAUCACCGAGAAGUAUAAUACGCCUGACCUACCGCCAGCGUACUCCCCGGCGAUCUCUUGGGGCAAGCCACCGCAUCGCGAUCCCGGUCGACCCUUCAAA .....(((((((.(((((.(((.(.(((.....((((..............))))))).).))).))))).))))))).......((.((((......)))))).......... (-29.65 = -29.37 + -0.27)

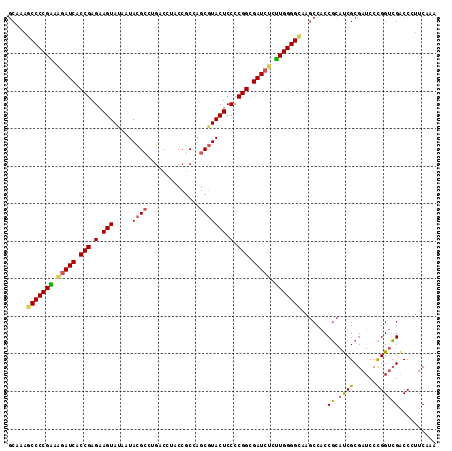
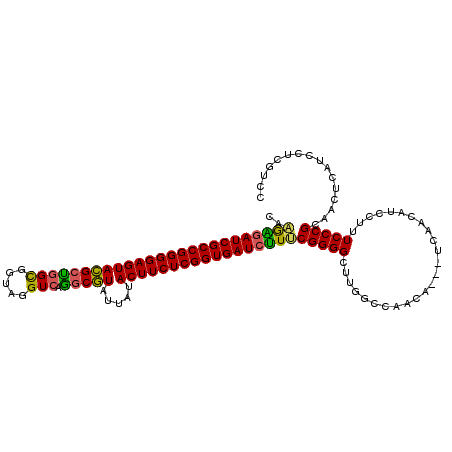
| Location | 15,201,205 – 15,201,316 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 83.81 |
| Mean single sequence MFE | -39.99 |
| Consensus MFE | -34.87 |
| Energy contribution | -34.01 |
| Covariance contribution | -0.86 |
| Combinations/Pair | 1.27 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.87 |
| SVM decision value | 0.58 |
| SVM RNA-class probability | 0.788355 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 15201205 111 + 27905053 CAAGAGAUCGCCGGGGAGUACGCUGGCGGUAGGUCAGGCGUAUUAUACUUCUCGGUGAUCUUUCGGGGCUUUGCCAACA---UCAACAUCCUUUCCCGCAACUCAUCCUCGUCC ..(((((((((((((((((((((((((.....)))).)))).....)))))))))))))))))(((((..((((.....---...............)))).....)))))... ( -39.15) >DroSec_CAF1 124518 111 + 1 CAAGAGAUCGCCGGGGAGUACGCUGGUGGUAGGUCAGGCGUAUUAUACUUCUCGGUGAUCUUUCGGGGCUUUGCCAACA---UCAACAUUCUUUCCCGCAACUCAUCCUCGUCC ..((((((((((((((((((((((.(........).))))).....)))))))))))))))))(((((..((((.....---...............)))).....)))))... ( -35.65) >DroSim_CAF1 127199 111 + 1 CAAGAGAUCGCCGGGGAGUACGCUGGCGGUAGGUCAGGCGUAUUAUACUUCUCGGUGAUCUUUCGGGGCUUUGCCAACA---UCAACAUUCUUUCCCGCAACUCAUCCUCGUCC ..(((((((((((((((((((((((((.....)))).)))).....)))))))))))))))))(((((..((((.....---...............)))).....)))))... ( -39.15) >DroEre_CAF1 122696 111 + 1 CAAAAGAUCGCCGGGGAGUACGCCGGCGGUCGGUCCGGCGUAUUGUACUUCUCGGUGAUCUUUCGGGGUUUGGCCAGCA---GCAACAUCCUUUCCCGCAACUCAUCCUCCUCC ..((((((((((((((((((((.((.(((.....))).))...)))))))))))))))))))).((((..(((...((.---...............))...)))..))))... ( -44.99) >DroYak_CAF1 130426 111 + 1 CAAGGGAUCGCCGGGGAGUACGCUGGCGGUUGGUCUGGCGUAUUAUACUUCUCGGUGAUCUUCCGGGGCUUGGCCAACA---UUAAGAUCCUUUCCCGCAACUCUUCCUCGUCC .(((((((((((((.(....).))))).(((((((.((((((...)))..(((((.......)))))))).))))))).---....)))))))).................... ( -40.20) >DroAna_CAF1 120364 111 + 1 CACACAAUCGCCGGGGAGUAGGCUGGCGGAUCGUAGAUCUUAGUAUACUUCUCGGUGAUAGUGCGGGGCAUGGAAAGCGCUUCCAACUUCUCUUCCCG---UUCAUCCGGAUCU ..(((.((((((((((((((.((((..(((((...))))))))).)))))))))))))).))).((((..(((((.....)))))...))))...(((---......))).... ( -40.80) >consensus CAAGAGAUCGCCGGGGAGUACGCUGGCGGUAGGUCAGGCGUAUUAUACUUCUCGGUGAUCUUUCGGGGCUUGGCCAACA___UCAACAUCCUUUCCCGCAACUCAUCCUCGUCC ..(((((((((((((((((((((((((.....))).))))).....)))))))))))))))))(((((.........................)))))................ (-34.87 = -34.01 + -0.86)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:05:28 2006