| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
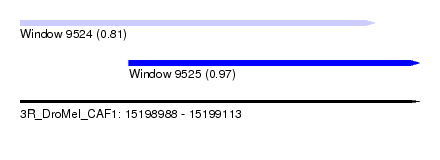
| Location | 15,198,988 – 15,199,113 |
| Length | 125 |
| Max. P | 0.970450 |

| Location | 15,198,988 – 15,199,099 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 79.74 |
| Mean single sequence MFE | -23.33 |
| Consensus MFE | -14.96 |
| Energy contribution | -14.68 |
| Covariance contribution | -0.28 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.86 |
| Structure conservation index | 0.64 |
| SVM decision value | 0.66 |
| SVM RNA-class probability | 0.814649 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
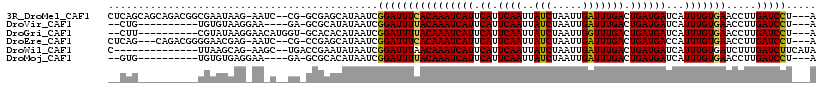
>3R_DroMel_CAF1 15198988 111 + 27905053 CUCAGCAGCAGACGGCGAAUAAG-AAUC--CG-GCGAGCAUAAUCGGAUUUCACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGAUCAUUUGUGAACCUUGAUCCU---A .......((.....))((.((((-((((--((-(.........)))))))(((((((((((.((.((((..(((.....))))))).)))))))...))))))..)))).))..---. ( -22.20) >DroVir_CAF1 141870 98 + 1 --CUG----------UGUGUAAGGAA----GA-GCGCAUAUAAUCGGAUUUUACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGAUCAUUUGUGAACCUUGAUCCU---A --.((----------(((((......----..-))))))).....((((((((((((((((.((.((((..(((.....))))))).)))))))...)))))).....))))).---. ( -21.10) >DroGri_CAF1 165306 102 + 1 --CUU----------CGUAUAAGGAACAUGGU-GCACACAUAAUCGGAUUUUACAAAUCAUUCAUUCAAUUAUCUAAUUGGUUUGACUGAUGAUCAUUUGUGAACCUUGAUCCU---A --...----------......((((.((.(((-.((((.((.((((.......)..((((.(((.((((((....))))))..))).))))))).)).)))).))).)).))))---. ( -23.40) >DroEre_CAF1 120580 108 + 1 CUCAG---CAGACGGGGAACGAG-AAUC--CG-CCGAGCAUAAUCGGAUUUCACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGACCAUUUGUGAACCUUGAUCCU---A .....---......((((.((((-....--..-((((......))))..((((((((((((.((.((((..(((.....))))))).))))))...)))))))).)))).))))---. ( -27.60) >DroWil_CAF1 146930 101 + 1 C--------------UUAAGCAG-AAGC--UGACCGAAUAUAAUCGGAUUUAACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGAUCAUUUGUGAUCUUUGAUCUUCAUA .--------------.......(-(((.--...((((......))))......(((((((.(((.((((((....))))))..))).))))(((((....))))).)))..))))... ( -21.00) >DroMoj_CAF1 142403 98 + 1 --GUG----------UGUGUGAGGAA----GA-GCGCACAUAAUCGGAUUUUACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGAUCAUUUGUGAACCUUGAUCCU---A --(((----------(((((......----..-))))))))....((((((((((((((((.((.((((..(((.....))))))).)))))))...)))))).....))))).---. ( -24.70) >consensus __CUG__________UGAAUAAG_AA_C__GG_GCGAACAUAAUCGGAUUUUACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGAUCAUUUGUGAACCUUGAUCCU___A .............................................((((((((((((((((.((.((((..(((.....))))))).))))))...))))))).....)))))..... (-14.96 = -14.68 + -0.28)

| Location | 15,199,022 – 15,199,113 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.48 |
| Mean single sequence MFE | -22.00 |
| Consensus MFE | -14.92 |
| Energy contribution | -14.78 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.08 |
| Mean z-score | -2.05 |
| Structure conservation index | 0.68 |
| SVM decision value | 1.66 |
| SVM RNA-class probability | 0.970450 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 15199022 91 + 27905053 CAUAAUCGGAUUUCACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGAUCAUUUGUGAACCUUGAUCCU---AGC---------------------U-CCCUGGGCC----CG ....(((((..((((((((((((.((.((((..(((.....))))))).)))))))...)))))))..)))))(((---((.---------------------.-..)))))..----.. ( -20.70) >DroPse_CAF1 137156 117 + 1 CAUAAUCGGAUUUUACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGAUCAUUUGUGAACCUUGAUCCU---AGAGGAUCCACAGUCUGCGCAAUCUGCCUCGGGUCUCUCUG .......((((((((((((((((.((.((((..(((.....))))))).)))))))...)))))).....)))))(---(((((((((...(....)((.....))...))))).))))) ( -27.00) >DroEre_CAF1 120611 95 + 1 CAUAAUCGGAUUUCACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGACCAUUUGUGAACCUUGAUCCU---AGC---------------------U-CCUUGGGCCAAAGCG .......((((((((((((((((.((.((((..(((.....))))))).))))))...))))))).....))))).---.((---------------------(-.....)))....... ( -19.70) >DroWil_CAF1 146951 107 + 1 UAUAAUCGGAUUUAACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGAUCAUUUGUGAUCUUUGAUCUUCAUAGAGGUCCCA--------GCCAACU-CCUCGUUCU-U---U ....((((((........((((.(((.((((((....))))))..))).))))(((((....))))))))))).......((((.....--------.......-)))).....-.---. ( -19.40) >DroMoj_CAF1 142424 86 + 1 CAUAAUCGGAUUUUACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGAUCAUUUGUGAACCUUGAUCCU---AGC---------------------C-G----AGCC----A- .....((((.........((((.(((.((((((....))))))..))).))))(((((...(....)..)))))..---..)---------------------)-)----)...----.- ( -18.20) >DroPer_CAF1 132554 117 + 1 CAUAAUCGGAUUUUACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGAUCAUUUGUGAACCUUGAUCCU---AGAGGAUCCACAGUCUGCGCAAUCUGCCUCGGGUCUCUCUG .......((((((((((((((((.((.((((..(((.....))))))).)))))))...)))))).....)))))(---(((((((((...(....)((.....))...))))).))))) ( -27.00) >consensus CAUAAUCGGAUUUUACAAAUCAUUCAUUCAAUUAUCUAAUUGAUUUGACUGAUGAUCAUUUGUGAACCUUGAUCCU___AGA_____________________U_CCUCGGGCC____CG .......((((((((((((((((.((.((((..(((.....))))))).))))))...))))))).....)))))............................................. (-14.92 = -14.78 + -0.14)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:05:26 2006