
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 14,984,803 – 14,984,963 |
| Length | 160 |
| Max. P | 0.726184 |

| Location | 14,984,803 – 14,984,923 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.89 |
| Mean single sequence MFE | -41.58 |
| Consensus MFE | -24.42 |
| Energy contribution | -25.32 |
| Covariance contribution | 0.89 |
| Combinations/Pair | 1.33 |
| Mean z-score | -1.65 |
| Structure conservation index | 0.59 |
| SVM decision value | 0.17 |
| SVM RNA-class probability | 0.616207 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14984803 120 + 27905053 UGAGAAUAACAACGAACCAGAGGCGGUUAUCAAUAGCCACUCAGUGCGAGAUAACUUUAACCGAGUGCUGUGCCCCAAACUACGCACCUACGUGUGCCCCAUCUGCGGGGCAUCUGGGGA ................(((((((.(((.....(((((.((((.((..(((....)))..)).)))))))))))))).......((((....))))(((((......))))).)))))... ( -37.40) >DroVir_CAF1 3897 120 + 1 UGAAAACAAUAACGAACCCGAAGCUGUGGUCAACAGCCAUACAGUGCGCGACGCGUACGGACGUGUGUUGUGUCCCAAGCUUCGCACUUAUGUGUGCCCCAUAUGCGGAGCAUCCGGCGA (((..(((....((....))....)))..)))...(((((((((..((((.((....))..))))..)))))).....((((((((..((((.......))))))))))))....))).. ( -37.70) >DroGri_CAF1 3634 120 + 1 UGAAAACAAUAAUGAACCCGAGGCUGUGGUCAACAGCCAUUCCGUGCGCGACACUUUCGGACGUGUAUUGUGUCCCAAAUUGCGCACCUAUGUGUGCCCCAUAUGUGGAGCAUCGGGAGA ................(((((((((((.....)))))).((((((((((((...(((.(((((.......))))).))))))))))).((((((.....)))))).))))..)))))... ( -43.30) >DroWil_CAF1 12004 120 + 1 UGAGAACAACAAUGAACCCGAGGCAGUGAUCUACAGUCAUACUGUGCGCGAUAGUUUUAAUCGUGUGCUAUGCCCCAAAUUGCGCACAUACGUGUGCCCCAUAUGUGGGGCAUCAGGUGA ...............(((.(((((........((((.....))))(((((((.......)))))))))).(((((((....((((((....))))))........))))))))).))).. ( -38.60) >DroMoj_CAF1 3497 120 + 1 UGAAAACAACAAUGAACCGGAAGCUGUGGUCAACAGCCAUUCGGUGCGUGACUCCUACGGACGAGUGCUGUGCCCCAAGCUGCGCACCUAUGUAUGCCCCAUUUGUGGGGCAUCCGGCGA ................(((((.(((((.....)))))(((..(((((((.(((((....)..))))(((........))).))))))).)))..(((((((....))))))))))))... ( -46.40) >DroAna_CAF1 10159 120 + 1 CGAGAACAACAGCGAACCGGAGGCAGUGGUUAACAGCCACACGGUUCGCGACAACUUCAAUCGUGUUCUUUGCCCCAAGCUACGCACUUAUGUGUGCCCCAUUUGUGGAGCAUCUGGGGA .(((((((...(((((((((.(((.((.....)).))).).))))))))((.....)).....)))))))..(((((.(((.(((((....)))))..(((....))))))...))))). ( -46.10) >consensus UGAAAACAACAACGAACCCGAGGCUGUGGUCAACAGCCAUACAGUGCGCGACACCUUCAAACGUGUGCUGUGCCCCAAACUGCGCACCUAUGUGUGCCCCAUAUGUGGAGCAUCCGGCGA ................(((((.((.(((((.....))))).(((((((((...........)))))))))..(((((......((((......))))......)).))))).)))))... (-24.42 = -25.32 + 0.89)

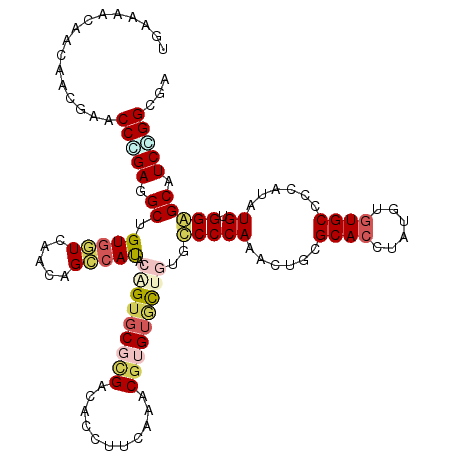
| Location | 14,984,803 – 14,984,923 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 78.89 |
| Mean single sequence MFE | -42.23 |
| Consensus MFE | -24.20 |
| Energy contribution | -23.01 |
| Covariance contribution | -1.19 |
| Combinations/Pair | 1.52 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.57 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.510887 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14984803 120 - 27905053 UCCCCAGAUGCCCCGCAGAUGGGGCACACGUAGGUGCGUAGUUUGGGGCACAGCACUCGGUUAAAGUUAUCUCGCACUGAGUGGCUAUUGAUAACCGCCUCUGGUUCGUUGUUAUUCUCA .((((((((((((((....))))))..((((....)))).))))))))...((((((((((...((....))...))))))).)))..(((((((.(((...)))..)))))))...... ( -41.90) >DroVir_CAF1 3897 120 - 1 UCGCCGGAUGCUCCGCAUAUGGGGCACACAUAAGUGCGAAGCUUGGGACACAACACACGUCCGUACGCGUCGCGCACUGUAUGGCUGUUGACCACAGCUUCGGGUUCGUUAUUGUUUUCA ...((((((((((((....)))))))..((((((((((..((.((((((.........)))).)).))....)))))).))))(((((.....))))))))))................. ( -38.70) >DroGri_CAF1 3634 120 - 1 UCUCCCGAUGCUCCACAUAUGGGGCACACAUAGGUGCGCAAUUUGGGACACAAUACACGUCCGAAAGUGUCGCGCACGGAAUGGCUGUUGACCACAGCCUCGGGUUCAUUAUUGUUUUCA ...((((((((((((....))))))).......((((((..(((.((((.........)))).))).....)))))).....((((((.....)))))))))))................ ( -43.10) >DroWil_CAF1 12004 120 - 1 UCACCUGAUGCCCCACAUAUGGGGCACACGUAUGUGCGCAAUUUGGGGCAUAGCACACGAUUAAAACUAUCGCGCACAGUAUGACUGUAGAUCACUGCCUCGGGUUCAUUGUUGUUCUCA ..(((((((((((((....)))))))..(((((((((((....((..((...)).)).(((.......)))))))))).))))...((((....)))).))))))............... ( -36.70) >DroMoj_CAF1 3497 120 - 1 UCGCCGGAUGCCCCACAAAUGGGGCAUACAUAGGUGCGCAGCUUGGGGCACAGCACUCGUCCGUAGGAGUCACGCACCGAAUGGCUGUUGACCACAGCUUCCGGUUCAUUGUUGUUUUCA ..(((((((((((((....)))))))..(((.((((((..((((.((((.........))))....))))..))))))..)))(((((.....))))).))))))............... ( -46.50) >DroAna_CAF1 10159 120 - 1 UCCCCAGAUGCUCCACAAAUGGGGCACACAUAAGUGCGUAGCUUGGGGCAAAGAACACGAUUGAAGUUGUCGCGAACCGUGUGGCUGUUAACCACUGCCUCCGGUUCGCUGUUGUUCUCG .((((((.(((((((....)))))))(((....)))......))))))...((((((.(((.......)))((((((((.(.(((.((.....)).))).)))))))))...)))))).. ( -46.50) >consensus UCCCCAGAUGCCCCACAUAUGGGGCACACAUAGGUGCGCAGCUUGGGGCACAGCACACGUCCGAAGGUGUCGCGCACCGAAUGGCUGUUGACCACAGCCUCGGGUUCAUUGUUGUUCUCA ...((((((((((((....)))))))......((((((..((((((..............))))....))..))))))....(((.((.....)).))))))))................ (-24.20 = -23.01 + -1.19)



| Location | 14,984,843 – 14,984,963 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 79.39 |
| Mean single sequence MFE | -43.03 |
| Consensus MFE | -28.77 |
| Energy contribution | -27.97 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.51 |
| Mean z-score | -1.50 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.41 |
| SVM RNA-class probability | 0.726184 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14984843 120 - 27905053 GAUCGGCUUCUUGGGGCAGUACUUAAUCGUGUGCGCCGAGUCCCCAGAUGCCCCGCAGAUGGGGCACACGUAGGUGCGUAGUUUGGGGCACAGCACUCGGUUAAAGUUAUCUCGCACUGA ..........(..(.((((.(((((((((.((((.....((((((((((((((((....))))))..((((....)))).)))))))).)).)))).))))).))))...)).)).)..) ( -43.90) >DroVir_CAF1 3937 120 - 1 AAUGGGUUUCUUGGGACAGUAUUUAAUGGUGUGCGCCGAGUCGCCGGAUGCUCCGCAUAUGGGGCACACAUAAGUGCGAAGCUUGGGACACAACACACGUCCGUACGCGUCGCGCACUGU ...............(((((........((((((.(((.((.((.((.....)))))).))).))))))....((((((.((.((((((.........)))).)).)).))))))))))) ( -41.80) >DroGri_CAF1 3674 120 - 1 UAUGGGUUUCUUGGGGCAAUACUUAAUGGUGUGUGCCGAGUCUCCCGAUGCUCCACAUAUGGGGCACACAUAGGUGCGCAAUUUGGGACACAAUACACGUCCGAAAGUGUCGCGCACGGA ..((((...(((((.(((.((((....))))))).)))))...)))).(((((((....))))))).......((((((..(((.((((.........)))).))).....))))))... ( -46.80) >DroWil_CAF1 12044 120 - 1 AAUAGGUUUCUUUGGGCAGUAUUUAAUGGUAUGCGCAGAAUCACCUGAUGCCCCACAUAUGGGGCACACGUAUGUGCGCAAUUUGGGGCAUAGCACACGAUUAAAACUAUCGCGCACAGU .........((.((.((.(((((((((.((.(((.(((......)))((((((((.((...(.(((((....))))).).)).)))))))).))).)).))))))..))).)).)).)). ( -39.40) >DroMoj_CAF1 3537 120 - 1 AAUAGGUUUCUUAGGACAGUACUUGAUGGUGUGCGCCGAGUCGCCGGAUGCCCCACAAAUGGGGCAUACAUAGGUGCGCAGCUUGGGGCACAGCACUCGUCCGUAGGAGUCACGCACCGA ....((((((((((((((((.......(.(((((.((((((((((.(((((((((....)))))))).)...))))....)))))).))))).)))).)))).))))))......))).. ( -50.11) >DroAna_CAF1 10199 120 - 1 GAUUGGCUUCUUGGGGCAAUAUUUAAUAGUGUGCGCCGAAUCCCCAGAUGCUCCACAAAUGGGGCACACAUAAGUGCGUAGCUUGGGGCAAAGAACACGAUUGAAGUUGUCGCGAACCGU ....((.(((..(.((((((.((((((.((((.........((((((.(((((((....)))))))(((....)))......))))))......)))).)))))))))))).)))))).. ( -36.16) >consensus AAUAGGUUUCUUGGGGCAGUACUUAAUGGUGUGCGCCGAGUCCCCAGAUGCCCCACAUAUGGGGCACACAUAGGUGCGCAGCUUGGGGCACAGCACACGUCCGAAGGUGUCGCGCACCGA ..((((...(((((.(((.((((....))))))).)))))...)))).(((((((....)))))))......((((((..((((((..............))))....))..)))))).. (-28.77 = -27.97 + -0.80)
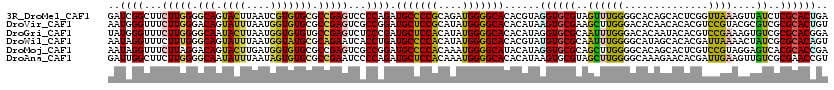


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:03:59 2006