
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 14,983,499 – 14,983,632 |
| Length | 133 |
| Max. P | 0.996805 |

| Location | 14,983,499 – 14,983,598 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 77.04 |
| Mean single sequence MFE | -36.42 |
| Consensus MFE | -22.61 |
| Energy contribution | -23.90 |
| Covariance contribution | 1.29 |
| Combinations/Pair | 1.38 |
| Mean z-score | -3.43 |
| Structure conservation index | 0.62 |
| SVM decision value | 2.51 |
| SVM RNA-class probability | 0.994762 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
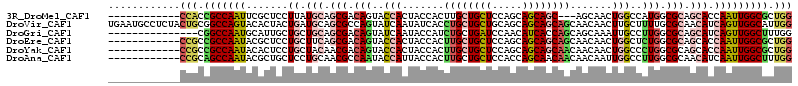
>3R_DroMel_CAF1 14983499 99 + 27905053 ------------CCACCGCCAAUUCGCUCCUUAUGCAGCGACAGUACCACUACCACUUGCUGCUCCAGCAGCAGC---AGCAACUGGCCAUGGCGCAGCACCAAUUGGCGCUGG ------------(((.((((((((((((.(....).)))))..(..(((...(((.((((((((.(....).)))---))))).)))...)))..).......))))))).))) ( -39.70) >DroVir_CAF1 2946 114 + 1 UGAAUGCCUCUACUGCGGCCAGUACACUACUGAUGCAGCGCCAGUAUCAAUAUCACCUGCUGCUGCAGCAGCAGCAGCAACAACUUGCUUUUGCGCAACAUCAGUUGGCAUUGG ..((((((..(((((....)))))....(((((((..((((.((((...........(((((((((....)))))))))......))))...))))..))))))).)))))).. ( -43.33) >DroGri_CAF1 2684 99 + 1 ---------------CGGCCAAUGCAUUGCUGCUGCAGCGACAGUAUCAAUACCAUCUGCUGAUCCAACAUCACCAGCAGCAAAUUGCCUUGGCGCAGCAUCAGUUGGCUUUGG ---------------..(((((.((((((((((((((((((..(((....)))..)).))))............)))))))))..))).))))).((((....))))....... ( -32.80) >DroEre_CAF1 3017 102 + 1 ------------CCGCCGCCAAUACGCUCCUGCUUCAGCGACAGUACCACUACCACUUGCUGCUCCAGCAGCAGCAGCAACAACUGGCUCUGGCGCAGCACCAAUUGGCGCUGG ------------(((.(((((((.((((........)))).................((((((.((((.(((..(((......)))))))))).))))))...))))))).))) ( -38.70) >DroYak_CAF1 2653 102 + 1 ------------CCGCCGCCAAUACACUCCUGCUACAACGACAGUACCACUACCACUUGCUGCUCCAGCAGCAGCAACAACAACUGGCCCUGGCGCAGCACCAAUUGGCGCUGG ------------(((.(((((((.......((((....((.(((..(((.......((((((((.....)))))))).......)))..))).)).))))...))))))).))) ( -32.44) >DroAna_CAF1 8682 102 + 1 ------------CCGCAGCCAAUACGCUGCUCCUGCAACGCCAAUACCAUUACCACUUGCUGCUCCACCAGCAACAACAACAAUUGGCCUUGGCGCAACAUCAAUUGGCUUUGG ------------(((.(((((((....(((....))).((((((..(((.......((((((......))))))..........)))..))))))........))))))).))) ( -31.53) >consensus ____________CCGCCGCCAAUACACUCCUGCUGCAGCGACAGUACCACUACCACUUGCUGCUCCAGCAGCAGCAGCAACAACUGGCCUUGGCGCAGCACCAAUUGGCGCUGG ............(((.(((((((.......((.(((.(((.(((..(((.......((((((((.....)))))))).......)))..))).))).))).))))))))).))) (-22.61 = -23.90 + 1.29)


| Location | 14,983,499 – 14,983,598 |
|---|---|
| Length | 99 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 77.04 |
| Mean single sequence MFE | -47.40 |
| Consensus MFE | -26.99 |
| Energy contribution | -27.99 |
| Covariance contribution | 1.01 |
| Combinations/Pair | 1.31 |
| Mean z-score | -3.90 |
| Structure conservation index | 0.57 |
| SVM decision value | 2.75 |
| SVM RNA-class probability | 0.996805 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
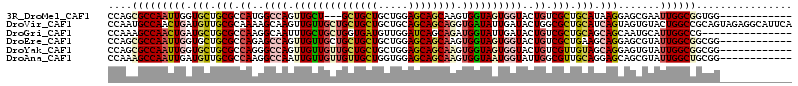
>3R_DroMel_CAF1 14983499 99 - 27905053 CCAGCGCCAAUUGGUGCUGCGCCAUGGCCAGUUGCU---GCUGCUGCUGGAGCAGCAAGUGGUAGUGGUACUGUCGCUGCAUAAGGAGCGAAUUGGCGGUGG------------ (((.(((((((.(((((..(......((((.(((((---(((.(....).)))))))).)))).)..))))).(((((.(....).)))))))))))).)))------------ ( -53.80) >DroVir_CAF1 2946 114 - 1 CCAAUGCCAACUGAUGUUGCGCAAAAGCAAGUUGUUGCUGCUGCUGCUGCAGCAGCAGGUGAUAUUGAUACUGGCGCUGCAUCAGUAGUGUACUGGCCGCAGUAGAGGCAUUCA ..((((((.((((((((.((((.....(((((..((((((((((....))))))))).)..)).)))......)))).))))))))..(.(((((....))))).))))))).. ( -52.20) >DroGri_CAF1 2684 99 - 1 CCAAAGCCAACUGAUGCUGCGCCAAGGCAAUUUGCUGCUGGUGAUGUUGGAUCAGCAGAUGGUAUUGAUACUGUCGCUGCAGCAGCAAUGCAUUGGCCG--------------- .....(((((.((.((((((((....))....((((((((((........)))))))(((((((....)))))))...)))))))))...)))))))..--------------- ( -37.30) >DroEre_CAF1 3017 102 - 1 CCAGCGCCAAUUGGUGCUGCGCCAGAGCCAGUUGUUGCUGCUGCUGCUGGAGCAGCAAGUGGUAGUGGUACUGUCGCUGAAGCAGGAGCGUAUUGGCGGCGG------------ .(((((((....)))))))((((...((((((((((.((((((((((....))))).((((..((.....))..))))..))))).)))).)))))))))).------------ ( -54.40) >DroYak_CAF1 2653 102 - 1 CCAGCGCCAAUUGGUGCUGCGCCAGGGCCAGUUGUUGUUGCUGCUGCUGGAGCAGCAAGUGGUAGUGGUACUGUCGUUGUAGCAGGAGUGUAUUGGCGGCGG------------ .(((((((....))))))).((((..((((.((((((((.(.......).)))))))).))))..)))).(((((((((..(((....)))..)))))))))------------ ( -45.40) >DroAna_CAF1 8682 102 - 1 CCAAAGCCAAUUGAUGUUGCGCCAAGGCCAAUUGUUGUUGUUGCUGGUGGAGCAGCAAGUGGUAAUGGUAUUGGCGUUGCAGGAGCAGCGUAUUGGCUGCGG------------ ((..(((((((..((((((((((((.((((.(((..(((((((((.....)))))))).)..))))))).))))))).((....))))))))))))))..))------------ ( -41.30) >consensus CCAACGCCAAUUGAUGCUGCGCCAAGGCCAGUUGUUGCUGCUGCUGCUGGAGCAGCAAGUGGUAGUGGUACUGUCGCUGCAGCAGGAGCGUAUUGGCGGCGG____________ ....(((((((((.(((.(((.((..((((.((((((((((((((.....)))))))).))))))))))..)).))).))).))).......))))))................ (-26.99 = -27.99 + 1.01)


| Location | 14,983,521 – 14,983,632 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 78.61 |
| Mean single sequence MFE | -42.08 |
| Consensus MFE | -25.75 |
| Energy contribution | -24.95 |
| Covariance contribution | -0.80 |
| Combinations/Pair | 1.35 |
| Mean z-score | -2.93 |
| Structure conservation index | 0.61 |
| SVM decision value | 1.91 |
| SVM RNA-class probability | 0.982267 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
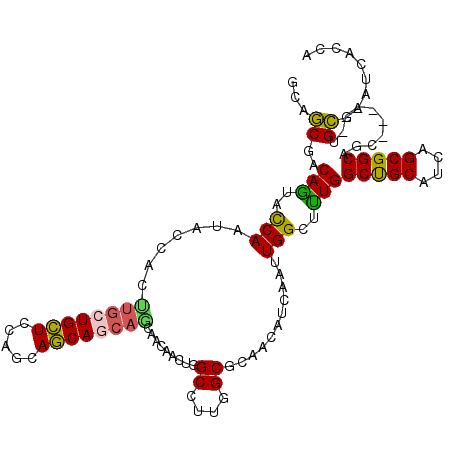
>3R_DroMel_CAF1 14983521 111 + 27905053 GCAGCGACAGUACCACUACCACUUGCUGCUCCAGCAGCAGC---AGCAACUGGCCAUGGCGCAGCACCAAUUGGCGCUGGCUGCAUCAGCGGCAGCGGCUAGUGCGAGUCACCA .....(((...............(((((((.....))))))---)(((.((((((......((((.((....)).))))(((((.......))))))))))))))..))).... ( -46.40) >DroVir_CAF1 2980 108 + 1 GCAGCGCCAGUAUCAAUAUCACCUGCUGCUGCAGCAGCAGCAGCAACAACUUGCUUUUGCGCAACAUCAGUUGGCAUUGGCCGCCUCAGCGGCUGC------UGCGAAUCAUCA ((((((((((.............(((((((((....))))))))).....((((......))))......)))))...((((((....))))))))------)))......... ( -46.80) >DroGri_CAF1 2703 108 + 1 GCAGCGACAGUAUCAAUACCAUCUGCUGAUCCAACAUCACCAGCAGCAAAUUGCCUUGGCGCAGCAUCAGUUGGCUUUGGCCGCUUCAGCGGCUGC------UGCGAAUCAUCA .((((((..(((....)))..)).))))...........((((((((.....(((..(((.((((....)))))))..)))(((....))))))))------)).)........ ( -36.60) >DroEre_CAF1 3039 114 + 1 UCAGCGACAGUACCACUACCACUUGCUGCUCCAGCAGCAGCAGCAACAACUGGCUCUGGCGCAGCACCAAUUGGCGCUGGCUGCAUCAGCGGCAGCGGCUAGUGCGAAUCACCA ((.(((.(((..(((.......((((((((.....)))))))).......)))..))).))).((((.....(.(((((.((((....))))))))).)..))))))....... ( -43.94) >DroMoj_CAF1 2650 108 + 1 GCAGCGCCAGUACCAAUAUCACUUACUGCUGCAACAGCAGCAGCAGCAGUUGGCUUUGGCGAAACAUCAGUUGGCUUUGGCUGCGUCUGCGGCCGC------UGCGAAUCACCA ((((((((((..(((((....((..(((((((....))))))).))..)))))..)))))..................((((((....))))))))------)))......... ( -45.40) >DroAna_CAF1 8704 114 + 1 GCAACGCCAAUACCAUUACCACUUGCUGCUCCACCAGCAACAACAACAAUUGGCCUUGGCGCAACAUCAAUUGGCUUUGGCUGCAUCGGCGGCCGCGGUAAAUGUUAAUAAUCA ....((((((..(((.......((((((......))))))..........)))..)))))).(((((......(((.(((((((....))))))).)))..)))))........ ( -33.33) >consensus GCAGCGACAGUACCAAUACCACUUGCUGCUCCAGCAGCAGCAGCAACAACUGGCCUUGGCGCAACAUCAAUUGGCUUUGGCUGCAUCAGCGGCAGC______UGCGAAUCACCA ...((..(((..(((.......((((((((.....)))))))).........((....))...........)))..)))(((((....)))))..........))......... (-25.75 = -24.95 + -0.80)


| Location | 14,983,521 – 14,983,632 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 78.61 |
| Mean single sequence MFE | -48.02 |
| Consensus MFE | -23.59 |
| Energy contribution | -25.12 |
| Covariance contribution | 1.53 |
| Combinations/Pair | 1.36 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.49 |
| SVM decision value | 1.53 |
| SVM RNA-class probability | 0.961409 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
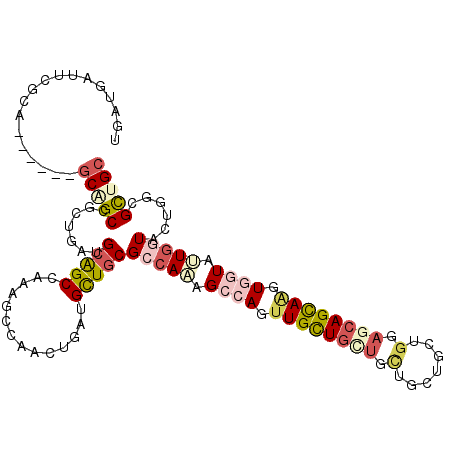
>3R_DroMel_CAF1 14983521 111 - 27905053 UGGUGACUCGCACUAGCCGCUGCCGCUGAUGCAGCCAGCGCCAAUUGGUGCUGCGCCAUGGCCAGUUGCU---GCUGCUGCUGGAGCAGCAAGUGGUAGUGGUACUGUCGCUGC .((((((........((((((((((((...((((((((((((....))))))).((....)).....)))---))((((((....))))))))))))))))))...)))))).. ( -57.90) >DroVir_CAF1 2980 108 - 1 UGAUGAUUCGCA------GCAGCCGCUGAGGCGGCCAAUGCCAACUGAUGUUGCGCAAAAGCAAGUUGUUGCUGCUGCUGCUGCAGCAGCAGGUGAUAUUGAUACUGGCGCUGC .........(((------((.(((((....)))))....((((..((((((..(((....))......((((((((((....)))))))))))..))))))....))))))))) ( -49.50) >DroGri_CAF1 2703 108 - 1 UGAUGAUUCGCA------GCAGCCGCUGAAGCGGCCAAAGCCAACUGAUGCUGCGCCAAGGCAAUUUGCUGCUGGUGAUGUUGGAUCAGCAGAUGGUAUUGAUACUGUCGCUGC ....((((((((------((((((((....)))))((........)).))))))((((.(((.....)))..))))......)))))(((.(((((((....)))))))))).. ( -40.60) >DroEre_CAF1 3039 114 - 1 UGGUGAUUCGCACUAGCCGCUGCCGCUGAUGCAGCCAGCGCCAAUUGGUGCUGCGCCAGAGCCAGUUGUUGCUGCUGCUGCUGGAGCAGCAAGUGGUAGUGGUACUGUCGCUGA .((((((........((((((((((((...(((((.((((.((((((((.(((...))).)))))))).)))))))))(((((...)))))))))))))))))...)))))).. ( -55.90) >DroMoj_CAF1 2650 108 - 1 UGGUGAUUCGCA------GCGGCCGCAGACGCAGCCAAAGCCAACUGAUGUUUCGCCAAAGCCAACUGCUGCUGCUGCUGUUGCAGCAGUAAGUGAUAUUGGUACUGGCGCUGC .........(((------(((((.((....)).)))...((((((..(((((.(.....(((.....)))((((((((....))))))))..).)))))..))..))))))))) ( -45.60) >DroAna_CAF1 8704 114 - 1 UGAUUAUUAACAUUUACCGCGGCCGCCGAUGCAGCCAAAGCCAAUUGAUGUUGCGCCAAGGCCAAUUGUUGUUGUUGCUGGUGGAGCAGCAAGUGGUAAUGGUAUUGGCGUUGC ..................(((((.((((((...((((..((((.(((.((((.(((((..((((((....))))..))))))).)))).))).))))..))))))))))))))) ( -38.60) >consensus UGAUGAUUCGCA______GCAGCCGCUGAUGCAGCCAAAGCCAACUGAUGCUGCGCCAAAGCCAGUUGCUGCUGCUGCUGCUGGAGCAGCAAGUGGUAUUGGUACUGGCGCUGC ..................(((((.......(((((..............)))))(((((.((((.((((((((.(.......).)))))))).)))).)))))......))))) (-23.59 = -25.12 + 1.53)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:03:54 2006