
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 14,928,291 – 14,928,489 |
| Length | 198 |
| Max. P | 0.998470 |

| Location | 14,928,291 – 14,928,411 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.82 |
| Mean single sequence MFE | -40.00 |
| Consensus MFE | -35.88 |
| Energy contribution | -36.16 |
| Covariance contribution | 0.28 |
| Combinations/Pair | 1.12 |
| Mean z-score | -2.17 |
| Structure conservation index | 0.90 |
| SVM decision value | 3.11 |
| SVM RNA-class probability | 0.998470 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14928291 120 - 27905053 AAGGGGCAGUGCUGCUGAUCGUUAGGGCUAGCAGUGACUUACUGUUCUUCUUGAUUGACAUGCUUAGCACUCCAGAUGCUAUCGGUAUCUGUAUCUAGCAUCCAACAAUCUGUGGCCCCA ..(((((((((((((((.(((((((((..(((((((...)))))))..)))))).))))).))..))))))...(((((((..((((....)))))))))))............))))). ( -42.30) >DroSec_CAF1 58287 120 - 1 AAGGGGCAGCGCUGCUGAUCGUUAGGGCUAGCAGCGACUUACUGUUCUUCUUGAUUGACAUGCUUAGCACUCCAGAUGCUAUCUGUAUCUGUAUCUGGCAUUCAACAAUCUGUGGCCACA .((((((((((((((((..(.....)..)))))))).....)))))))).(((.((((.(((((.((.....(((((((.....)))))))...))))))))))))))............ ( -37.00) >DroSim_CAF1 56980 120 - 1 AAGGGGCAGUGCUGCUGAUCGUUAGGGCUAGCAGCGACUUACUGUUCUUCUUGAUUGACAUGCUUAGCACUCCAGAUGCUAUCUGUAUCUGUAUCUGGCAUCCAACAAUCUGUGGCCCCA ..(((((((((((((((..(.....)..))))))).))).............(((((..(((((.((.....(((((((.....)))))))...)))))))....)))))....))))). ( -42.00) >DroEre_CAF1 54834 114 - 1 AAGGGGCAGAGCUGCUGAUCGUUAGGGCUAGCAGCGACUUACUGUUCUUCUUGAUUGACAUGCUUAGCACUCCAGAU------GCUAUCUGUAUCUGGCAUCCAACUAUCUGUGGCCCCA ..(((((((((((((((..(.....)..)))))))(((.....)))..))).......((((..(((....((((((------((.....)))))))).......)))..))))))))). ( -39.00) >DroYak_CAF1 61655 118 - 1 AAGGGGCAGAGCUGCUGAUCGUUAGGGCUAGCAGCGACUUACUGUUCUUCUUGAUUGACAUGCUUAGCACUGUAUAUGG--GCGCUAUCUGUAUCUGGCAUCCAACAAUCUGUGGCCCCA ..(((((((((((((((..(.....)..)))))))(((.....)))..))).(((((((((((...))).)))...(((--..((((........))))..))).)))))....))))). ( -39.70) >consensus AAGGGGCAGAGCUGCUGAUCGUUAGGGCUAGCAGCGACUUACUGUUCUUCUUGAUUGACAUGCUUAGCACUCCAGAUGCUAUCGGUAUCUGUAUCUGGCAUCCAACAAUCUGUGGCCCCA ..(((((...(((((((..(.....)..))))))).................(((((..(((((.((.....(((((((.....)))))))...)))))))....)))))....))))). (-35.88 = -36.16 + 0.28)


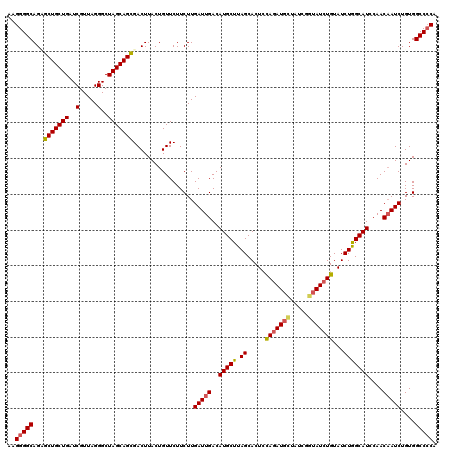
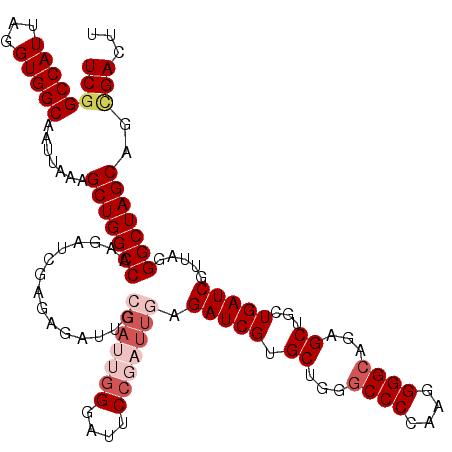
| Location | 14,928,371 – 14,928,489 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 89.23 |
| Mean single sequence MFE | -48.04 |
| Consensus MFE | -36.72 |
| Energy contribution | -38.80 |
| Covariance contribution | 2.08 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.86 |
| Structure conservation index | 0.76 |
| SVM decision value | 1.99 |
| SVM RNA-class probability | 0.985033 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14928371 118 - 27905053 UCGGCCAUUAGGUGGCAAUUAAAGCUGGCCAAGAUCGA--GAUUGCGAUUGGGAUGCCGAUUGAGAUCGUGCUGGGCCCCAAGGGGCAGUGCUGCUGAUCGUUAGGGCUAGCAGUGACUU ...(((((...))))).......(((((((..(((((.--(((..(((((((....)))))))..)))(..((..((((....))))))..)...))))).....)))))))........ ( -48.30) >DroSec_CAF1 58367 120 - 1 UCGGCCAUUAGGUGGCAAUUAAAGCUGGCCAAGAUCGAGAGAUUGCGAUUGGGAUUCCGAUUGAGAUCGUGCUGGGCCCCAAGGGGCAGCGCUGCUGAUCGUUAGGGCUAGCAGCGACUU ((((((((...))))).......(((((((..(((((...(((..(((((((....)))))))..)))(((((..((((....)))))))))...))))).....)))))))..)))... ( -52.20) >DroSim_CAF1 57060 120 - 1 UCGGCCAUUAGGUGGCAAUUAAAGCUGGCCAAGAUCGAGAGAUUGCGAUUGGGAUUCCGAUUGAGAUCGUGCUGGGCCCCAAGGGGCAGUGCUGCUGAUCGUUAGGGCUAGCAGCGACUU ((((((((...))))).......(((((((..(((((...(((..(((((((....)))))))..)))(..((..((((....))))))..)...))))).....)))))))..)))... ( -49.80) >DroEre_CAF1 54908 120 - 1 UCAGCCAUUAGGUGGCAAUUAAAGCUGGCCAAGAUUUGGAUUCCGAGACAGGGAUUCCGAUUGAGAUCGUGCUGGGCCCCAAGGGGCAGAGCUGCUGAUCGUUAGGGCUAGCAGCGACUU ...(((((...))))).......(((((((..((((.(((((((......))))))).))))..(((((.(((..((((....))))..)))...))))).....)))))))........ ( -49.00) >DroYak_CAF1 61733 106 - 1 UCGGCCAUUAGGUGGCAAUUAAUGCUGGCCAAGAUU--------------GGCAUUCAGAUUGAGAUCGUGCUGGACCCCAAGGGGCAGAGCUGCUGAUCGUUAGGGCUAGCAGCGACUU ((((((((...)))))......((((((((..(((.--------------.(((....(((....)))..(((.(.(((....))))..))))))..))).....)))))))).)))... ( -40.90) >consensus UCGGCCAUUAGGUGGCAAUUAAAGCUGGCCAAGAUCGAGAGAUUGCGAUUGGGAUUCCGAUUGAGAUCGUGCUGGGCCCCAAGGGGCAGAGCUGCUGAUCGUUAGGGCUAGCAGCGACUU ((((((((...))))).......(((((((...............(((((((....))))))).(((((.((...((((....))))...))...))))).....)))))))..)))... (-36.72 = -38.80 + 2.08)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:02:11 2006