
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 14,831,475 – 14,831,823 |
| Length | 348 |
| Max. P | 0.980837 |

| Location | 14,831,475 – 14,831,583 |
|---|---|
| Length | 108 |
| Sequences | 3 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.22 |
| Mean single sequence MFE | -35.77 |
| Consensus MFE | -31.49 |
| Energy contribution | -31.27 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.88 |
| SVM decision value | 0.12 |
| SVM RNA-class probability | 0.592403 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14831475 108 + 27905053 CUG------------GCAUAGUAUUAGUGGAAGUGGUACCCACUCCGCCCCCUUAGAACUCGUUUUGUGCAAAUUAUGCUACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCUAGAU (((------------((((((.(((.(((((.((((...)))))))))........(((.(((((((((((.....))).)))))))).))))))))))).(((((...))))))))).. ( -32.90) >DroSec_CAF1 9647 120 + 1 CUGCUGGUGCACGAAGCAUAGUAUUAGUGGAAGUGGUACCCACUCCGCCCUCUUAGAACUCGUUUUGUGCAAAUUAUGCUACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCCAGAU ((((.((((((.....(((((.(((.(((((.((((...)))))))))........(((.(((((((((((.....))).)))))))).)))))))))))......)))))).).))).. ( -37.20) >DroSim_CAF1 9810 120 + 1 CUGCUGGUGCACGAAGCAUAGUAUUAGUGGAAGUGGUACCCACUCCGCCCUCUUAGAACUCGUUUUGUGCAAAUUAUGCUACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCCAGAU ((((.((((((.....(((((.(((.(((((.((((...)))))))))........(((.(((((((((((.....))).)))))))).)))))))))))......)))))).).))).. ( -37.20) >consensus CUGCUGGUGCACGAAGCAUAGUAUUAGUGGAAGUGGUACCCACUCCGCCCUCUUAGAACUCGUUUUGUGCAAAUUAUGCUACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCCAGAU (((.............(((((.(((.(((((.((((...)))))))))........(((.(((((((((((.....))).)))))))).))))))))))).(((((...))))).))).. (-31.49 = -31.27 + -0.22)


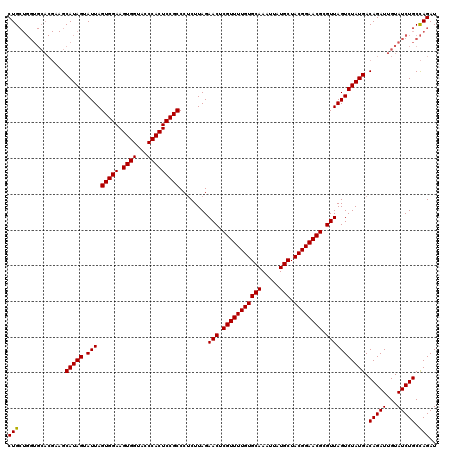
| Location | 14,831,503 – 14,831,623 |
|---|---|
| Length | 120 |
| Sequences | 3 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 97.78 |
| Mean single sequence MFE | -29.37 |
| Consensus MFE | -28.73 |
| Energy contribution | -29.07 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.03 |
| Structure conservation index | 0.98 |
| SVM decision value | 1.00 |
| SVM RNA-class probability | 0.897789 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14831503 120 + 27905053 CACUCCGCCCCCUUAGAACUCGUUUUGUGCAAAUUAUGCUACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCUAGAUACAGCAUACAUAUAUGUAUGCACCAUAUAUAGCCAUUCGU ................(((.(((((((((((.....))).)))))))).))).(.(((((......((((((.....))))))(((((((....)))))))......))))).)...... ( -30.60) >DroSec_CAF1 9687 120 + 1 CACUCCGCCCUCUUAGAACUCGUUUUGUGCAAAUUAUGCUACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCCAGAUACAGCAUACAUAUAUGUAUGCACCAUAUAUAGCUAUUCGU ................(((.(((((((((((.....))).)))))))).)))((.(((((......((((((.....))))))(((((((....)))))))......)))))))...... ( -31.20) >DroSim_CAF1 9850 120 + 1 CACUCCGCCCUCUUAGAACUCGUUUUGUGCAAAUUAUGCUACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCCAGAUACAGCAUACAUAUAUGUAAGCACCAUAUAUAGCCAUUCGU ................(((.(((((((((((.....))).)))))))).))).(.(((((......((((((.....))))))((.((((....)))).))......))))).)...... ( -26.30) >consensus CACUCCGCCCUCUUAGAACUCGUUUUGUGCAAAUUAUGCUACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCCAGAUACAGCAUACAUAUAUGUAUGCACCAUAUAUAGCCAUUCGU ......((........(((.(((((((((((.....))).)))))))).)))....((((......((((((.....))))))(((((((....)))))))..))))....))....... (-28.73 = -29.07 + 0.33)



| Location | 14,831,543 – 14,831,663 |
|---|---|
| Length | 120 |
| Sequences | 3 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.33 |
| Mean single sequence MFE | -38.73 |
| Consensus MFE | -38.00 |
| Energy contribution | -38.33 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.98 |
| SVM decision value | 1.21 |
| SVM RNA-class probability | 0.931556 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14831543 120 + 27905053 ACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCUAGAUACAGCAUACAUAUAUGUAUGCACCAUAUAUAGCCAUUCGUGGAUGCCGACGUGCCGGCGGAGCUUUUAUGCUGCACGUAC .(((..((((((.((((((((.....((((((.....))))))(((((((....)))))))...............))))))))...)))))))))(((((((......))).).))).. ( -39.40) >DroSec_CAF1 9727 120 + 1 ACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCCAGAUACAGCAUACAUAUAUGUAUGCACCAUAUAUAGCUAUUCGUGGAUGCCGACGUGCCGGCGGAGCUUUUAUGCUGCACGUAC .(((..((((.(((.(((((......((((((.....))))))(((((((....)))))))......))))))))..))))....)))((((((.((((.........)))))))))).. ( -41.70) >DroSim_CAF1 9890 120 + 1 ACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCCAGAUACAGCAUACAUAUAUGUAAGCACCAUAUAUAGCCAUUCGUGGAUGCCGACGUGCCGGCGGAGCUUUUAUGCUGCACGUAC .(((..((((((.((((((((.....((((((.....))))))((.((((....)))).))...............))))))))...)))))))))(((((((......))).).))).. ( -35.10) >consensus ACGGAACGCGUUAGUCUAUGACAGAUUGUAUCUGCCAGAUACAGCAUACAUAUAUGUAUGCACCAUAUAUAGCCAUUCGUGGAUGCCGACGUGCCGGCGGAGCUUUUAUGCUGCACGUAC .(((..((((((.((((((((.....((((((.....))))))(((((((....)))))))...............))))))))...)))))))))(((((((......))).).))).. (-38.00 = -38.33 + 0.33)

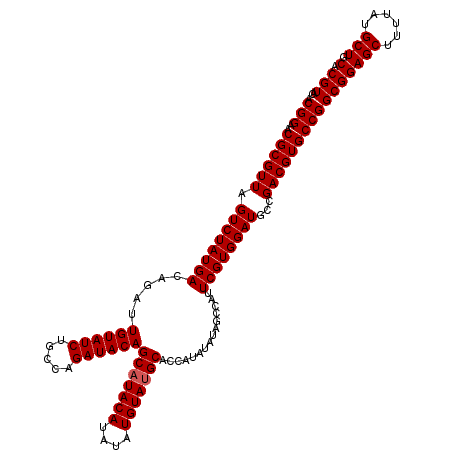

| Location | 14,831,543 – 14,831,663 |
|---|---|
| Length | 120 |
| Sequences | 3 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.33 |
| Mean single sequence MFE | -35.30 |
| Consensus MFE | -34.24 |
| Energy contribution | -33.80 |
| Covariance contribution | -0.44 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.97 |
| SVM decision value | 0.21 |
| SVM RNA-class probability | 0.634442 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14831543 120 - 27905053 GUACGUGCAGCAUAAAAGCUCCGCCGGCACGUCGGCAUCCACGAAUGGCUAUAUAUGGUGCAUACAUAUAUGUAUGCUGUAUCUAGCAGAUACAAUCUGUCAUAGACUAACGCGUUCCGU ..((((((((((((........(((((....))))).............((((((((.......)))))))))))))))).((((((((((...))))))..)))).....))))..... ( -34.20) >DroSec_CAF1 9727 120 - 1 GUACGUGCAGCAUAAAAGCUCCGCCGGCACGUCGGCAUCCACGAAUAGCUAUAUAUGGUGCAUACAUAUAUGUAUGCUGUAUCUGGCAGAUACAAUCUGUCAUAGACUAACGCGUUCCGU ..((((((((((((........(((((....))))).............((((((((.......))))))))))))))))...((((((((...)))))))).........))))..... ( -35.70) >DroSim_CAF1 9890 120 - 1 GUACGUGCAGCAUAAAAGCUCCGCCGGCACGUCGGCAUCCACGAAUGGCUAUAUAUGGUGCUUACAUAUAUGUAUGCUGUAUCUGGCAGAUACAAUCUGUCAUAGACUAACGCGUUCCGU ..(((((((((((..((((.(((((((....)))))..(((....)))........)).))))(((....))))))))))...((((((((...)))))))).........))))..... ( -36.00) >consensus GUACGUGCAGCAUAAAAGCUCCGCCGGCACGUCGGCAUCCACGAAUGGCUAUAUAUGGUGCAUACAUAUAUGUAUGCUGUAUCUGGCAGAUACAAUCUGUCAUAGACUAACGCGUUCCGU ..((((((((((((..((((..(((((....))))).((...))..))))(((((((.......))))))).)))))))).((((((((((...))))))..)))).....))))..... (-34.24 = -33.80 + -0.44)

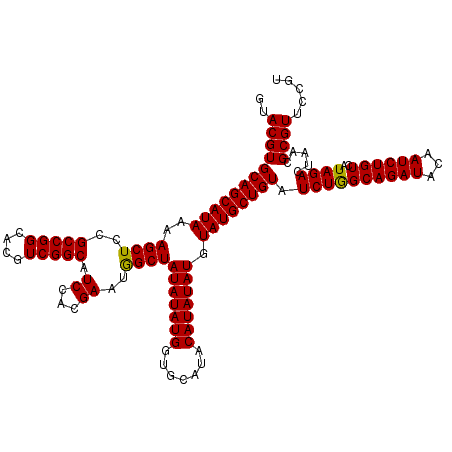
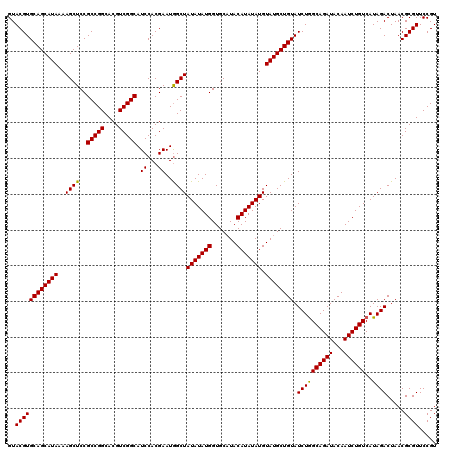
| Location | 14,831,583 – 14,831,703 |
|---|---|
| Length | 120 |
| Sequences | 3 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.89 |
| Mean single sequence MFE | -43.27 |
| Consensus MFE | -43.10 |
| Energy contribution | -43.43 |
| Covariance contribution | 0.33 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.65 |
| Structure conservation index | 1.00 |
| SVM decision value | 1.87 |
| SVM RNA-class probability | 0.980837 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14831583 120 + 27905053 ACAGCAUACAUAUAUGUAUGCACCAUAUAUAGCCAUUCGUGGAUGCCGACGUGCCGGCGGAGCUUUUAUGCUGCACGUACCGGCGUCUCUCUGGGCGUCAAUUUAUGGCCAGGGUAAAAA ...(((((((....)))))))..........(((......((((((((((((((.((((.........))))))))))..))))))))..((((.(((......))).)))))))..... ( -44.80) >DroSec_CAF1 9767 120 + 1 ACAGCAUACAUAUAUGUAUGCACCAUAUAUAGCUAUUCGUGGAUGCCGACGUGCCGGCGGAGCUUUUAUGCUGCACGUACCGGCGUCUCUCUGGGCGUCAAUUUAUGGCCAGGGUAAAAA ...(((((((....)))))))(((................((((((((((((((.((((.........))))))))))..))))))))..((((.(((......))).)))))))..... ( -44.50) >DroSim_CAF1 9930 120 + 1 ACAGCAUACAUAUAUGUAAGCACCAUAUAUAGCCAUUCGUGGAUGCCGACGUGCCGGCGGAGCUUUUAUGCUGCACGUACCGGCGUCUCUCUGGGCGUCAAUUUAUGGCCAGGGUAAAAA ...((.((((....)))).))..........(((......((((((((((((((.((((.........))))))))))..))))))))..((((.(((......))).)))))))..... ( -40.50) >consensus ACAGCAUACAUAUAUGUAUGCACCAUAUAUAGCCAUUCGUGGAUGCCGACGUGCCGGCGGAGCUUUUAUGCUGCACGUACCGGCGUCUCUCUGGGCGUCAAUUUAUGGCCAGGGUAAAAA ...(((((((....)))))))(((................((((((((((((((.((((.........))))))))))..))))))))..((((.(((......))).)))))))..... (-43.10 = -43.43 + 0.33)


| Location | 14,831,623 – 14,831,743 |
|---|---|
| Length | 120 |
| Sequences | 3 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.89 |
| Mean single sequence MFE | -40.67 |
| Consensus MFE | -40.75 |
| Energy contribution | -40.53 |
| Covariance contribution | -0.22 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.30 |
| Structure conservation index | 1.00 |
| SVM decision value | -0.06 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14831623 120 - 27905053 CUGCUCCUAAGUGCGGCGGUGCUGGUUCUUUUUAGCACAAUUUUUACCCUGGCCAUAAAUUGACGCCCAGAGAGACGCCGGUACGUGCAGCAUAAAAGCUCCGCCGGCACGUCGGCAUCC .((((.....(((((((((((((((((((((((.((.((((((...(....)....))))))..))..))))))).))))))))....(((......))).)))).))))...))))... ( -39.80) >DroSec_CAF1 9807 120 - 1 CUGCUCCUAAGUGCAGCGGUGCUGGCUCUUUUUAGCACAAUUUUUACCCUGGCCAUAAAUUGACGCCCAGAGAGACGCCGGUACGUGCAGCAUAAAAGCUCCGCCGGCACGUCGGCAUCC ..(((.....((((.((((((((((((((((((.((.((((((...(....)....))))))..))..))))))).)))))))).))).))))...)))...(((((....))))).... ( -41.10) >DroSim_CAF1 9970 120 - 1 CUGCUCCUAAGUGCAGCGGUGCUGGCUCUUUUUAGCACAAUUUUUACCCUGGCCAUAAAUUGACGCCCAGAGAGACGCCGGUACGUGCAGCAUAAAAGCUCCGCCGGCACGUCGGCAUCC ..(((.....((((.((((((((((((((((((.((.((((((...(....)....))))))..))..))))))).)))))))).))).))))...)))...(((((....))))).... ( -41.10) >consensus CUGCUCCUAAGUGCAGCGGUGCUGGCUCUUUUUAGCACAAUUUUUACCCUGGCCAUAAAUUGACGCCCAGAGAGACGCCGGUACGUGCAGCAUAAAAGCUCCGCCGGCACGUCGGCAUCC ..(((.....((((.((((((((((((((((((.((.((((((...(....)....))))))..))..))))))).)))))))).))).))))...)))...(((((....))))).... (-40.75 = -40.53 + -0.22)



| Location | 14,831,703 – 14,831,823 |
|---|---|
| Length | 120 |
| Sequences | 3 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.44 |
| Mean single sequence MFE | -40.33 |
| Consensus MFE | -36.79 |
| Energy contribution | -36.80 |
| Covariance contribution | 0.01 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.97 |
| Structure conservation index | 0.91 |
| SVM decision value | 0.51 |
| SVM RNA-class probability | 0.763143 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14831703 120 - 27905053 UAAAGCCGCAAGUAAAUGAGAUUUACACAAGAUGUUGGCCAACUAGAGAGAACGGUACAGAGUACCCAGCGGGCCAGCUGCUGCUCCUAAGUGCGGCGGUGCUGGUUCUUUUUAGCACAA ....((((((.((((((...)))))).......(((((((..((.(......)(((((...))))).))..))))))).............)))))).(((((((......))))))).. ( -40.30) >DroSec_CAF1 9887 120 - 1 UAAAGCCGCAAGUAAAUGAGAUUUGCACAAGAUGUUGGCCAACUAGAGAGAACGGAAUGGAGUACCCAGCAGGCCAGCUGCUGCUCCUAAGUGCAGCGGUGCUGGCUCUUUUUAGCACAA ....(((((((((.......))))))((.....)).)))...(((((((((......(((.....)))....((((((((((((........))))))..)))))))))))))))..... ( -39.50) >DroSim_CAF1 10050 120 - 1 UGAAGCCGCAAGUAAAUGAGAUUUGCACAAGAUGUUGGCCAACUAGAGAGAACGGUGUGGAGUACCCAGCAAGCCAGCUGCUGCUCCUAAGUGCAGCGGUGCUGGCUCUUUUUAGCACAA ....(((((((((.......))))))((.....)).)))...(((((((((..(.((.((....)))).)..((((((((((((........))))))..)))))))))))))))..... ( -41.20) >consensus UAAAGCCGCAAGUAAAUGAGAUUUGCACAAGAUGUUGGCCAACUAGAGAGAACGGUAUGGAGUACCCAGCAGGCCAGCUGCUGCUCCUAAGUGCAGCGGUGCUGGCUCUUUUUAGCACAA ....(((((((((.......))))))((.....)).)))...(((((((((..((((.....))))......((((((((((((........))))))..)))))))))))))))..... (-36.79 = -36.80 + 0.01)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 12:00:22 2006