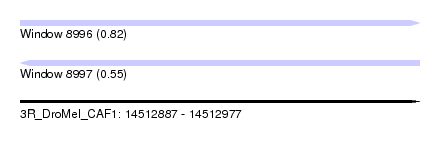
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 14,512,887 – 14,512,977 |
| Length | 90 |
| Max. P | 0.818566 |

| Location | 14,512,887 – 14,512,977 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | forward |
| Mean pairwise identity | 75.68 |
| Mean single sequence MFE | -29.98 |
| Consensus MFE | -6.10 |
| Energy contribution | -7.02 |
| Covariance contribution | 0.92 |
| Combinations/Pair | 1.14 |
| Mean z-score | -3.16 |
| Structure conservation index | 0.20 |
| SVM decision value | 0.67 |
| SVM RNA-class probability | 0.818566 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14512887 90 + 27905053 --C------CAUCUGAGAUCUCAACUGCUUGCAAUUAGCAUGCUAACGAUGCA--GUUGCAGUUGCAUCUGCAGUUGGCUCUGUUGCAUCUGCAGUGGCA --(------((.(((((((..(((((((.(((((((.((((((.......)))--..))))))))))...)))))))((......)))))).)))))).. ( -33.90) >DroSec_CAF1 9849 90 + 1 --C------CAUCUGCGAUCUCAAGUGCUUGCAAUUAGCAUGCUAACGAUGCA--GUUGCAGUUGCAAUUGCAGUUGGCUCUGUUCCAUCUGCAGUGGCA --(------((.(((((((.....(((((.......)))))((((((..((((--(((((....)))))))))))))))........))).))))))).. ( -33.30) >DroSim_CAF1 9817 90 + 1 --C------CAUCUGCGAUCUCAACUGCUUGCAAUUAGCAUGCUAACGAGGCA--GUUGCAGUUGCAAUUGCAGUUGGCUCUGUUGCAUCUGCAGUGGCA --(------((.(((((((..((((((((.......)))).((((((...(((--(((((....)))))))).))))))...)))).))).))))))).. ( -35.80) >DroEre_CAF1 9247 86 + 1 AACCAUAGAGAUCUGCGAUCUCAACUGGUUUCAAUUAGCAUGCUAAUGAUGCA--GUUGCAGCUGU------------AUCUGUUGCAUCUGCUGUGGCA (((((..((((((...))))))...)))))..........(((((.....(((--(.((((((...------------....)))))).))))..))))) ( -29.00) >DroYak_CAF1 9032 87 + 1 --C------CAUCUGCGAUCUCAACUGGUUUCAAUUAGCAUGCUAAUGAUGCA--GUUGCUGUUGAAUCU---GUUGGAUCUGUUGCAUCUGCUGUGGCA --.------.......((((.((((.((.((((((.(((((((.......)))--..)))))))))).))---)))))))).((..((.....))..)). ( -26.00) >DroAna_CAF1 8839 80 + 1 A-C------AAUCUGCCAUCUCAAGUAGUCUCAAUUAGCAUGCUAAUGAAGCUUGCUUGAAGCUACUAUUGC----------UUUGCAGUUGCU---GCA .-(------((.((((.......(((((..(((...((((.(((.....))).)))))))..))))).....----------...)))))))..---... ( -21.86) >consensus __C______CAUCUGCGAUCUCAACUGCUUGCAAUUAGCAUGCUAACGAUGCA__GUUGCAGUUGCAAUUGC_GUUGGCUCUGUUGCAUCUGCAGUGGCA .....................................((..((....((((((.....((((..................)))))))))).))....)). ( -6.10 = -7.02 + 0.92)



| Location | 14,512,887 – 14,512,977 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 100 |
| Reading direction | reverse |
| Mean pairwise identity | 75.68 |
| Mean single sequence MFE | -26.24 |
| Consensus MFE | -7.11 |
| Energy contribution | -8.13 |
| Covariance contribution | 1.03 |
| Combinations/Pair | 1.06 |
| Mean z-score | -2.48 |
| Structure conservation index | 0.27 |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.551752 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14512887 90 - 27905053 UGCCACUGCAGAUGCAACAGAGCCAACUGCAGAUGCAACUGCAAC--UGCAUCGUUAGCAUGCUAAUUGCAAGCAGUUGAGAUCUCAGAUG------G-- ..((((((.((((((......))(((((((.((((((........--))))))....(((.......)))..)))))))..))))))).))------)-- ( -30.70) >DroSec_CAF1 9849 90 - 1 UGCCACUGCAGAUGGAACAGAGCCAACUGCAAUUGCAACUGCAAC--UGCAUCGUUAGCAUGCUAAUUGCAAGCACUUGAGAUCGCAGAUG------G-- ..(((.(((((.(((.......))).))))).((((....))))(--((((((....((.(((.....))).))......))).)))).))------)-- ( -26.40) >DroSim_CAF1 9817 90 - 1 UGCCACUGCAGAUGCAACAGAGCCAACUGCAAUUGCAACUGCAAC--UGCCUCGUUAGCAUGCUAAUUGCAAGCAGUUGAGAUCGCAGAUG------G-- ..(((((((.(((((......))(((((((..((((((..(((..--(((.......))))))...)))))))))))))..))))))).))------)-- ( -30.30) >DroEre_CAF1 9247 86 - 1 UGCCACAGCAGAUGCAACAGAU------------ACAGCUGCAAC--UGCAUCAUUAGCAUGCUAAUUGAAACCAGUUGAGAUCGCAGAUCUCUAUGGUU .......((((.((((.(....------------...).)))).)--))).(((((((....)))).)))(((((...((((((...))))))..))))) ( -25.80) >DroYak_CAF1 9032 87 - 1 UGCCACAGCAGAUGCAACAGAUCCAAC---AGAUUCAACAGCAAC--UGCAUCAUUAGCAUGCUAAUUGAAACCAGUUGAGAUCGCAGAUG------G-- (((....((....))....((((((((---.(.(((((.((((..--(((.......)))))))..))))).)..)))).)))))))....------.-- ( -20.50) >DroAna_CAF1 8839 80 - 1 UGC---AGCAACUGCAAA----------GCAAUAGUAGCUUCAAGCAAGCUUCAUUAGCAUGCUAAUUGAGACUACUUGAGAUGGCAGAUU------G-U ...---.((((((((...----------.....(((((..(((((((.(((.....))).))))...)))..))))).......)))).))------)-) ( -23.76) >consensus UGCCACAGCAGAUGCAACAGAGCCAAC_GCAAUUGCAACUGCAAC__UGCAUCAUUAGCAUGCUAAUUGAAACCAGUUGAGAUCGCAGAUG______G__ (((....)))............................((((......(((((....).))))((((((....)))))).....))))............ ( -7.11 = -8.13 + 1.03)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:57:02 2006