
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
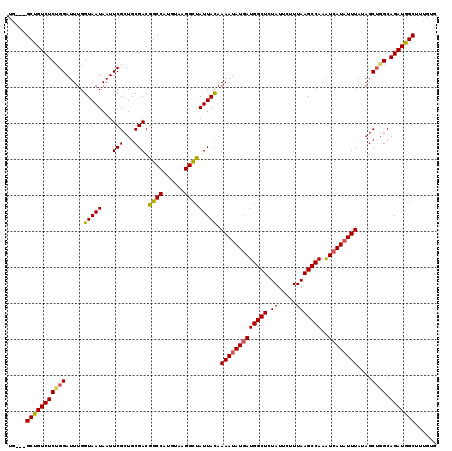
| Location | 14,453,083 – 14,453,239 |
| Length | 156 |
| Max. P | 0.972028 |

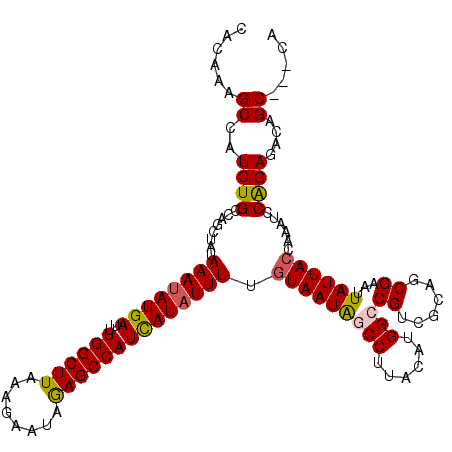
| Location | 14,453,083 – 14,453,199 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 87.81 |
| Mean single sequence MFE | -32.98 |
| Consensus MFE | -27.88 |
| Energy contribution | -27.82 |
| Covariance contribution | -0.06 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.07 |
| Structure conservation index | 0.85 |
| SVM decision value | 1.69 |
| SVM RNA-class probability | 0.972028 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14453083 116 + 27905053 UG---GCUGUCUCUGGAUUUGGUAAUAAUUCGCUGCGACGGCCAUGUAAGGCCAUUACAAAAUAUGAUGGCUCUAUUCUUUAAGCCAAAUCAUAUUUAUAGCUGGCAGAUGGCUUUGUG ((---((((((.(((((((.......))))))..).))))))))..((((((((((...(((((((((((((..(....)..)))))..))))))))...(....).)))))))))).. ( -36.90) >DroSec_CAF1 10274 119 + 1 UGGUGGCUGUCUCCGGAUUUGGUAAUAAUUCGCUACGACGGCCAUGUAAGGUUAUUACAAAAUAUGAUGGCUCUAUUCUUUAAGCCAAGUCAUAUUUAUAGCUGGCAGAUGGCUUUGUG ..(((((((((..((((((.......))))))....))))))))).((((((((((...(((((((((((((..(....)..)))))..))))))))...(....).)))))))))).. ( -35.30) >DroSim_CAF1 10851 119 + 1 UGGUGGCUGUCUCCGGAUUUGGUAAUAAUUCGCUGCGACGGCCAUGUAAGGUUAUUACAAAAUAUGAUGGCUCUAUUCUUUAAGCCAAAUCAUAUUUAUAGCUGGCAGAUGGCUUUGUG ..(((((((((.(((((((.......))))))..).))))))))).((((((((((...(((((((((((((..(....)..)))))..))))))))...(....).)))))))))).. ( -35.50) >DroEre_CAF1 10018 114 + 1 UG---GCUGUCUCUGGAUUUGGUAAUAAUUCGAUGCGACGGCCUUAUGAGGCCAUUAUAAAAUAUGAUGGCUCUAUUCUUUAAGCCAAAUCAUUUUU--AGCUUGCAGAUAGCUUUGGG .(---((((((..((((((.......)))))).(((((((((((....)))))....(((((.(((((((((..(....)..)))))..))))))))--)).))))))))))))..... ( -32.40) >DroYak_CAF1 11405 106 + 1 UA---GCUGUCUCUGGAUUUGGUAAUAAUUCGCUGCGACGG----------CUAUUACAAAAUAUAAUGGCUUUAUUCUUUAAGCCAAAUCAUAUUUAAAGCAGGCAGAUGGCUUUGUG .(---((((((((((......((((((....((((...)))----------))))))).((((((..((((((........))))))....))))))....)))..))))))))..... ( -24.80) >consensus UG___GCUGUCUCUGGAUUUGGUAAUAAUUCGCUGCGACGGCCAUGUAAGGCUAUUACAAAAUAUGAUGGCUCUAUUCUUUAAGCCAAAUCAUAUUUAUAGCUGGCAGAUGGCUUUGUG .....(((((((((((.....(((((...(((...))).((((......))))))))).(((((((((((((.((.....)))))))..))))))))....)))).)))))))...... (-27.88 = -27.82 + -0.06)


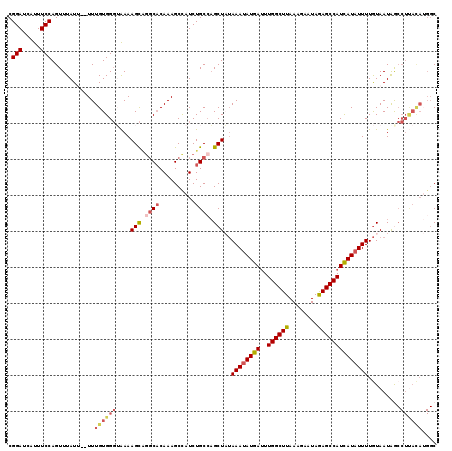
| Location | 14,453,083 – 14,453,199 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 87.81 |
| Mean single sequence MFE | -25.78 |
| Consensus MFE | -19.10 |
| Energy contribution | -19.20 |
| Covariance contribution | 0.10 |
| Combinations/Pair | 1.13 |
| Mean z-score | -2.02 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.66 |
| SVM RNA-class probability | 0.815177 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14453083 116 - 27905053 CACAAAGCCAUCUGCCAGCUAUAAAUAUGAUUUGGCUUAAAGAAUAGAGCCAUCAUAUUUUGUAAUGGCCUUACAUGGCCGUCGCAGCGAAUUAUUACCAAAUCCAGAGACAGC---CA ......((..((((........((((((((..((((((........))))))))))))))((..((((((......))))))..))..................))))....))---.. ( -30.90) >DroSec_CAF1 10274 119 - 1 CACAAAGCCAUCUGCCAGCUAUAAAUAUGACUUGGCUUAAAGAAUAGAGCCAUCAUAUUUUGUAAUAACCUUACAUGGCCGUCGUAGCGAAUUAUUACCAAAUCCGGAGACAGCCACCA ......((..(((.((.(((((((((((((..((((((........)))))))))))))).((((.....)))))))))(((....)))................)))))..))..... ( -25.60) >DroSim_CAF1 10851 119 - 1 CACAAAGCCAUCUGCCAGCUAUAAAUAUGAUUUGGCUUAAAGAAUAGAGCCAUCAUAUUUUGUAAUAACCUUACAUGGCCGUCGCAGCGAAUUAUUACCAAAUCCGGAGACAGCCACCA ......((..(((.((.(((((((((((((..((((((........)))))))))))))).((((.....)))))))))(((....)))................)))))..))..... ( -25.60) >DroEre_CAF1 10018 114 - 1 CCCAAAGCUAUCUGCAAGCU--AAAAAUGAUUUGGCUUAAAGAAUAGAGCCAUCAUAUUUUAUAAUGGCCUCAUAAGGCCGUCGCAUCGAAUUAUUACCAAAUCCAGAGACAGC---CA ......(((.((((.....(--((((((((..((((((........)))))))))).)))))..(((((((....)))))))......................))))...)))---.. ( -25.80) >DroYak_CAF1 11405 106 - 1 CACAAAGCCAUCUGCCUGCUUUAAAUAUGAUUUGGCUUAAAGAAUAAAGCCAUUAUAUUUUGUAAUAG----------CCGUCGCAGCGAAUUAUUACCAAAUCCAGAGACAGC---UA .....(((..((((........((((((((..((((((........)))))))))))))).(((((((----------.(((....)))..)))))))......))))....))---). ( -21.00) >consensus CACAAAGCCAUCUGCCAGCUAUAAAUAUGAUUUGGCUUAAAGAAUAGAGCCAUCAUAUUUUGUAAUAGCCUUACAUGGCCGUCGCAGCGAAUUAUUACCAAAUCCAGAGACAGC___CA ......((..((((........((((((((..((((((........)))))))))))))).(((((((((......)))((......))...))))))......))))....))..... (-19.10 = -19.20 + 0.10)


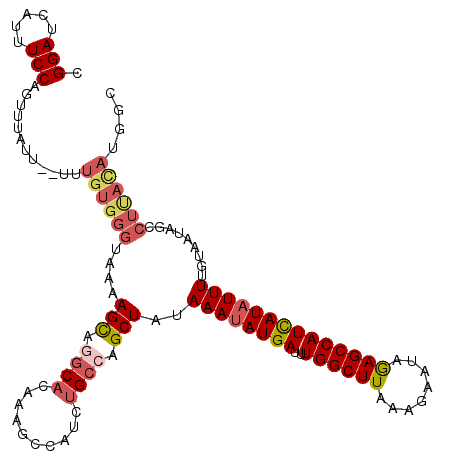
| Location | 14,453,120 – 14,453,239 |
|---|---|
| Length | 119 |
| Sequences | 5 |
| Columns | 119 |
| Reading direction | reverse |
| Mean pairwise identity | 86.84 |
| Mean single sequence MFE | -28.62 |
| Consensus MFE | -20.80 |
| Energy contribution | -22.28 |
| Covariance contribution | 1.48 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.73 |
| SVM decision value | 0.44 |
| SVM RNA-class probability | 0.736668 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14453120 119 - 27905053 CGGAUCAUUUCCAGUUUAUAUUUUUGUGGGUAAAAGCAGGCACAAAGCCAUCUGCCAGCUAUAAAUAUGAUUUGGCUUAAAGAAUAGAGCCAUCAUAUUUUGUAAUGGCCUUACAUGGC .(((.....)))..........((((((.((....))...))))))(((((..((((.....((((((((..((((((........)))))))))))))).....)))).....))))) ( -33.20) >DroSec_CAF1 10314 117 - 1 CGGAUCAUUUCCAGUUUAUA--UUUGUGGGUAAAAGCCGGCACAAAGCCAUCUGCCAGCUAUAAAUAUGACUUGGCUUAAAGAAUAGAGCCAUCAUAUUUUGUAAUAACCUUACAUGGC .(((.....)))........--..(((((((...(((.((((..........)))).)))..((((((((..((((((........)))))))))))))).......))).)))).... ( -29.50) >DroSim_CAF1 10891 117 - 1 CGGAUCAUUUCCAGUUUAUU--UUUGUGGGUAAAAGCAGGCACAAAGCCAUCUGCCAGCUAUAAAUAUGAUUUGGCUUAAAGAAUAGAGCCAUCAUAUUUUGUAAUAACCUUACAUGGC .(((.....)))........--..(((((((...(((.((((..........)))).)))..((((((((..((((((........)))))))))))))).......))).)))).... ( -28.80) >DroEre_CAF1 10055 115 - 1 CGGAUCAUUUCCAGUUUAUU--UUUGUGGGGAAAAGCAGUCCCAAAGCUAUCUGCAAGCU--AAAAAUGAUUUGGCUUAAAGAAUAGAGCCAUCAUAUUUUAUAAUGGCCUCAUAAGGC .(((.....))).......(--(((((((((....((((............))))....(--((((((((..((((((........)))))))))).)))))......)))))))))). ( -28.00) >DroYak_CAF1 11442 107 - 1 CGGAUCAUUUCCAGUUUAUU--UUUGUGGUGGAAAGUGUGCACAAAGCCAUCUGCCUGCUUUAAAUAUGAUUUGGCUUAAAGAAUAAAGCCAUUAUAUUUUGUAAUAG----------C .(((.....)))........--.....((..((...((.((.....))))))..))(((...((((((((..((((((........)))))))))))))).)))....----------. ( -23.60) >consensus CGGAUCAUUUCCAGUUUAUU__UUUGUGGGUAAAAGCAGGCACAAAGCCAUCUGCCAGCUAUAAAUAUGAUUUGGCUUAAAGAAUAGAGCCAUCAUAUUUUGUAAUAGCCUUACAUGGC .(((.....)))............((((((....(((.((((..........)))).)))..((((((((..((((((........)))))))))))))).........)))))).... (-20.80 = -22.28 + 1.48)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:55:56 2006