
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
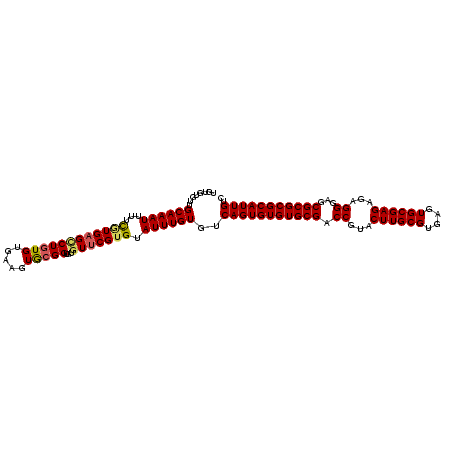
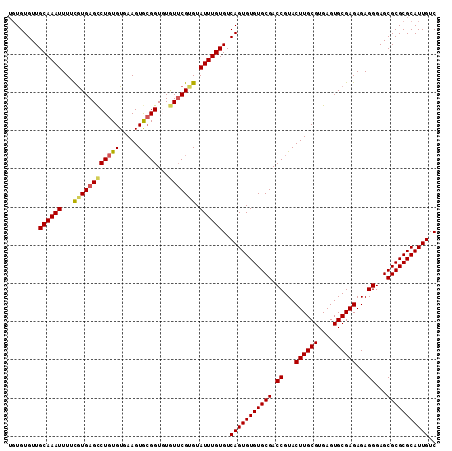
| Location | 14,022,920 – 14,023,064 |
| Length | 144 |
| Max. P | 0.921048 |

| Location | 14,022,920 – 14,023,031 |
|---|---|
| Length | 111 |
| Sequences | 5 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 95.89 |
| Mean single sequence MFE | -37.30 |
| Consensus MFE | -34.70 |
| Energy contribution | -34.94 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.11 |
| Mean z-score | -1.41 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.14 |
| SVM RNA-class probability | 0.921048 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14022920 111 + 27905053 UGUGUGUUGCAAAUUUUCGUGAGCCUGUGUGAAGUGCGGUGUGU-CGUGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUC ........((((((...((...((((((.......)))).))..-))...))))))..(((((((((((.((...((((((....))))))...))...))))))))))).. ( -37.40) >DroSec_CAF1 8015 110 + 1 --UGUGUUGCAAAUUUUCGUGAGUCUGUGUGAAGUGCGGUGUGUUCGUGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUC --......((((((...((.....((((.......))))......))...))))))..(((((((((((.((...((((((....))))))...))...))))))))))).. ( -34.90) >DroSim_CAF1 8028 112 + 1 UGUGUGUUGCAAAUUUUCAUGAGCCUGUGUGAAGUGCGGUGUGUUCGUGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUC ........((((((...(((((((((((.......))))...))))))).))))))..(((((((((((.((...((((((....))))))...))...))))))))))).. ( -40.70) >DroEre_CAF1 8006 112 + 1 UGUGUGUUGCAAAUUUUCGUGAGCCUGUGUGAAGUGCGGUCUGUUCGUGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUC .(((((((.(...(((((((...........((((((((((....((..((((......))))..)).))))))))))((....)))))))))..).)))))))........ ( -37.30) >DroYak_CAF1 7954 112 + 1 UGUGUGUUGCAAAUUUUUGUGAGCCUGUGUGAAGUAAGGUCUAUUCGGGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUC ........((((((.((((..(((((..........))).))...)))).))))))..(((((((((((.((...((((((....))))))...))...))))))))))).. ( -36.20) >consensus UGUGUGUUGCAAAUUUUCGUGAGCCUGUGUGAAGUGCGGUGUGUUCGUGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUC ........((((((...((((((((((((.....)))))...))))))).))))))..(((((((((((.((...((((((....))))))...))...))))))))))).. (-34.70 = -34.94 + 0.24)

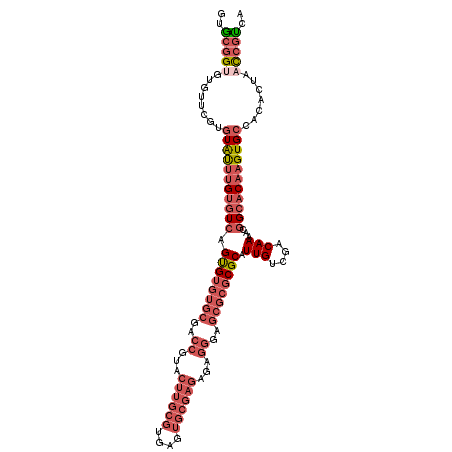
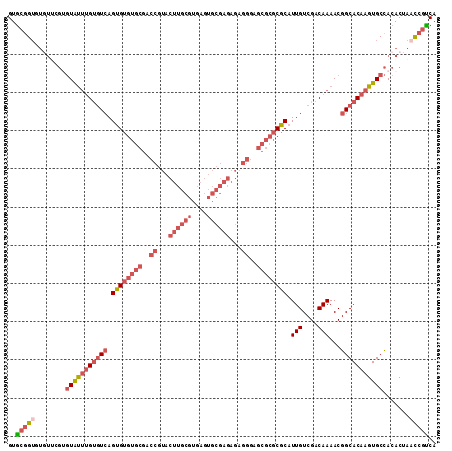
| Location | 14,022,953 – 14,023,064 |
|---|---|
| Length | 111 |
| Sequences | 6 |
| Columns | 112 |
| Reading direction | forward |
| Mean pairwise identity | 84.75 |
| Mean single sequence MFE | -45.69 |
| Consensus MFE | -32.86 |
| Energy contribution | -35.86 |
| Covariance contribution | 3.00 |
| Combinations/Pair | 1.17 |
| Mean z-score | -2.76 |
| Structure conservation index | 0.72 |
| SVM decision value | 0.37 |
| SVM RNA-class probability | 0.709240 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14022953 111 + 27905053 GUGCGGUGUGU-CGUGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUCGACAAAACGGCACAAGUGCCACACUAACCGUCA ..(((((((((-.(.(((((((((((.((((((((..((...((((((....))))))...))..)))))))).(((....)))...))))))))))))))))..))))).. ( -50.80) >DroVir_CAF1 9082 84 + 1 GUGCGG---------CUGCG-GCUUGAGCG------------------CGUGUGAGAGAGAGGGAGCGCGCGCAUUGUCGACAAAACGGCACAAGUGCUACACUGUUAGCCA ....((---------(((((-(((((.(((------------------(((((............))))))))..(((((......))))))))).......))).))))). ( -29.91) >DroSec_CAF1 8046 112 + 1 GUGCGGUGUGUUCGUGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUCGACAAAACGGCACAAGUGCCACACUAACCGUCA ..(((((((((..(.(((((((((((.((((((((..((...((((((....))))))...))..)))))))).(((....)))...))))))))))))))))..))))).. ( -49.20) >DroSim_CAF1 8061 112 + 1 GUGCGGUGUGUUCGUGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUCGACAAAACGGCACAAGUGCCACACUACCCGUCA ..((((.((((..(.(((((((((((.((((((((..((...((((((....))))))...))..)))))))).(((....)))...))))))))))))))))...)))).. ( -48.30) >DroEre_CAF1 8039 112 + 1 GUGCGGUCUGUUCGUGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUCGACAAAACGGCACAAGUGCCACACUAACCGUCA ..(((((..((..(((((((((((((.((((((((..((...((((((....))))))...))..)))))))).(((....)))...)))))))))).)))))..))))).. ( -48.50) >DroYak_CAF1 7987 112 + 1 GUAAGGUCUAUUCGGGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUCGACAAAACGGCACAAGUGCCACACUAACCGUCA ....(((.......((((((((((((.((((((((..((...((((((....))))))...))..)))))))).(((....)))...))))))))))))......))).... ( -47.42) >consensus GUGCGGUGUGUUCGUGUAUUUGUGUCAGUGUGUGCGACCGUACUUGCGUGAGUGCGAGAGAGGGAGCGCGCGCAUUGUCGACAAAACGGCACAAGUGCCACACUAACCGUCA ..(((((........(((((((((((.((((((((..((...((((((....))))))...))..)))))))).(((....)))...))))))))))).......))))).. (-32.86 = -35.86 + 3.00)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:52:32 2006