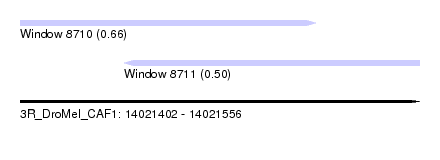
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 14,021,402 – 14,021,556 |
| Length | 154 |
| Max. P | 0.664088 |

| Location | 14,021,402 – 14,021,516 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 78.31 |
| Mean single sequence MFE | -55.83 |
| Consensus MFE | -33.69 |
| Energy contribution | -34.30 |
| Covariance contribution | 0.62 |
| Combinations/Pair | 1.51 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.60 |
| SVM decision value | 0.28 |
| SVM RNA-class probability | 0.664088 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
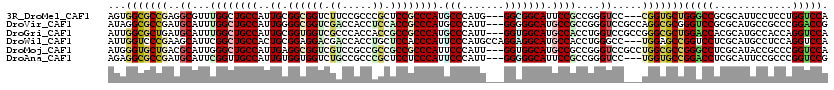
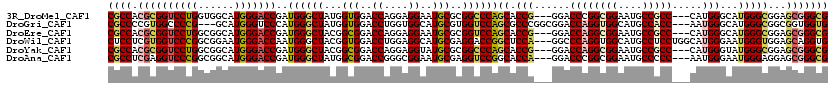
>3R_DroMel_CAF1 14021402 114 + 27905053 AGUGGCGCCGAGGCGUUUGGCUGCCAUUGCGGCGGUCUUCCGCCCGCUCCGCCCAUGCCCAUG---GGCGGCAUUCCGCCGGGUCC---CGGUGCUGGGCCGCGCAUUCCUCCUGGUCCA ...(((((((.((.(..((((((((...(((((((....))).))))...((((((....)))---)))))))....))))..)))---)))))))((((((...........)))))). ( -56.90) >DroVir_CAF1 7546 117 + 1 AUAGGCGCCGAUGCAUUUGGCUGCCAUUGGGGCGGUCGACCACCUCCACCGCCCAUGCCCAUU---GGGGGCAUGCCGCCGGGUCCGCCAGGCGCGGGUCCGCGCAUGCCGCCCGGACCG ...((((((((.....))))).)))....(((((((.((.....)).))))))).......((---((((((((((.((.((..((((.....))))..))))))))))).))))).... ( -64.00) >DroGri_CAF1 8615 117 + 1 AUUGGCGCUGAUGCAUUUGGCUGCCAUUGCGGUGGUCGCCCACCACCGCCGCCCAUGCCCAUU---GGUGGCAUGCCACCUGGUCCGCCGGGCGCUGGACCACGCAUGCCACCAGGUCCA ..((((((..(.....)..)).))))..((((((((.....)))))))).......(((...(---((((((((((....((((((((.....)).)))))).))))))))))))))... ( -57.90) >DroWil_CAF1 7641 117 + 1 AUUGGUCCCGAAGCAUUCGGCUGCCACUGCGGAGGACGACCACCUGCUCCACCCAUUCCCAUGCCAGGAGGCAUGCCACCUGGGCC---UGGAGCCGGUCCUCGCAUGCCUCCAGGUCCA ...(((.((((.....))))..)))(((..(((((.(((..(((.((((((((((....((((((....)))))).....))))..---)))))).)))..)))....))))).)))... ( -49.40) >DroMoj_CAF1 8913 117 + 1 AUGGGUGCUGACGCAUUGGGCUGCCAUUGAGGCGGUCGUCCGCCGCCGCCGCCCAUUCCCAUU---GGUGGCAUGCCGCCGGGUCCGCCUGGCGCCGGGCCUCGCAUACCGCCCGGUCCA .(((((((....))))..(((((((((..((((((....)))))((....)).........).---.)))))).)))((((((....))))))(((((((..........)))))))))) ( -56.40) >DroAna_CAF1 6334 114 + 1 AGAGGCGCCGAUGCAUUCGGUUGCCAUUGUGGUGGUCUGCCGCCCGCUCCUCCCAUUCCCAUU---GGGGGCAUUCCGCCGGGUCC---UGGUGCCGGACCUCGCAUUCCGCCCGGUCCG ...(((((((..(.(((((((((((...(((((((....))).))))....((((.......)---)))))))....))))))).)---)))))))(((((..((.....))..))))). ( -50.40) >consensus AUUGGCGCCGAUGCAUUUGGCUGCCAUUGCGGCGGUCGACCACCCCCUCCGCCCAUGCCCAUU___GGGGGCAUGCCGCCGGGUCC___UGGCGCCGGACCUCGCAUGCCGCCCGGUCCA ...(((((((..((...((((((((..((.((((((.((.....)).)))))))).(((.......))))))).))))....)).....)))))))(((((.............))))). (-33.69 = -34.30 + 0.62)
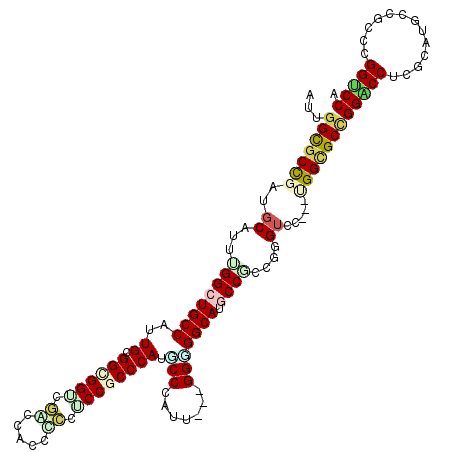
| Location | 14,021,442 – 14,021,556 |
|---|---|
| Length | 114 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 82.53 |
| Mean single sequence MFE | -60.90 |
| Consensus MFE | -38.89 |
| Energy contribution | -39.45 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.70 |
| Structure conservation index | 0.64 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 14021442 114 - 27905053 CGCCACGCGGUCCUGGUGGCAUGGGACCGAUGGGCUAUGGUGGACCAGGAGGAAUGCGCGGCCCAGCACCG---GGACCCGGCGGAAUGCCGCC---CAUGGGCAUGGGCGGAGCGGGCG ((((.(((((((((((((.(.((((.(((.....((.(((....)))...))......)))))))))))))---)))))..........(((((---(((....)))))))).))))))) ( -62.70) >DroGri_CAF1 8655 114 - 1 CGCCCCGUGGCCCCG---GCAUGGGUCCCAUGGGCUAUGGUGGACCUGGUGGCAUGCGUGGUCCAGCGCCCGGCGGACCAGGUGGCAUGCCACC---AAUGGGCAUGGGCGGCGGUGGUG ((((((((.((((..---((..((((((.((.(....).))))))))((((((((((.((((((.((.....))))))))....))))))))))---.....))..)))).)))).)))) ( -66.20) >DroEre_CAF1 6547 114 - 1 CGCCACGCGGUCCUGGCGGCAUGGGACCGAUGGGCUACGGCGGACCAGGAGGAAUGCGCGGUCCAGCACCG---GGACCAGGCGGAAUGCCGCC---CAUGGGCAUGGGCGGAGCGGGCG ((((.(((((((((((..((...((((((.((.((....))...((....))....)))))))).)).)))---)))))..........(((((---(((....)))))))).))))))) ( -62.90) >DroWil_CAF1 7681 117 - 1 CUCCUCGUGGUCCCGGCGGAAUGGGACCAAUGGGCUACGGUGGACCUGGAGGCAUGCGAGGACCGGCUCCA---GGCCCAGGUGGCAUGCCUCCUGGCAUGGGAAUGGGUGGAGCAGGUG ..(((((((((((((......))))))).....(((.(((.....)))..)))..))))))(((.(((((.---.(((((.....((((((....))))))....)))))))))).))). ( -57.80) >DroYak_CAF1 6504 114 - 1 CGCCACGCGGUCCUGGCGGCAUGGGACCGAUGGGCUACGGCGGACCAGGAGGUAUGCGCGGCCCAGCACCG---GGACCAGGCGGAAUGCCGCC---CAUGGGUAUGGGCGGAGCGGGCG ((((.(((((((((((..((.((((.(((.((.((....))..(((....)))...))))))))))).)))---)))))..........(((((---(((....)))))))).))))))) ( -63.40) >DroAna_CAF1 6374 114 - 1 CGCCUCGAGGUCCCGGCGGCAUGGGACCGAUGGGCUAUGGCGGACCGGGCGGAAUGCGAGGUCCGGCACCA---GGACCCGGCGGAAUGCCCCC---AAUGGGAAUGGGAGGAGCGGGCG ((((.((...(((((..(((((....(((.((((((.((.((((((..((.....))..)))))).))...---)).)))).))).)))))(((---...)))..)))))....)))))) ( -52.40) >consensus CGCCACGCGGUCCCGGCGGCAUGGGACCGAUGGGCUACGGCGGACCAGGAGGAAUGCGCGGUCCAGCACCG___GGACCAGGCGGAAUGCCGCC___CAUGGGCAUGGGCGGAGCGGGCG ((((.((((((((((......)))))))..((((((...(((..((....))..)))..))))))((.(((......((((((((....))))).....)))...)))))...))))))) (-38.89 = -39.45 + 0.56)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:52:28 2006