
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 13,957,495 – 13,957,652 |
| Length | 157 |
| Max. P | 0.992379 |

| Location | 13,957,495 – 13,957,612 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | forward |
| Mean pairwise identity | 70.59 |
| Mean single sequence MFE | -48.44 |
| Consensus MFE | -25.62 |
| Energy contribution | -28.80 |
| Covariance contribution | 3.18 |
| Combinations/Pair | 1.17 |
| Mean z-score | -1.61 |
| Structure conservation index | 0.53 |
| SVM decision value | 0.19 |
| SVM RNA-class probability | 0.625666 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
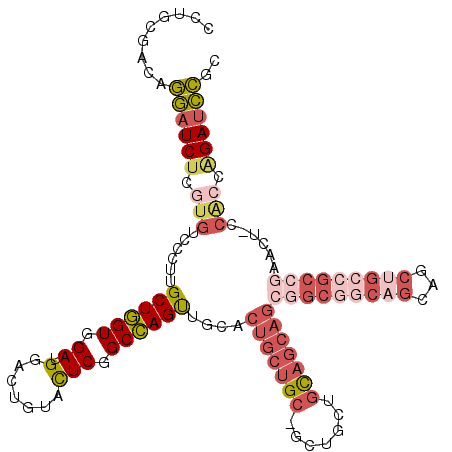
>3R_DroMel_CAF1 13957495 117 + 27905053 CCCGAUAUGCAGCCCAGCUCGAAUGGCACCGGCUAUUCCGGCGGAUCUGGUGGCAGUUCGGCGGCAGCUGCUGCCGCCGCUGCUGCAGCAGCAGCAGCAGUGCAACUGGCCGAGUAC .........(((.((.(((.(((((((....))))))).)))))..))).(((((((((((((((((...))))))))((((((((....))))))))...).)))).))))..... ( -57.10) >DroSec_CAF1 228383 114 + 1 CCCGACAUGCAGCCCAGCUCGAAUGGCACGGGCUAUUCCGGCGGAUCUGGUGGCAGUUCGGCGGCAGCUGCUGCCGCCGCUGCUGCAGCA---GCAGCAGUGCAACUGGCCGAGUAC ....((...(((.((.(((.(((((((....))))))).)))))..))).(((((((((((((((((...))))))))((((((((....---))))))))).)))).)))).)).. ( -54.80) >DroSim_CAF1 236829 114 + 1 CCCGACAUGCAGCCCAGCUCGAAUGGCACGGGCUAUUCCGGCGGAUCCGGUGGGAGUUCGGCGGCAGCUGCUGCCGCCGCUGCUGCAGCA---GCAGCAGUGCAACUGGCCGAGUAC .......(((...((.(((.(((((((....))))))).)))))...((((.((.....((((((((...))))))))((((((((....---))))))))....)).)))).))). ( -55.00) >DroWil_CAF1 202388 96 + 1 CCAG------AGCCCAGUUCAAAUGGCACCGGUUACUCAGUGACAUCUGGCGC--------CGGGAGCAGUGG----CAAUGCUAAU---GCUACAGCAGUGCAACUAGCUGAAUAU .(((------.((((.((((...((((.((.(((((...)))))....)).))--------)).)))).).))----)..(((...(---((....)))..))).....)))..... ( -30.70) >DroMoj_CAF1 314596 96 + 1 ACGGACAUGCAGCCCAGCUCGAACGGCACAGGCUACUCCAGCGCCUC---------------GGCUGCUGCGG---CUGC---CGCAGCCGCUGCUGCCGUCCAGCUGGCCGAGUAC .(((......((((..(((.....)))...))))...(((((....(---------------(((.((.((((---(((.---..))))))).)).))))....))))))))..... ( -44.60) >consensus CCCGACAUGCAGCCCAGCUCGAAUGGCACCGGCUAUUCCGGCGGAUCUGGUGG_AGUUCGGCGGCAGCUGCUGCCGCCGCUGCUGCAGCAGC_GCAGCAGUGCAACUGGCCGAGUAC ................(((((((((((....))))))).(((................(((((((....))))))).(((((((((.......)))))))))......))))))).. (-25.62 = -28.80 + 3.18)



| Location | 13,957,495 – 13,957,612 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 70.59 |
| Mean single sequence MFE | -52.36 |
| Consensus MFE | -23.90 |
| Energy contribution | -26.82 |
| Covariance contribution | 2.92 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.34 |
| Structure conservation index | 0.46 |
| SVM decision value | 2.32 |
| SVM RNA-class probability | 0.992379 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 13957495 117 - 27905053 GUACUCGGCCAGUUGCACUGCUGCUGCUGCUGCAGCAGCGGCGGCAGCAGCUGCCGCCGAACUGCCACCAGAUCCGCCGGAAUAGCCGGUGCCAUUCGAGCUGGGCUGCAUAUCGGG .....((((.((((((..((((((((((((....))))))))))))))))))))))((((..(((..((((.((((((((.....))))))......)).))))...)))..)))). ( -62.10) >DroSec_CAF1 228383 114 - 1 GUACUCGGCCAGUUGCACUGCUGC---UGCUGCAGCAGCGGCGGCAGCAGCUGCCGCCGAACUGCCACCAGAUCCGCCGGAAUAGCCCGUGCCAUUCGAGCUGGGCUGCAUGUCGGG ......(((.((((...(((((((---....)))))))(((((((((...)))))))))))))))).......(((.((...(((((((.(((....).)))))))))..)).))). ( -55.80) >DroSim_CAF1 236829 114 - 1 GUACUCGGCCAGUUGCACUGCUGC---UGCUGCAGCAGCGGCGGCAGCAGCUGCCGCCGAACUCCCACCGGAUCCGCCGGAAUAGCCCGUGCCAUUCGAGCUGGGCUGCAUGUCGGG ....((((((((((((.(((((((---(((....))))))))))..)))))))..)))))...(((.((((.....))))..(((((((.(((....).)))))))))......))) ( -59.60) >DroWil_CAF1 202388 96 - 1 AUAUUCAGCUAGUUGCACUGCUGUAGC---AUUAGCAUUG----CCACUGCUCCCG--------GCGCCAGAUGUCACUGAGUAACCGGUGCCAUUUGAACUGGGCU------CUGG ......(((.(((.(((.(((((....---..))))).))----).))))))((((--------(...((((((.(((((......))))).))))))..)))))..------.... ( -34.00) >DroMoj_CAF1 314596 96 - 1 GUACUCGGCCAGCUGGACGGCAGCAGCGGCUGCG---GCAG---CCGCAGCAGCC---------------GAGGCGCUGGAGUAGCCUGUGCCGUUCGAGCUGGGCUGCAUGUCCGU (.((.(((((((((((((((((((..(((((((.---((..---..)).))))))---------------)..))((((...))))...)))))))).)))).)))))...)).).. ( -50.30) >consensus GUACUCGGCCAGUUGCACUGCUGC_GCUGCUGCAGCAGCGGCGGCAGCAGCUGCCGCCGAACU_CCACCAGAUCCGCCGGAAUAGCCCGUGCCAUUCGAGCUGGGCUGCAUGUCGGG (((((((((((((.((((((((((.......))))))((((((((....))))))))...............................)))).))).).)))))).)))........ (-23.90 = -26.82 + 2.92)



| Location | 13,957,535 – 13,957,652 |
|---|---|
| Length | 117 |
| Sequences | 5 |
| Columns | 117 |
| Reading direction | reverse |
| Mean pairwise identity | 73.36 |
| Mean single sequence MFE | -47.40 |
| Consensus MFE | -29.38 |
| Energy contribution | -32.84 |
| Covariance contribution | 3.46 |
| Combinations/Pair | 1.24 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.62 |
| SVM decision value | 0.53 |
| SVM RNA-class probability | 0.770736 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 13957535 117 - 27905053 CCUGCGACAGGAUCUCGUGUCCCUUGCUGGUGGAUGGACUGUACUCGGCCAGUUGCACUGCUGCUGCUGCUGCAGCAGCGGCGGCAGCAGCUGCCGCCGAACUGCCACCAGAUCCGC ...((((((.(....).)))).....(((((((.......((..((((((((((((..((((((((((((....)))))))))))))))))))..)))))))..)))))))....)) ( -59.00) >DroSec_CAF1 228423 114 - 1 CCUGCGACAGGAUCUCGUGUCCCUUGCUGGUGGAUGGACUGUACUCGGCCAGUUGCACUGCUGC---UGCUGCAGCAGCGGCGGCAGCAGCUGCCGCCGAACUGCCACCAGAUCCGC .........((((((.(((......((((((.((.(.......))).)))))).((((((((((---....)))))))(((((((((...)))))))))...)))))).)))))).. ( -55.50) >DroSim_CAF1 236869 114 - 1 CCUGCGACAGGAUCUCGUGUCCCUUGCUGGUGGAUGGACUGUACUCGGCCAGUUGCACUGCUGC---UGCUGCAGCAGCGGCGGCAGCAGCUGCCGCCGAACUCCCACCGGAUCCGC ...((((((.(....).)))).....(((((((.(((.(((....))))))((.((((((((((---((((((....))))))))))))).))).)).......)))))))....)) ( -54.80) >DroWil_CAF1 202422 102 - 1 CUUGUGACAAUAUCUCAUGACCCUUGCUAGUAGAAGGACUAUAUUCAGCUAGUUGCACUGCUGUAGC---AUUAGCAUUG----CCACUGCUCCCG--------GCGCCAGAUGUCA ....(((((...(((..((.((...((((((.(((........))).)))))).(((.(((((....---..))))).))----)..........)--------)))..)))))))) ( -24.00) >DroMoj_CAF1 314636 96 - 1 CCUGCGACAGUAUCUCGUGGCCCUUGCUGGUGGACGGACUGUACUCGGCCAGCUGGACGGCAGCAGCGGCUGCG---GCAG---CCGCAGCAGCC---------------GAGGCGC ...((((.......)))).(((((.((((((.((.(.......))).)))))).)).((((.((.(((((((..---.)))---)))).)).)))---------------).))).. ( -43.70) >consensus CCUGCGACAGGAUCUCGUGUCCCUUGCUGGUGGAUGGACUGUACUCGGCCAGUUGCACUGCUGC_GCUGCUGCAGCAGCGGCGGCAGCAGCUGCCGCCGAACU_CCACCAGAUCCGC .........((((((.(((......((((((.((.(.......))).))))))....(((((((.......)))))))(((((((((...)))))))))......))).)))))).. (-29.38 = -32.84 + 3.46)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:52:02 2006