
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 13,710,166 – 13,710,274 |
| Length | 108 |
| Max. P | 0.740774 |

| Location | 13,710,166 – 13,710,274 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 82.48 |
| Mean single sequence MFE | -49.67 |
| Consensus MFE | -33.34 |
| Energy contribution | -34.48 |
| Covariance contribution | 1.14 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.23 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.45 |
| SVM RNA-class probability | 0.740774 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
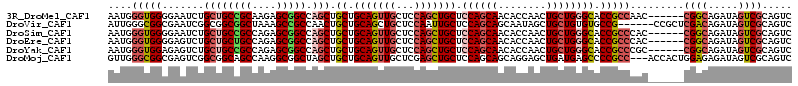
>3R_DroMel_CAF1 13710166 108 + 27905053 AAUGGGUGGGGAAUCUGCUGCCGCAAGAGCGGCCAGCUGCUGCAGUUGCUCCAGCUGCUCCAGCAACACCAACUGCUGGGCACCGCCAAC------CGGCAGAUAGUCGCAGUC ....(((((.......((((((((....)))).))))(((.(((((((...))))))).((((((........))))))))))))))...------((((.....))))..... ( -45.60) >DroVir_CAF1 8761 108 + 1 AUUGGGCGGCGAAUCGGCGGCGGCUAAAGCCGCCAACUGCUGCAGCUGCUCCAAUUGCUCCAGCAGCAAUAGCUGCUGUGUGCCG------CCGCUCGACAGAUAGUCGCAGUC ....(((((((...((((((((((....))))))..((((((.(((..........))).))))))........))))..)))))------))(((((((.....)))).))). ( -54.20) >DroSim_CAF1 21743 108 + 1 AAUGGGUGGGGAAUCUGCUGCCGCCAGAGCGGCCAGCUGCUGCAGUUGCUCCAGCUGCUCCAGCAACACCAACUGCUGGGCACCGCCCAC------CGGCAGAUAGUCGCAGUC ..(((((((.......((((((((....)))).))))(((.(((((((...))))))).((((((........)))))))))))))))).------((((.....))))..... ( -49.10) >DroEre_CAF1 20542 108 + 1 AAUGGGUGGGGAGUCUGCUGCUGCCAGAGCGGCCAGCUGCUGCAGUUGCUCCAGCUGCUCCAGCAACACCAACUGCUGGGCACCGCCCAC------CGGCAGAUAGUCGCAGUC ..((((((((((((((((.((((((.....)).))))....))))..)))))...(((.((((((........)))))))))))))))).------((((.....))))..... ( -50.20) >DroYak_CAF1 28764 108 + 1 AAUGGGUGGAGAGUCUGCUGCCGCCAGAGCGGCCAGCUGCUGCAGUUGCUCCAGCUGCUCCAGCAACACCAACUGCUGGGCACCGCCCGC------CGGCAGAUAGUCGCAGUC ...((((((.......((((((((....)))).))))(((.(((((((...))))))).((((((........)))))))))))))))((------.(((.....))))).... ( -49.10) >DroMoj_CAF1 10481 111 + 1 GUUGGGCGGCGAGUCGGCGGCAGCCAAGGCGGCUAGCUGCUGCAGUUGCUCGAGCUGCUCCAGCAGCAGGAGCUGAUGAGCCCCGCC---ACCACUGGAGAGAUAGUCGCAGUC .....(((((..((((((....)))..((.(((..(((((((.(((.((....)).))).))))))).((.(((....))).)))))---.))........))).))))).... ( -49.80) >consensus AAUGGGUGGGGAAUCUGCUGCCGCCAGAGCGGCCAGCUGCUGCAGUUGCUCCAGCUGCUCCAGCAACACCAACUGCUGGGCACCGCCCAC______CGGCAGAUAGUCGCAGUC ....(((((.......((((((((....))))).))).((.(((((((...))))))).((((((........)))))))).))))).........((((.....))))..... (-33.34 = -34.48 + 1.14)

| Location | 13,710,166 – 13,710,274 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 82.48 |
| Mean single sequence MFE | -49.95 |
| Consensus MFE | -34.38 |
| Energy contribution | -34.97 |
| Covariance contribution | 0.59 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.94 |
| Structure conservation index | 0.69 |
| SVM decision value | 0.32 |
| SVM RNA-class probability | 0.685768 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
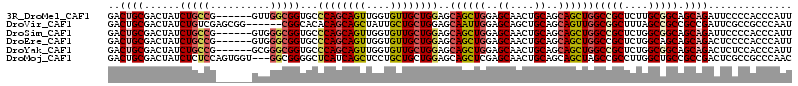
>3R_DroMel_CAF1 13710166 108 - 27905053 GACUGCGACUAUCUGCCG------GUUGGCGGUGCCCAGCAGUUGGUGUUGCUGGAGCAGCUGGAGCAACUGCAGCAGCUGGCCGCUCUUGCGGCAGCAGAUUCCCCACCCAUU ((((((......(((((.------...)))))......))))))((((.(((((.((..((....))..)).)))))((((.((((....))))))))........)))).... ( -47.10) >DroVir_CAF1 8761 108 - 1 GACUGCGACUAUCUGUCGAGCGG------CGGCACACAGCAGCUAUUGCUGCUGGAGCAAUUGGAGCAGCUGCAGCAGUUGGCGGCUUUAGCCGCCGCCGAUUCGCCGCCCAAU (.((.((((.....)))))))((------((((...(((((((....)))))))..(.((((((...(((((...)))))((((((....)))))).))))))))))))).... ( -54.00) >DroSim_CAF1 21743 108 - 1 GACUGCGACUAUCUGCCG------GUGGGCGGUGCCCAGCAGUUGGUGUUGCUGGAGCAGCUGGAGCAACUGCAGCAGCUGGCCGCUCUGGCGGCAGCAGAUUCCCCACCCAUU ..((((......((((((------(..(((((((((((((((......))))))).))(((((..((....))..))))).)))))))))))))..)))).............. ( -48.10) >DroEre_CAF1 20542 108 - 1 GACUGCGACUAUCUGCCG------GUGGGCGGUGCCCAGCAGUUGGUGUUGCUGGAGCAGCUGGAGCAACUGCAGCAGCUGGCCGCUCUGGCAGCAGCAGACUCCCCACCCAUU ..((((......((((((------(..(((((((((((((((......))))))).))(((((..((....))..))))).)))))))))))))..)))).............. ( -48.80) >DroYak_CAF1 28764 108 - 1 GACUGCGACUAUCUGCCG------GCGGGCGGUGCCCAGCAGUUGGUGUUGCUGGAGCAGCUGGAGCAACUGCAGCAGCUGGCCGCUCUGGCGGCAGCAGACUCUCCACCCAUU ..(((((.....)(((((------.(((((((((((((((((......))))))).))(((((..((....))..))))).))))).))).))))))))).............. ( -50.80) >DroMoj_CAF1 10481 111 - 1 GACUGCGACUAUCUCUCCAGUGGU---GGCGGGGCUCAUCAGCUCCUGCUGCUGGAGCAGCUCGAGCAACUGCAGCAGCUAGCCGCCUUGGCUGCCGCCGACUCGCCGCCCAAC .((((.((.......))))))(((---((((((........((((..(((((....)))))..))))..(.((.((((((((.....)))))))).)).).))))))))).... ( -50.90) >consensus GACUGCGACUAUCUGCCG______GUGGGCGGUGCCCAGCAGUUGGUGUUGCUGGAGCAGCUGGAGCAACUGCAGCAGCUGGCCGCUCUGGCGGCAGCAGACUCCCCACCCAUU ..((((......(((((..........)))))...((((((((....))))))))..((((((..((....))..))))))(((((....))))).)))).............. (-34.38 = -34.97 + 0.59)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:50:25 2006