
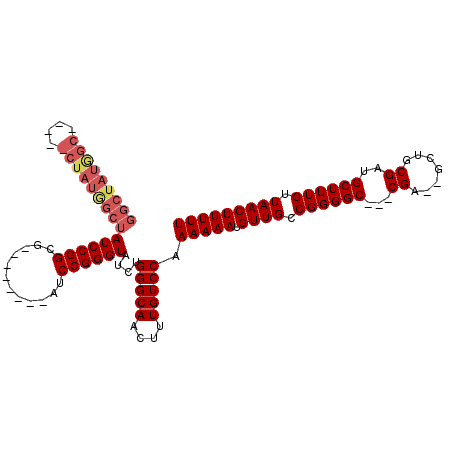
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 13,193,071 – 13,193,200 |
| Length | 129 |
| Max. P | 0.998871 |

| Location | 13,193,071 – 13,193,166 |
|---|---|
| Length | 95 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 84.74 |
| Mean single sequence MFE | -38.58 |
| Consensus MFE | -25.12 |
| Energy contribution | -26.78 |
| Covariance contribution | 1.66 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.72 |
| Structure conservation index | 0.65 |
| SVM decision value | 1.15 |
| SVM RNA-class probability | 0.922351 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 13193071 95 - 27905053 GGCUAUGG------CUAUGGCUAUCCGGCG--------AUCCGGGUAUCUGGGCAACUUUGUCCAAAAAAUGUUGCCGGGGC---CGA--GCUGCGAUGCUUUGUUAACUUUUU ((((.(((------((.(((((((((((..--------..)))))))..((((((....)))))).........)))).)))---)))--)))((((....))))......... ( -33.90) >DroSec_CAF1 220984 104 - 1 GGCUAUGGCUAUGGCUAUGGCUAUCCGGCG--------AUCCGGGUAUCUGGGCAACUUUGUCCAAAAAAUGUUGCCGGGGCCGCCGA--GCUGCGAUGCUUUGUUAACUUUUU (((((((((....)))))))))...(((((--------..((.((((.(((((((....))))))......).)))).))..)))))(--((......)))............. ( -41.00) >DroSim_CAF1 207042 98 - 1 GGCUAUGG------CUAUGGCUAUCCGGCG--------AUCCGGGUAUCUGGGCAACUUUGUCCAAAAAAUGUUGCCGGGGCCGCCGA--GCUGCGAUGCUUUGUUAACUUUUU (((...((------((.(((((((((((..--------..)))))))..((((((....)))))).........)))).)))))))((--((......))))............ ( -35.20) >DroEre_CAF1 211724 109 - 1 GGCUAUAGCUAUAGCUAUAGCUAUCCGGCGAUCCGGGGAUCCGGGUUGCCGGGCAACUUUGUCCAAAAAAUGUUGCCGGGGC---CGA--GCGGCGAUGCUUUGUUAACUUUUU (((((((((....)))))))))..((((((((((((....)))))))))).(((((((((.......))).))))))))(((---.((--(((....))))).)))........ ( -49.30) >DroYak_CAF1 220769 91 - 1 UGCUAUAGC------------UAUCCGGCG--------AUCCGGGUAGCUGGGCAACUUUGUCCAAAAAAUGUUGCCGGGGC---CGAAAGCAACGAUGCUUUGUUAACUUUUU .(((.((((------------(((((((..--------..)))))))))))(((((((((.......))).))))))..)))---..((((((....))))))........... ( -33.50) >consensus GGCUAUGGC_____CUAUGGCUAUCCGGCG________AUCCGGGUAUCUGGGCAACUUUGUCCAAAAAAUGUUGCCGGGGC___CGA__GCUGCGAUGCUUUGUUAACUUUUU (((((((((....)))))))))((((((............))))))....(((((....))))).(((((.((((.((((((...((.......))..)))))).))))))))) (-25.12 = -26.78 + 1.66)


| Location | 13,193,100 – 13,193,200 |
|---|---|
| Length | 100 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | forward |
| Mean pairwise identity | 87.73 |
| Mean single sequence MFE | -37.71 |
| Consensus MFE | -24.42 |
| Energy contribution | -24.86 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.92 |
| Structure conservation index | 0.65 |
| SVM decision value | 0.61 |
| SVM RNA-class probability | 0.799967 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 13193100 100 + 27905053 CCCGGCAACAUUUUUUGGACAAAGUUGCCCAGAUACCCGGAU--------CGCCGGAUAGCCAUAG------CCAUAGCCAUGGCCGUGCAGUCCGGCCGUAUAUCGCCAUAUA ...(((.........(((.((....)).)))((((..((...--------.(((((((.((.(..(------((((....)))))..))).)))))))))..)))))))..... ( -35.20) >DroSec_CAF1 221016 106 + 1 CCCGGCAACAUUUUUUGGACAAAGUUGCCCAGAUACCCGGAU--------CGCCGGAUAGCCAUAGCCAUAGCCAUAGCCAUGGCCGUGCAGUCCGGCCGUAUAUCGCCAUAUA ...(((.........(((.((....)).)))((((..((...--------.(((((((.((.(..(((((.((....)).)))))..))).)))))))))..)))))))..... ( -38.20) >DroSim_CAF1 207074 100 + 1 CCCGGCAACAUUUUUUGGACAAAGUUGCCCAGAUACCCGGAU--------CGCCGGAUAGCCAUAG------CCAUAGCCAUGGCCGUGCAGUCCGGCCGUAUAUCGCCAUAUA ...(((.........(((.((....)).)))((((..((...--------.(((((((.((.(..(------((((....)))))..))).)))))))))..)))))))..... ( -35.20) >DroEre_CAF1 211753 114 + 1 CCCGGCAACAUUUUUUGGACAAAGUUGCCCGGCAACCCGGAUCCCCGGAUCGCCGGAUAGCUAUAGCUAUAGCUAUAGCCAUGGCCGUGCAGUUCGGCCGUAUAUCGCCAUAUA ...((((((..............))))))((((...((((....))))...))))((((((((((((....)))))))).(((((((.......))))))).))))........ ( -46.84) >DroYak_CAF1 220800 94 + 1 CCCGGCAACAUUUUUUGGACAAAGUUGCCCAGCUACCCGGAU--------CGCCGGAUA------------GCUAUAGCAAUGGCCGUGCACUCCGGCCGUAUAUCGCCAUAUA ...(((.................(((((..(((((.((((..--------..)))).))------------)))...)))))(((((.......))))).......)))..... ( -33.10) >consensus CCCGGCAACAUUUUUUGGACAAAGUUGCCCAGAUACCCGGAU________CGCCGGAUAGCCAUAG_____GCCAUAGCCAUGGCCGUGCAGUCCGGCCGUAUAUCGCCAUAUA ...(((.........(((.((....)).)))((((.((((............))))........................(((((((.......))))))).)))))))..... (-24.42 = -24.86 + 0.44)


| Location | 13,193,100 – 13,193,200 |
|---|---|
| Length | 100 |
| Sequences | 5 |
| Columns | 114 |
| Reading direction | reverse |
| Mean pairwise identity | 87.73 |
| Mean single sequence MFE | -48.56 |
| Consensus MFE | -37.86 |
| Energy contribution | -39.52 |
| Covariance contribution | 1.66 |
| Combinations/Pair | 1.06 |
| Mean z-score | -4.56 |
| Structure conservation index | 0.78 |
| SVM decision value | 3.26 |
| SVM RNA-class probability | 0.998871 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 13193100 100 - 27905053 UAUAUGGCGAUAUACGGCCGGACUGCACGGCCAUGGCUAUGG------CUAUGGCUAUCCGGCG--------AUCCGGGUAUCUGGGCAACUUUGUCCAAAAAAUGUUGCCGGG ....(((((((((....(((((.(((..(((((((((....)------)))))))).....)))--------.))))).....((((((....))))))....))))))))).. ( -44.80) >DroSec_CAF1 221016 106 - 1 UAUAUGGCGAUAUACGGCCGGACUGCACGGCCAUGGCUAUGGCUAUGGCUAUGGCUAUCCGGCG--------AUCCGGGUAUCUGGGCAACUUUGUCCAAAAAAUGUUGCCGGG ....(((((((((....(((((.(((.(((..(((((((((((....)))))))))))))))))--------.))))).....((((((....))))))....))))))))).. ( -49.80) >DroSim_CAF1 207074 100 - 1 UAUAUGGCGAUAUACGGCCGGACUGCACGGCCAUGGCUAUGG------CUAUGGCUAUCCGGCG--------AUCCGGGUAUCUGGGCAACUUUGUCCAAAAAAUGUUGCCGGG ....(((((((((....(((((.(((..(((((((((....)------)))))))).....)))--------.))))).....((((((....))))))....))))))))).. ( -44.80) >DroEre_CAF1 211753 114 - 1 UAUAUGGCGAUAUACGGCCGAACUGCACGGCCAUGGCUAUAGCUAUAGCUAUAGCUAUCCGGCGAUCCGGGGAUCCGGGUUGCCGGGCAACUUUGUCCAAAAAAUGUUGCCGGG ....(((((((((..(((((.......)))))(((((((((((....)))))))))))..((((((((((....))))))))))(((((....))))).....))))))))).. ( -59.50) >DroYak_CAF1 220800 94 - 1 UAUAUGGCGAUAUACGGCCGGAGUGCACGGCCAUUGCUAUAGC------------UAUCCGGCG--------AUCCGGGUAGCUGGGCAACUUUGUCCAAAAAAUGUUGCCGGG ....(((((((((..(((((.......))))).(((((.((((------------(((((((..--------..)))))))))))))))).............))))))))).. ( -43.90) >consensus UAUAUGGCGAUAUACGGCCGGACUGCACGGCCAUGGCUAUGGC_____CUAUGGCUAUCCGGCG________AUCCGGGUAUCUGGGCAACUUUGUCCAAAAAAUGUUGCCGGG ....(((((((((..(((((.......)))))..(((((((((....)))))))))((((((............))))))....(((((....))))).....))))))))).. (-37.86 = -39.52 + 1.66)



Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:44:07 2006