
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 12,893,559 – 12,893,700 |
| Length | 141 |
| Max. P | 0.994204 |

| Location | 12,893,559 – 12,893,662 |
|---|---|
| Length | 103 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 80.54 |
| Mean single sequence MFE | -27.05 |
| Consensus MFE | -18.98 |
| Energy contribution | -18.85 |
| Covariance contribution | -0.13 |
| Combinations/Pair | 1.10 |
| Mean z-score | -2.72 |
| Structure conservation index | 0.70 |
| SVM decision value | 2.46 |
| SVM RNA-class probability | 0.994204 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
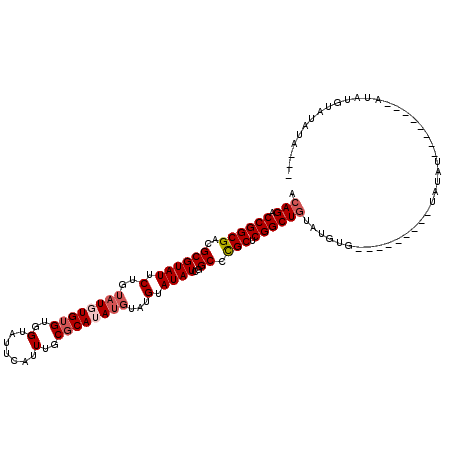
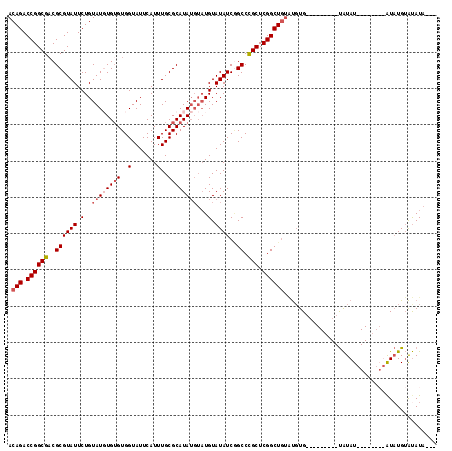
>3R_DroMel_CAF1 12893559 103 + 27905053 CAAAUGAACCCAUAAACAGACUCUGUGGGUUUGUGCACUAAUAUAUGUUCAUAU--------AUACA---------CACAGCCAGCCGAGCGGGCCGAUAUACAUACAUAUGCGCAAAUG .....(((((((((.........)))))))))(((((......(((((...(((--------((...---------....(((.((...)).)))..)))))...))))))))))..... ( -24.00) >DroSec_CAF1 11165 100 + 1 CAAAUGAACCCAUAAACAGACUCUGUGGGUUUGUGCACUA---UAUAUACAUAU--------AUAUA---------CACAUACAGCCGAGCGGGCAGAUAUACAUACAUAUGCGCAAAUG .....(((((((((.........)))))))))(((((.((---(((((....))--------)))))---------........(((.....)))...............)))))..... ( -23.60) >DroSim_CAF1 10177 104 + 1 CAAAUGAACCCAUAAACAGACUCUGUGGGUUUGUGCACUA---UAUACACAUAUAUGU----AUAUA---------CACGUACAGCCGAGCGGGCCGAUAUACAUACAUAUGCGCAAAUG .....(((((((((.........)))))))))(((((.((---((((((......)))----)))))---------...((((.(((.....))).)...))).......)))))..... ( -26.10) >DroYak_CAF1 11439 113 + 1 CAAAUGAACCCAUAAACAGACUCUGUGGGUUUGCGUACUA---UAUAUACAGACAUACAUACAUAUGUAUGUAUAUCAAACAGAGCCGAGCAGGCUGAUAUACA----UAUGCGCAAAUG .....(((((((((.........)))))))))(((((.((---(.((((..((.((((((((....)))))))).))......((((.....))))..)))).)----)))))))..... ( -34.50) >consensus CAAAUGAACCCAUAAACAGACUCUGUGGGUUUGUGCACUA___UAUAUACAUAU________AUAUA_________CACAUACAGCCGAGCGGGCCGAUAUACAUACAUAUGCGCAAAUG .....(((((((((.........)))))))))((((................................................(((.....)))..((((......))))))))..... (-18.98 = -18.85 + -0.13)

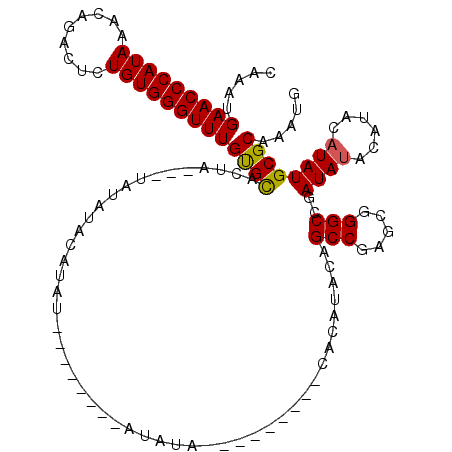
| Location | 12,893,559 – 12,893,662 |
|---|---|
| Length | 103 |
| Sequences | 4 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.54 |
| Mean single sequence MFE | -30.90 |
| Consensus MFE | -16.22 |
| Energy contribution | -17.29 |
| Covariance contribution | 1.06 |
| Combinations/Pair | 1.14 |
| Mean z-score | -2.66 |
| Structure conservation index | 0.52 |
| SVM decision value | 0.81 |
| SVM RNA-class probability | 0.855506 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 12893559 103 - 27905053 CAUUUGCGCAUAUGUAUGUAUAUCGGCCCGCUCGGCUGGCUGUG---------UGUAU--------AUAUGAACAUAUAUUAGUGCACAAACCCACAGAGUCUGUUUAUGGGUUCAUUUG ....((((((((((((((((((((((((.((...)).))))).)---------)))))--------))....))))))....)))))..((((((.(((.....))).))))))...... ( -29.00) >DroSec_CAF1 11165 100 - 1 CAUUUGCGCAUAUGUAUGUAUAUCUGCCCGCUCGGCUGUAUGUG---------UAUAU--------AUAUGUAUAUA---UAGUGCACAAACCCACAGAGUCUGUUUAUGGGUUCAUUUG ....(((((((((((((((((((.((((.((......))..).)---------)).))--------)))))))))))---).)))))..((((((.(((.....))).))))))...... ( -26.30) >DroSim_CAF1 10177 104 - 1 CAUUUGCGCAUAUGUAUGUAUAUCGGCCCGCUCGGCUGUACGUG---------UAUAU----ACAUAUAUGUGUAUA---UAGUGCACAAACCCACAGAGUCUGUUUAUGGGUUCAUUUG .........(((((((((((((((((((.....)))))...)))---------)))))----)))))).(((((((.---..)))))))((((((.(((.....))).))))))...... ( -32.20) >DroYak_CAF1 11439 113 - 1 CAUUUGCGCAUA----UGUAUAUCAGCCUGCUCGGCUCUGUUUGAUAUACAUACAUAUGUAUGUAUGUCUGUAUAUA---UAGUACGCAAACCCACAGAGUCUGUUUAUGGGUUCAUUUG ...(((((..((----(((((((.((((.....))))......(((((((((((....))))))))))).)))))))---))...)))))(((((.(((.....))).)))))....... ( -36.10) >consensus CAUUUGCGCAUAUGUAUGUAUAUCGGCCCGCUCGGCUGUAUGUG_________UAUAU________AUAUGUAUAUA___UAGUGCACAAACCCACAGAGUCUGUUUAUGGGUUCAUUUG ....(((((.....((((((((((((((.....)))))..............................))))))))).....)))))..((((((.(((.....))).))))))...... (-16.22 = -17.29 + 1.06)

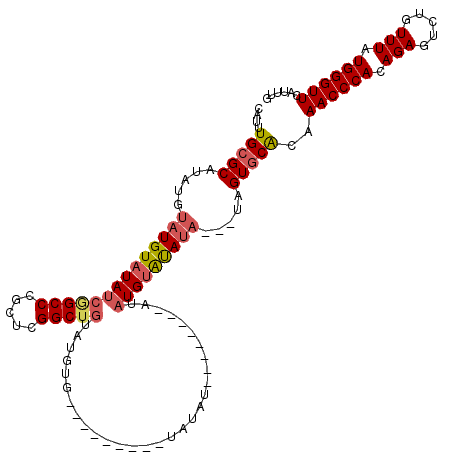
| Location | 12,893,599 – 12,893,700 |
|---|---|
| Length | 101 |
| Sequences | 4 |
| Columns | 118 |
| Reading direction | reverse |
| Mean pairwise identity | 79.57 |
| Mean single sequence MFE | -31.50 |
| Consensus MFE | -18.61 |
| Energy contribution | -19.93 |
| Covariance contribution | 1.31 |
| Combinations/Pair | 1.04 |
| Mean z-score | -2.33 |
| Structure conservation index | 0.59 |
| SVM decision value | 1.12 |
| SVM RNA-class probability | 0.918440 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 12893599 101 - 27905053 ACAGACCGGCGUCGCGUAUUCUGUAUGUGUGUGGUAUUCAUUUGCGCAUAUGUAUGUAUAUCGGCCCGCUCGGCUGGCUGUG---------UGUAU--------AUAUGAACAUAUAU (((((...(((...)))..)))))((((((((.(((......))).((((((((..((((((((((.....)))))).))))---------..)))--------))))).)))))))) ( -33.00) >DroSec_CAF1 11205 98 - 1 ACAGACCGGCGACGCGUAUUCUGUAUUUGUGUGGUAUUCAUUUGCGCAUAUGUAUGUAUAUCUGCCCGCUCGGCUGUAUGUG---------UAUAU--------AUAUGUAUAUA--- ((((.((((((.((.((((...((((.((((..(.......)..))))...))))..)))).))..))).))))))).((((---------((((.--------...))))))))--- ( -25.50) >DroSim_CAF1 10217 102 - 1 ACAGACCGGCGACGCGUAUUCUGUAUGUGUGUGGUAUUCAUUUGCGCAUAUGUAUGUAUAUCGGCCCGCUCGGCUGUACGUG---------UAUAU----ACAUAUAUGUGUAUA--- ....((((.(.((((((((...))))))))))))).......((((((((((((((((((((((((.....)))))...)))---------)))))----)))..))))))))..--- ( -34.30) >DroYak_CAF1 11479 111 - 1 ACAGACCGGCGACGCGUAUUCUGUAUUUGAGUGGUAUUCAUUUGCGCAUA----UGUAUAUCAGCCUGCUCGGCUCUGUUUGAUAUACAUACAUAUGUAUGUAUGUCUGUAUAUA--- (((((((((((..((..((((.......))))(((((.(((........)----)).))))).)).))).))).)))))..(((((((((((....)))))))))))........--- ( -33.20) >consensus ACAGACCGGCGACGCGUAUUCUGUAUGUGUGUGGUAUUCAUUUGCGCAUAUGUAUGUAUAUCGGCCCGCUCGGCUGUAUGUG_________UAUAU________AUAUGUAUAUA___ .(((.((((((..((((((.(..((((((((..(.......)..))))))))...).))))..)).))).)))))).......................................... (-18.61 = -19.93 + 1.31)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:41:51 2006