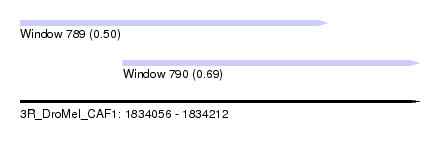
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 1,834,056 – 1,834,212 |
| Length | 156 |
| Max. P | 0.689233 |

| Location | 1,834,056 – 1,834,176 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.65 |
| Mean single sequence MFE | -44.32 |
| Consensus MFE | -30.48 |
| Energy contribution | -30.79 |
| Covariance contribution | 0.31 |
| Combinations/Pair | 1.29 |
| Mean z-score | -1.01 |
| Structure conservation index | 0.69 |
| SVM decision value | -0.07 |
| SVM RNA-class probability | 0.500000 |
| Prediction | OTHER |
Download alignment: ClustalW | MAF
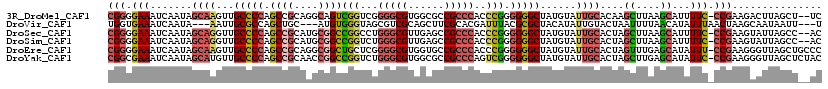
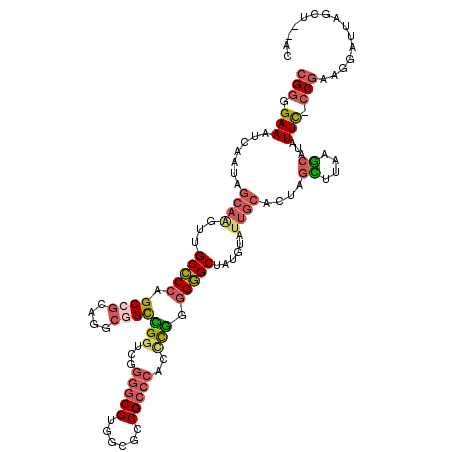
>3R_DroMel_CAF1 1834056 120 + 27905053 GACGUUGCGUUAUACAUUUCUCGGAGCUCGCGCCACUUCUCGGGGAAAUCAAUAGCAAGUUGCCCCAGCCGCAGGCAGUCGGUCGGGGCGUGGCGCCGCCCACCCGGGGGGCUAUGUAUU ...((..((((((..(((((((.(((............))).)))))))..))))).....(((((.(((((.....).)))).))))))..))(((.(((....))).)))........ ( -48.50) >DroVir_CAF1 315 105 + 1 GACGUUGCGUUAUACAUUUUUCCAUGCUGU---------GUGGUGAAAUCAAUA---AAUUGCGCCAGCUGC---AUGUGGGUAGCGUCGCAGCUUCGCACGAUUUACGCGCUACAUAUU ...((.((((.....(((((.((((.....---------)))).)))))...((---(((((.((.((((((---((((.....)))).))))))..)).))))))).)))).))..... ( -31.80) >DroSec_CAF1 297 120 + 1 GACGUUGCGUUAUACAUUUCUCGGAACUCGGGCCACUUCUCGGGGAAAUCAAUAGCAGGUUGCCCCAGCCGCAUGCGGCCGGCCUGGGCGUUGAGCCGCCCACCCGGGGGGCUAUGUAUU ((((...))))((((((...((((..((((((((.......((((.((((.......)))).)))).((((....)))).))))))))..))))(((.(((....))).))).)))))). ( -51.20) >DroSim_CAF1 312 120 + 1 GACGUUGCGUUAUACAUUUCUCGGAACUCGGGCCACUUCUCGGGGAAAUCAAUAGCAGGUUGCCCCAGCCGCAUGCGGCCGGUCUGGGCGUUGAGCCGCCCACCCGGGGGGCUAUGUAUU ((((...))))((((((...((((..((((((((.......((((.((((.......)))).)))).((((....)))).))))))))..))))(((.(((....))).))).)))))). ( -48.50) >DroEre_CAF1 294 111 + 1 GACGUUGCGUUAUACAUUUCUCGGAA---------CUUCUCGGGGAAAUCAAUAGCAAGUUGCCCCAGCCGCAGGCGGCUGCUCGGGGCGUGGUGCCGCCCACCCGGGGGGCUAUGUAUU (((.((((.......(((((((.((.---------....)).))))))).....)))))))((((((((((....))))))(((((((.(((....))).).))))))))))........ ( -45.20) >DroYak_CAF1 309 111 + 1 GACGUUUCGUUAUACAUCUCUCGGAA---------CUUUUCGGCGAAAUCAAUAGCAUGUUGCCCCAGCCGCAACCGGCCGGUCUGGGCGUGGCGCCGCCCAGUCGGGGGGCUAUGUAUU ...(((((((...........((((.---------...))))))))))).....(((((..(((((.((((....))))(((.(((((((......)))))))))).))))))))))... ( -40.70) >consensus GACGUUGCGUUAUACAUUUCUCGGAACUCG_____CUUCUCGGGGAAAUCAAUAGCAAGUUGCCCCAGCCGCAGGCGGCCGGUCGGGGCGUGGCGCCGCCCACCCGGGGGGCUAUGUAUU .((((...(((((..(((((((.((..............)).)))))))..))))).....(((((.((((....))))(((...(((((......)))))..))).))))).))))... (-30.48 = -30.79 + 0.31)

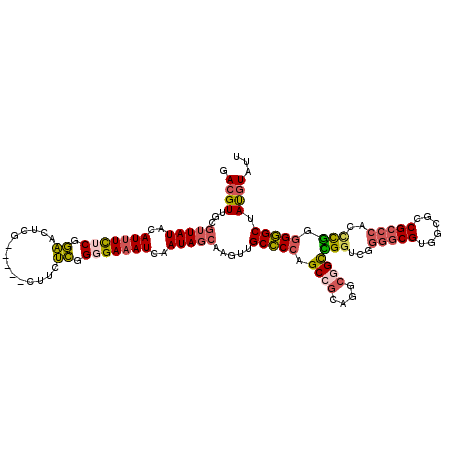
| Location | 1,834,096 – 1,834,212 |
|---|---|
| Length | 116 |
| Sequences | 6 |
| Columns | 119 |
| Reading direction | forward |
| Mean pairwise identity | 77.37 |
| Mean single sequence MFE | -44.36 |
| Consensus MFE | -28.16 |
| Energy contribution | -28.72 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.34 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.63 |
| SVM decision value | 0.32 |
| SVM RNA-class probability | 0.689233 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 1834096 116 + 27905053 CGGGGAAAUCAAUAGCAAGUUGCCCCAGCCGCAGGCAGUCGGUCGGGGCGUGGCGCCGCCCACCCGGGGGGCUAUGUAUUGCACAAGCUUAAGCAUUUUC-CCGAAGACUUAGCU--UC .((((((((.....((((...(((((.(((((.....).)))).)))))(((((.((.(((....))))))))))...))))....((....))))))))-))((((......))--)) ( -47.70) >DroVir_CAF1 346 110 + 1 UGGUGAAAUCAAUA---AAUUGCGCCAGCUGC---AUGUGGGUAGCGUCGCAGCUUCGCACGAUUUACGCGCUACAUAUUGUACUAAUUUUAACAUAUUUAACUAAGCAAUAAUU---U (((((.......((---(((((.((.((((((---((((.....)))).))))))..)).)))))))..)))))..((((((........................))))))...---. ( -23.96) >DroSec_CAF1 337 116 + 1 CGGGGAAAUCAAUAGCAGGUUGCCCCAGCCGCAUGCGGCCGGCCUGGGCGUUGAGCCGCCCACCCGGGGGGCUAUGUAUUGCACUAGCUUAAGCAUUUUC-CCGAAGUAUUAGCC--AC (((((((((.....((((...(((((.((((....))))(((..((((((......)))))).))).)))))......))))....((....))))))))-)))...........--.. ( -49.10) >DroSim_CAF1 352 116 + 1 CGGGGAAAUCAAUAGCAGGUUGCCCCAGCCGCAUGCGGCCGGUCUGGGCGUUGAGCCGCCCACCCGGGGGGCUAUGUAUUGCACUAGCUUAAGCAUUUUC-CCGAAGUAUUAGCC--AC (((((((((.....((((...(((((.((((....))))(((..((((((......)))))).))).)))))......))))....((....))))))))-)))...........--.. ( -49.70) >DroEre_CAF1 325 118 + 1 CGGGGAAAUCAAUAGCAAGUUGCCCCAGCCGCAGGCGGCUGCUCGGGGCGUGGUGCCGCCCACCCGGGGGGCUAUGUAUUGCACUAGUUUGAGCAUAUUU-CCGAAGGGUUAGCUGCCC (((..(..((((..((.(((.((((((((((....)))))....)))))(..((((.((((.(....)))))...))))..)))).)))))).....)..-)))..((((.....)))) ( -47.20) >DroYak_CAF1 340 118 + 1 CGGCGAAAUCAAUAGCAUGUUGCCCCAGCCGCAACCGGCCGGUCUGGGCGUGGCGCCGCCCAGUCGGGGGGCUAUGUAUUGCACUAGCUUGAGCAUAUUC-CCGAAGGGUUAGCUCUAC .(((.....(((((.((((..(((((.((((....))))(((.(((((((......)))))))))).)))))))))))))).....))).((((.((...-((....)).))))))... ( -48.50) >consensus CGGGGAAAUCAAUAGCAAGUUGCCCCAGCCGCAGGCGGCCGGUCGGGGCGUGGCGCCGCCCACCCGGGGGGCUAUGUAUUGCACUAGCUUAAGCAUAUUC_CCGAAGGAUUAGCU__AC (((.(((.......((((...(((((.((((....))))(((...(((((......)))))..))).)))))......))))....((....))...))).)))............... (-28.16 = -28.72 + 0.56)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 09:44:27 2006