
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 12,758,801 – 12,758,942 |
| Length | 141 |
| Max. P | 0.999628 |

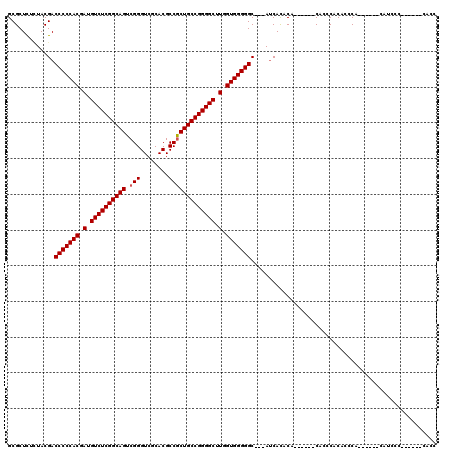
| Location | 12,758,801 – 12,758,902 |
|---|---|
| Length | 101 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 80.30 |
| Mean single sequence MFE | -42.47 |
| Consensus MFE | -39.17 |
| Energy contribution | -39.33 |
| Covariance contribution | 0.17 |
| Combinations/Pair | 1.00 |
| Mean z-score | -2.38 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.63 |
| SVM RNA-class probability | 0.968448 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
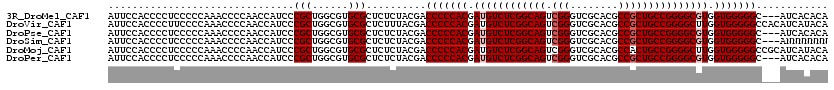
>3R_DroMel_CAF1 12758801 101 - 27905053 GCGCUCUCUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCGUGGUGGGGGC---AUCACACA----CACACCCACACCCA------CGUACC------CACC ...........(.(((((((.((((((((((((.(((........))))))))))))))).))))))))---........----..............------......------.... ( -44.90) >DroVir_CAF1 157753 98 - 1 GCGCUCUUUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCUUGGUGGGGGCCACAUCAUACA------CAUCCACAUCCGCACCCC---------------- (((.((.....))(((((((.(.((((((((((.(((........))))))))))))).).)))))))............------..........))).....---------------- ( -42.20) >DroGri_CAF1 220126 102 - 1 GCGCUCUCUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCUUGGUGGGGGCCACAUCACACA------CACCCACAUCCC---CCACAUCCGC--------- ...........(.(((((((.(.((((((((((.(((........))))))))))))).).))))))).)..........------............---..........--------- ( -41.00) >DroWil_CAF1 176870 105 - 1 GCGCUCUCUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCUUGGUGGGGGC---AUCAUACACACCCACAUCCACACCCA------CAUCCC------CAUC ...........(.(((((((.(.((((((((((.(((........))))))))))))).).))))))))---..........................------......------.... ( -41.00) >DroMoj_CAF1 164466 114 - 1 GCGCUCUCUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCACUGCCGGGGCUUGGUGGGGGCCGCAUCAUACA------CAUCCUCAUCCACAUCCACAUCCACACCCCCACC ..........((.(((((((.(.(((((((((((.((........))))))))))))).).))))))).)).........------.................................. ( -40.80) >DroAna_CAF1 120959 104 - 1 GCGCUUUAUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCGUGGUGGGGGC---ACCACACAC-CACACACCCACUCCCA------CACCCC------CACG ...........(.(((((((.((((((((((((.(((........))))))))))))))).))))))))---.........-................------......------.... ( -44.90) >consensus GCGCUCUCUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCUUGGUGGGGGC___AUCACACA______CACCCACACCCA______CAUCCC______CACC .............(((((((.(.((((((((((.(((........))))))))))))).).))))))).................................................... (-39.17 = -39.33 + 0.17)


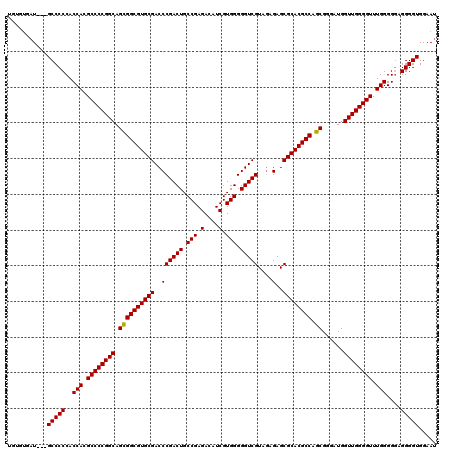
| Location | 12,758,825 – 12,758,942 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.01 |
| Mean single sequence MFE | -59.10 |
| Consensus MFE | -56.65 |
| Energy contribution | -56.52 |
| Covariance contribution | -0.14 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.18 |
| Structure conservation index | 0.96 |
| SVM decision value | 3.81 |
| SVM RNA-class probability | 0.999628 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 12758825 117 + 27905053 UGUGUGAU---GCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAGAGAGCGCACGCCAGCGGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAU ....(.((---.(((((.(((.((((((((.((((((((((..((((((.(((.(....).))).)))))....)..)))))))).))......)))))))).)))))).)).)).)... ( -58.20) >DroVir_CAF1 157771 120 + 1 UGUAUGAUGUGGCCCCCACCAAGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAAAGAGCGCACGCCAGCGGGAUGGUUGGGGUUUGGGGAAGGGGUGGAAU .........(.(((((..((((((((((((.((((((((((..((((((.(((.(....).))).)))))....)..)))))))).))......))))))))))))....))))).)... ( -60.60) >DroPse_CAF1 142541 117 + 1 UGUGUGAU---GCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAGAGAGCGCACGCCAGCGGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAU ....(.((---.(((((.(((.((((((((.((((((((((..((((((.(((.(....).))).)))))....)..)))))))).))......)))))))).)))))).)).)).)... ( -58.20) >DroSim_CAF1 135603 117 + 1 NNNNNNNU---GCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAGAGAGCGCACGCCAGCGGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAU ........---((((((.(((.((((((((.((((((((((..((((((.(((.(....).))).)))))....)..)))))))).))......)))))))).)))..).)))))..... ( -57.90) >DroMoj_CAF1 164500 120 + 1 UGUAUGAUGCGGCCCCCACCAAGCCCCGGCAGUGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAGAGAGCGCACGCCAGCGGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAU .........(.((((((.((((((((((((.((((((((((..((((((.(((.(....).))).)))))....)..))))))))).)......))))))))))))..).))))).)... ( -61.50) >DroPer_CAF1 149292 117 + 1 UGUGUGAU---GCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAGAGAGCGCACGCCAGCGGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAU ....(.((---.(((((.(((.((((((((.((((((((((..((((((.(((.(....).))).)))))....)..)))))))).))......)))))))).)))))).)).)).)... ( -58.20) >consensus UGUGUGAU___GCCCCCACCACGCCCCGGCAGCGGCGUGCGACCCGACUGCCGAGACAUCGUGGGGGUCGUAGAGAGCGCACGCCAGCGGGAUGGUUGGGGUUUGGGGGAGGGGUGGAAU ...........(((((..(((.((((((((.((((((((((..((((((.(((.(....).))).)))))....)..)))))))).))......)))))))).)))....)))))..... (-56.65 = -56.52 + -0.14)


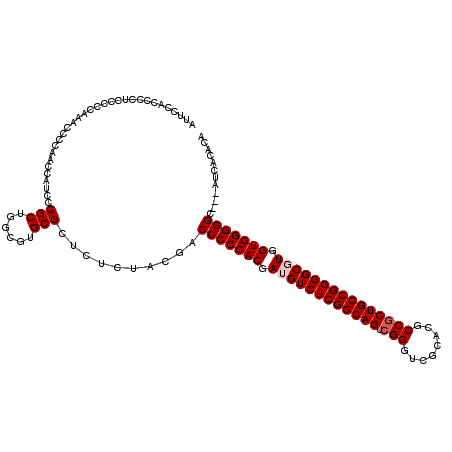
| Location | 12,758,825 – 12,758,942 |
|---|---|
| Length | 117 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.01 |
| Mean single sequence MFE | -46.90 |
| Consensus MFE | -44.90 |
| Energy contribution | -45.40 |
| Covariance contribution | 0.50 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.91 |
| Structure conservation index | 0.96 |
| SVM decision value | 3.20 |
| SVM RNA-class probability | 0.998734 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 12758825 117 - 27905053 AUUCCACCCCUCCCCCAAACCCCAACCAUCCCGCUGGCGUGCGCUCUCUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCGUGGUGGGGGC---AUCACACA ...............................(((......)))........(.(((((((.((((((((((((.(((........))))))))))))))).))))))))---........ ( -48.00) >DroVir_CAF1 157771 120 - 1 AUUCCACCCCUUCCCCAAACCCCAACCAUCCCGCUGGCGUGCGCUCUUUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCUUGGUGGGGGCCACAUCAUACA ......(((((...((((.((((...((....((.((((((((.......)(((((......(((....)))...))))))))))))))))..)))).)))).)))))............ ( -44.80) >DroPse_CAF1 142541 117 - 1 AUUCCACCCCUCCCCCAAACCCCAACCAUCCCGCUGGCGUGCGCUCUCUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCGUGGUGGGGGC---AUCACACA ...............................(((......)))........(.(((((((.((((((((((((.(((........))))))))))))))).))))))))---........ ( -48.00) >DroSim_CAF1 135603 117 - 1 AUUCCACCCCUCCCCCAAACCCCAACCAUCCCGCUGGCGUGCGCUCUCUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCGUGGUGGGGGC---ANNNNNNN ...............................(((......)))........(.(((((((.((((((((((((.(((........))))))))))))))).))))))))---........ ( -48.00) >DroMoj_CAF1 164500 120 - 1 AUUCCACCCCUCCCCCAAACCCCAACCAUCCCGCUGGCGUGCGCUCUCUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCACUGCCGGGGCUUGGUGGGGGCCGCAUCAUACA ...........((((((....((((...(((((((((((((((.......)(((((......(((....)))...)))))))))))))..).))))).))))))))))............ ( -44.60) >DroPer_CAF1 149292 117 - 1 AUUCCACCCCUCCCCCAAACCCCAACCAUCCCGCUGGCGUGCGCUCUCUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCGUGGUGGGGGC---AUCACACA ...............................(((......)))........(.(((((((.((((((((((((.(((........))))))))))))))).))))))))---........ ( -48.00) >consensus AUUCCACCCCUCCCCCAAACCCCAACCAUCCCGCUGGCGUGCGCUCUCUACGACCCCCACGAUGUCUCGGCAGUCGGGUCGCACGCCGCUGCCGGGGCGUGGUGGGGGC___AUCACACA ...............................(((......)))..........(((((((.((((((((((((.(((........))))))))))))))).)))))))............ (-44.90 = -45.40 + 0.50)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:40:36 2006