
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 12,421,646 – 12,421,750 |
| Length | 104 |
| Max. P | 0.582053 |

| Location | 12,421,646 – 12,421,750 |
|---|---|
| Length | 104 |
| Sequences | 6 |
| Columns | 118 |
| Reading direction | forward |
| Mean pairwise identity | 72.79 |
| Mean single sequence MFE | -31.28 |
| Consensus MFE | -17.69 |
| Energy contribution | -17.80 |
| Covariance contribution | 0.11 |
| Combinations/Pair | 1.22 |
| Mean z-score | -1.41 |
| Structure conservation index | 0.57 |
| SVM decision value | 0.10 |
| SVM RNA-class probability | 0.582053 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
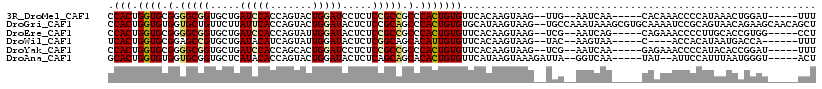
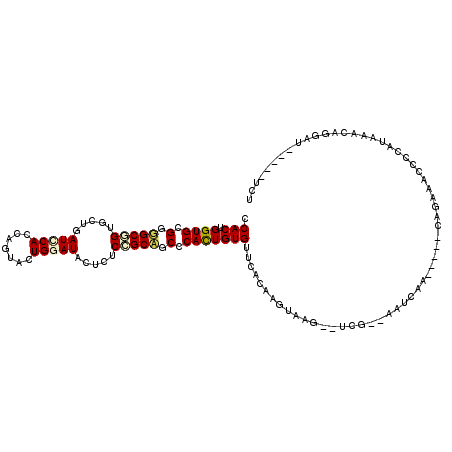
>3R_DroMel_CAF1 12421646 104 + 27905053 CCACUGGUGCGGGGCGGUGCUGAUCCACCAGUACUGGAUCCUCUCCGCCGCCCACUGUGUUCACAAGUAAG--UUG--AAUCAA-----CACAAACCCCAUAAACUGGAU-----UUU .(((.((((.(((((((.(..((((((.......))))))..).))))).)))))))))...........(--(((--...)))-----).......(((.....)))..-----... ( -31.50) >DroGri_CAF1 21589 116 + 1 CCACUGGUGUGGUGCUGUUCUUAUUCACCAGUACUGGAUACUCUCCGCAGCCCACUGUGUGCAUAAGUAAG--UGCCAAAUAAAGCGUGCAAAAUCCGCAGUAACAGAAGCAACAGCU ...(((...(((..((...(((((.(((((((.((((........).)))...)))).))).)))))..))--..)))......((.(((.......)))))..))).(((....))) ( -29.00) >DroEre_CAF1 20848 104 + 1 CCACUGGUGCGGGGCGGUGCUGAUCCACCAGUAUUGGAUACUCUCCGCCGCCCACUGUGUUCACAAGUAAG--UCG--AAUCAG-----CAGAAACCCCUUGCACCGUGG-----CCU ((((.((((((((((((((..((((((.......))))...))..)))))))).((((((((((......)--).)--)))..)-----)))........))))))))))-----... ( -42.00) >DroWil_CAF1 23078 99 + 1 UCACUGGUGCGGAGCCGUGCUGAUACAUCAGUAUUGGAUACUCUCGGCAGCACAUUGUGUUCACAAGUAAG--UAC--AAGUAA-----C----ACCACAUAAUGACCA------UUU ....((((((.(((..((((..((((....))))..).))).))).))....((((((((...........--...--......-----.----...))))))))))))------... ( -23.56) >DroYak_CAF1 21202 104 + 1 CCACUGGUGCGGGGCGGUGCUGAUCCACCAGCACUGGAUCCUCUCCGCCGCCCACUGUGUUCACAAGUAAG--UCG--AAUCAA-----GAGAAACCCCAUACACCGGAU-----UUU ...((((((..(((((((((((......))))...(((.....)))))))))).((..((((((......)--).)--)))..)-----)............))))))..-----... ( -34.90) >DroAna_CAF1 20004 104 + 1 GCACUGGUGUGGUGCGGUGCUCAUACACCAGUACUGGAUACUCUCAGCAGCACACUGUGUUCAUAAGUAAAGAUUA--GGUCAA-----UAU--AUUCCAUUUAAUGGGU-----ACU ((((.((((((.(((((((......))))((((.....))))....))).)))))))))).....((((..(((..--.)))..-----...--..(((((...))))))-----))) ( -26.70) >consensus CCACUGGUGCGGGGCGGUGCUGAUCCACCAGUACUGGAUACUCUCCGCAGCCCACUGUGUUCACAAGUAAG__UCG__AAUCAA_____CAGAAACCCCAUAAACAGGAU_____UCU .(((.((((.(.(((((.....(((((.......))))).....))))).).)))))))........................................................... (-17.69 = -17.80 + 0.11)
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:37:41 2006