
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 12,095,218 – 12,095,338 |
| Length | 120 |
| Max. P | 0.577284 |

| Location | 12,095,218 – 12,095,338 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 76.67 |
| Mean single sequence MFE | -49.34 |
| Consensus MFE | -27.24 |
| Energy contribution | -27.33 |
| Covariance contribution | 0.09 |
| Combinations/Pair | 1.54 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.02 |
| SVM RNA-class probability | 0.545931 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
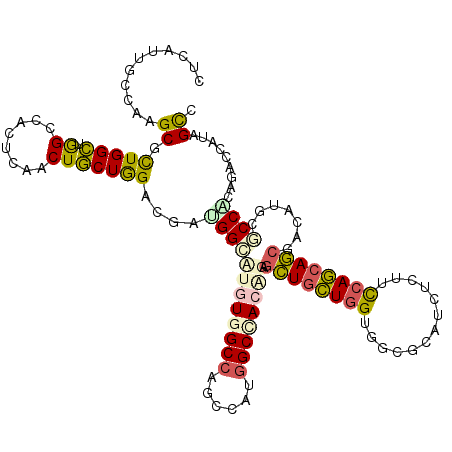
>3R_DroMel_CAF1 12095218 120 + 27905053 CGCUAUGGACUGCGGUGCAUACCGCUGCUGAAAGAGAUGCGCCACCAGUAGCUCAGUAGCCAUAUCAGGCCACAAGCCAUCGUCCAGCAGUUGUGUGGCCAGCCAGCGCUUGGCCAACAG .(((.(((((.(((((....))))).(((....(((.(((.......))).))).((.(((......))).)).)))....)))))..)))(((.((((((((....)).))))))))). ( -42.60) >DroVir_CAF1 992 120 + 1 GACAGUAGUGUGUGGCGUCUGUCGCUGCUGGAACAGAUGCGCCACAAGCAGCUCCGUCGCUAUGGACGGCCACAGACCAUCGUCCAGCAGCUGUGCAGCCAGCCAGCGCUUUGCAAUGAG ....((((.(((((((..((((.(((((((((...((((.((.....))....(((((......)))))........)))).)))))))))...))))...)))).))).))))...... ( -47.30) >DroPse_CAF1 2283 120 + 1 CGCUAUGGCCUGAGGAGCAUUUCGCUGCUGGUACAGAUGGGCAACCAGCAGCUCUGUGGCCAUCGGGGGCCAGAUCCCGUCAUCCAGCAGUUGAGUGGCCAGCCAGCGCUUGGCAAUCAG .((((.((((((.((..(((((.((((((((....((((((....(((.....)))(((((......)))))...))))))..)))))))).))))).))...))).)))))))...... ( -49.60) >DroGri_CAF1 1593 120 + 1 GGCUGUUGUAUGUGGCAUCUGUCGCUGUUGGAAGAGAUGCGCCACCAACAGUUCCGUUGCCAUGUCUGGCCACAGACCAUUGUCUAGCAGCUGGGUGGCCAGCCAACGCUUGGCAAUGAG ((((((((...(((((((((.((.(....)))..))))..))))))))))))).((((((((.((((((((((...((((((.....))).))))))))))).....)).)))))))).. ( -48.60) >DroAna_CAF1 1122 120 + 1 GGCAAUGGGCUGGGGGGCAUGGCGUUGCUGGAAGAGAUGCGCCACCAGCAACUCAGUGGCCAUAGCUGGCCACAAGCCCUCAUCCAGAAGCUGAGUGGCUAUCCAGCGCUUUGCCAGGAG (((((.(.((((((((((.(((.((((((((.............)))))))))))(((((((....)))))))..)))))).......((((....))))...)))).).)))))..... ( -58.32) >DroPer_CAF1 2267 120 + 1 CGCUAUGGCCUGAGGAGCAUUUCGCUGCUGGUACAGAUGGGCAACCAGCAGCUCUGUGGCCAUCGGGGGCCAGAUCCCGUCAUCCAGCAGUUGAGUGGCCAGCCAGCGCUUGGCAAUCAG .((((.((((((.((..(((((.((((((((....((((((....(((.....)))(((((......)))))...))))))..)))))))).))))).))...))).)))))))...... ( -49.60) >consensus CGCUAUGGCCUGAGGAGCAUGUCGCUGCUGGAACAGAUGCGCCACCAGCAGCUCAGUGGCCAUAGCGGGCCACAAACCAUCAUCCAGCAGCUGAGUGGCCAGCCAGCGCUUGGCAAUCAG .((((.(((....(((.(((((.(((((((((...((((.((.....))......((.(((......))).))....)))).))))))))).)))))....)))...)))))))...... (-27.24 = -27.33 + 0.09)



| Location | 12,095,218 – 12,095,338 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 76.67 |
| Mean single sequence MFE | -47.65 |
| Consensus MFE | -26.44 |
| Energy contribution | -26.84 |
| Covariance contribution | 0.40 |
| Combinations/Pair | 1.48 |
| Mean z-score | -1.60 |
| Structure conservation index | 0.55 |
| SVM decision value | 0.09 |
| SVM RNA-class probability | 0.577284 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 12095218 120 - 27905053 CUGUUGGCCAAGCGCUGGCUGGCCACACAACUGCUGGACGAUGGCUUGUGGCCUGAUAUGGCUACUGAGCUACUGGUGGCGCAUCUCUUUCAGCAGCGGUAUGCACCGCAGUCCAUAGCG .(((((((((.((....)))))))).)))...(((((((..((((((((((((......)))))).))))))...((((.((((((((......)).)).)))).)))).))))..))). ( -50.30) >DroVir_CAF1 992 120 - 1 CUCAUUGCAAAGCGCUGGCUGGCUGCACAGCUGCUGGACGAUGGUCUGUGGCCGUCCAUAGCGACGGAGCUGCUUGUGGCGCAUCUGUUCCAGCAGCGACAGACGCCACACACUACUGUC ..(((((...(((((((((.....)).)))).)))...)))))(.((((((....)))))))(((((((.((..(((((((..((((((.(....).))))))))))))))))).))))) ( -46.20) >DroPse_CAF1 2283 120 - 1 CUGAUUGCCAAGCGCUGGCUGGCCACUCAACUGCUGGAUGACGGGAUCUGGCCCCCGAUGGCCACAGAGCUGCUGGUUGCCCAUCUGUACCAGCAGCGAAAUGCUCCUCAGGCCAUAGCG .(((..((((.((....))))))...)))...(((......((((........))))((((((.....(((((((((.((......)))))))))))((........)).))))))))). ( -46.00) >DroGri_CAF1 1593 120 - 1 CUCAUUGCCAAGCGUUGGCUGGCCACCCAGCUGCUAGACAAUGGUCUGUGGCCAGACAUGGCAACGGAACUGUUGGUGGCGCAUCUCUUCCAACAGCGACAGAUGCCACAUACAACAGCC .((((((.(.(((...((((((....))))))))).).))))))(((((.((((....)))).))))).(((((((((..((((((.((.(....).)).))))))..))).)))))).. ( -44.40) >DroAna_CAF1 1122 120 - 1 CUCCUGGCAAAGCGCUGGAUAGCCACUCAGCUUCUGGAUGAGGGCUUGUGGCCAGCUAUGGCCACUGAGUUGCUGGUGGCGCAUCUCUUCCAGCAACGCCAUGCCCCCCAGCCCAUUGCC .....(((((.(.(((((.....((..(((...)))..)).((((..(((((((....)))))))...((((((((.((.......)).)))))))).....)))).))))).).))))) ( -53.00) >DroPer_CAF1 2267 120 - 1 CUGAUUGCCAAGCGCUGGCUGGCCACUCAACUGCUGGAUGACGGGAUCUGGCCCCCGAUGGCCACAGAGCUGCUGGUUGCCCAUCUGUACCAGCAGCGAAAUGCUCCUCAGGCCAUAGCG .(((..((((.((....))))))...)))...(((......((((........))))((((((.....(((((((((.((......)))))))))))((........)).))))))))). ( -46.00) >consensus CUCAUUGCCAAGCGCUGGCUGGCCACUCAACUGCUGGACGAUGGCAUGUGGCCAGCCAUGGCCACAGAGCUGCUGGUGGCGCAUCUCUUCCAGCAGCGACAUGCGCCACAGACCAUAGCC ...........((.(((((.((........)))))))....((((((((((((......)))))))).((((((((.............)))))))).......)))).........)). (-26.44 = -26.84 + 0.40)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:34:16 2006