
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 12,085,015 – 12,085,215 |
| Length | 200 |
| Max. P | 0.999697 |

| Location | 12,085,015 – 12,085,135 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.92 |
| Mean single sequence MFE | -49.02 |
| Consensus MFE | -45.10 |
| Energy contribution | -45.70 |
| Covariance contribution | 0.60 |
| Combinations/Pair | 1.05 |
| Mean z-score | -1.59 |
| Structure conservation index | 0.92 |
| SVM decision value | 1.63 |
| SVM RNA-class probability | 0.968631 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 12085015 120 + 27905053 CUCCAAUGUGUCGGCGGGCGCCAAUACCUGGGAGUACGGAUUCCUGGACAUGGAAUUCGGACUCGGUUCGGAGUUCACGGAGCUGGUACCCAGCUGUAAGCUCAGCUCCGAGGAUCUGUU ((((...((((.(((....))).))))...))))..((((((((.......))))))))..(((((....(((((.((..((((((...)))))))).)))))....)))))........ ( -48.50) >DroSec_CAF1 21706 120 + 1 CUCCAAUGUGUCGGCGGGCGCCAAUACCUGGGAGUACGGAUUCCUGGACAUGGAAUUCGGACUGGGGUCGGAGUUCACGGAGCUGGUACCCAGCUGUAAGCUCAGCUCCGAGGAUCUGUU ((((...((((.(((....))).))))...)))).((((((.(((.......((((((.(((....))).)))))).((((((((......(((.....)))))))))))))))))))). ( -50.80) >DroSim_CAF1 21894 120 + 1 CUCCAAUGUUUCGGCGGGCGCCAAUACCUGGGAGUACGGAUUCCUGGACAUGGAAUUCGGACUGGGGUCGGAGUUCACGGAGCUGGUACCCAGCUGUAAGCUCAGCUCCGAGGAUCUGUU ((((...((...(((....)))...))...)))).((((((.(((.......((((((.(((....))).)))))).((((((((......(((.....)))))))))))))))))))). ( -47.50) >DroEre_CAF1 23797 120 + 1 CUCCAAUGUGUCGGCGGGCGCCAAUACCUGGGAGUACGGAUUCCUGGACAUGGAGUUCGGACUGGGGUCGGAGUUCACGGAGCUGGUGCCGAGCUGUAAGCUCAGCUCCGAGGAUCUAUU ((((...((((.(((....))).))))...))))..((((((((.......))))))))(((....)))(((.(((.((((((((....)((((.....))))))))))).))))))... ( -48.70) >DroYak_CAF1 24179 120 + 1 CUCUAAUGUGUCGGCGGGAGCCAAUACCUGGGAGUACGGAUUCCUGGACAUGGAGUUCGGACUGGGGUCGGAGUUCACGGAGCUGGUGCCGAGCUGUAAGCUCAGCUCGGAGGAUCUCUU ((((...((((.(((....))).))))...))))..((((((((.......))))))))....((((((..(((((...)))))....((((((((......))))))))..)))))).. ( -49.60) >consensus CUCCAAUGUGUCGGCGGGCGCCAAUACCUGGGAGUACGGAUUCCUGGACAUGGAAUUCGGACUGGGGUCGGAGUUCACGGAGCUGGUACCCAGCUGUAAGCUCAGCUCCGAGGAUCUGUU ((((...((((.(((....))).))))...)))).((((((.(((.......((((((.(((....))).)))))).((((((((......(((.....)))))))))))))))))))). (-45.10 = -45.70 + 0.60)

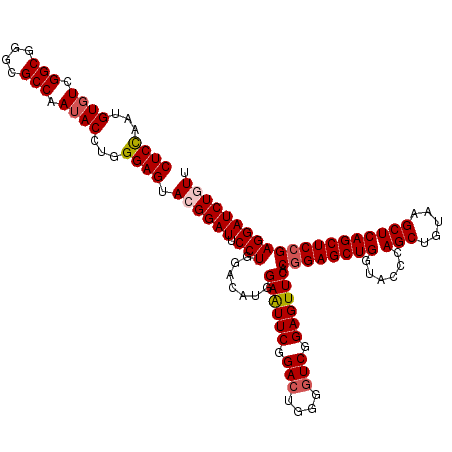
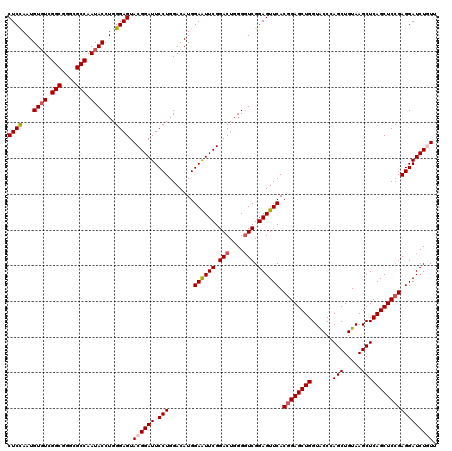
| Location | 12,085,015 – 12,085,135 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.92 |
| Mean single sequence MFE | -38.94 |
| Consensus MFE | -35.90 |
| Energy contribution | -36.86 |
| Covariance contribution | 0.96 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.16 |
| Structure conservation index | 0.92 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.612711 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 12085015 120 - 27905053 AACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCUCCGUGAACUCCGAACCGAGUCCGAAUUCCAUGUCCAGGAAUCCGUACUCCCAGGUAUUGGCGCCCGCCGACACAUUGGAG ......((..((((((((.(((((....)))))..)))))))).)).((((((...((((.((.(((((.......))))).)).))))....((.(((((....))))).)).)))))) ( -43.60) >DroSec_CAF1 21706 120 - 1 AACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCUCCGUGAACUCCGACCCCAGUCCGAAUUCCAUGUCCAGGAAUCCGUACUCCCAGGUAUUGGCGCCCGCCGACACAUUGGAG ......((..((((((((.(((((....)))))..)))))))).)).((((((....(((.((.(((((.......))))).)).))).....((.(((((....))))).)).)))))) ( -39.70) >DroSim_CAF1 21894 120 - 1 AACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCUCCGUGAACUCCGACCCCAGUCCGAAUUCCAUGUCCAGGAAUCCGUACUCCCAGGUAUUGGCGCCCGCCGAAACAUUGGAG ......((..((((((((.(((((....)))))..)))))))).)).((((((....(((.((.(((((.......))))).)).))).....((.(((((....))))).)).)))))) ( -39.70) >DroEre_CAF1 23797 120 - 1 AAUAGAUCCUCGGAGCUGAGCUUACAGCUCGGCACCAGCUCCGUGAACUCCGACCCCAGUCCGAACUCCAUGUCCAGGAAUCCGUACUCCCAGGUAUUGGCGCCCGCCGACACAUUGGAG ....((((((.((((((((((.....)))))))........((((...((.(((....))).))....)))))))))).)))....((((...((.(((((....))))).))...)))) ( -39.30) >DroYak_CAF1 24179 120 - 1 AAGAGAUCCUCCGAGCUGAGCUUACAGCUCGGCACCAGCUCCGUGAACUCCGACCCCAGUCCGAACUCCAUGUCCAGGAAUCCGUACUCCCAGGUAUUGGCUCCCGCCGACACAUUAGAG ..(((.....((((((((......))))))))......))).(((.......(((..(((.((...(((.......)))...)).)))....))).(((((....))))))))....... ( -32.40) >consensus AACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCUCCGUGAACUCCGACCCCAGUCCGAAUUCCAUGUCCAGGAAUCCGUACUCCCAGGUAUUGGCGCCCGCCGACACAUUGGAG ......((..((((((((.(((((....)))))..)))))))).)).((((((....(((.((.(((((.......))))).)).))).....((.(((((....))))).)).)))))) (-35.90 = -36.86 + 0.96)


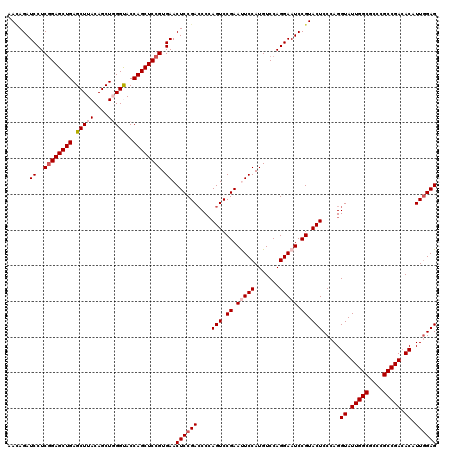
| Location | 12,085,055 – 12,085,175 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.42 |
| Mean single sequence MFE | -52.42 |
| Consensus MFE | -48.30 |
| Energy contribution | -48.86 |
| Covariance contribution | 0.56 |
| Combinations/Pair | 1.02 |
| Mean z-score | -1.73 |
| Structure conservation index | 0.92 |
| SVM decision value | 2.16 |
| SVM RNA-class probability | 0.989389 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 12085055 120 + 27905053 UUCCUGGACAUGGAAUUCGGACUCGGUUCGGAGUUCACGGAGCUGGUACCCAGCUGUAAGCUCAGCUCCGAGGAUCUGUUCAAGAGCGGAUUGGGCGGUCAGGUUGUGACCGCCUCCCGC .((.((((((.((..(((((((((......))))))..(((((((......(((.....)))))))))))))..)))))))).))((((...(((((((((.....))))))))).)))) ( -53.20) >DroSec_CAF1 21746 120 + 1 UUCCUGGACAUGGAAUUCGGACUGGGGUCGGAGUUCACGGAGCUGGUACCCAGCUGUAAGCUCAGCUCCGAGGAUCUGUUCAAGAGCGGAUUGGGCGGUCAGGUUGUGACAGCCUCGCGC .((.((((((..((((((.(((....))).)))))).((((((((......(((.....)))))))))))......)))))).))(((....((((.((((.....)))).))))))).. ( -47.60) >DroSim_CAF1 21934 120 + 1 UUCCUGGACAUGGAAUUCGGACUGGGGUCGGAGUUCACGGAGCUGGUACCCAGCUGUAAGCUCAGCUCCGAGGAUCUGUUCAAGAGCGGAUUGGGCGGUCAGGUUGUGACCGCCUCCCGC .((.((((((..((((((.(((....))).)))))).((((((((......(((.....)))))))))))......)))))).))((((...(((((((((.....))))))))).)))) ( -56.00) >DroEre_CAF1 23837 120 + 1 UUCCUGGACAUGGAGUUCGGACUGGGGUCGGAGUUCACGGAGCUGGUGCCGAGCUGUAAGCUCAGCUCCGAGGAUCUAUUCAAGAGCGGUCUGGGCGGUCAGGUUGUGACCGCCUCCCGC .((((.......((..((.(((....))).))..)).((((((((....)((((.....)))))))))))))))...........((((...(((((((((.....))))))))).)))) ( -52.80) >DroYak_CAF1 24219 120 + 1 UUCCUGGACAUGGAGUUCGGACUGGGGUCGGAGUUCACGGAGCUGGUGCCGAGCUGUAAGCUCAGCUCGGAGGAUCUCUUCAAGAGCGGACUGGGCGGUCAGGUUGUGACCGCCUCCCGC ...((((((.....))))))...((((((...((((..((((..(((.((((((((......))))))))...))).))))..)))).))).(((((((((.....)))))))))))).. ( -52.50) >consensus UUCCUGGACAUGGAAUUCGGACUGGGGUCGGAGUUCACGGAGCUGGUACCCAGCUGUAAGCUCAGCUCCGAGGAUCUGUUCAAGAGCGGAUUGGGCGGUCAGGUUGUGACCGCCUCCCGC .((((.......((((((.(((....))).)))))).((((((((......(((.....)))))))))))))))(((.....)))((((...(((((((((.....))))))))).)))) (-48.30 = -48.86 + 0.56)

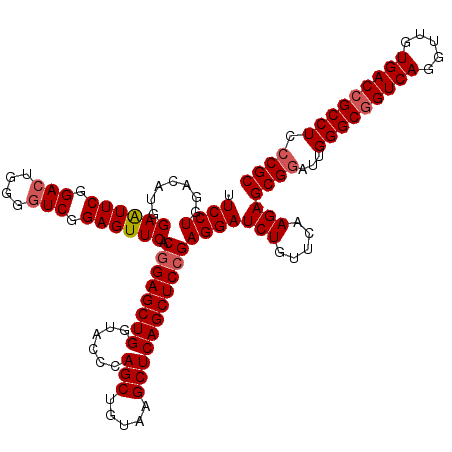
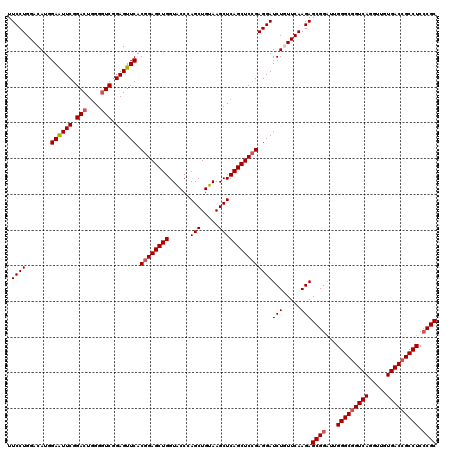
| Location | 12,085,055 – 12,085,175 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.42 |
| Mean single sequence MFE | -47.16 |
| Consensus MFE | -42.34 |
| Energy contribution | -43.98 |
| Covariance contribution | 1.64 |
| Combinations/Pair | 1.05 |
| Mean z-score | -2.69 |
| Structure conservation index | 0.90 |
| SVM decision value | 3.90 |
| SVM RNA-class probability | 0.999697 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 12085055 120 - 27905053 GCGGGAGGCGGUCACAACCUGACCGCCCAAUCCGCUCUUGAACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCUCCGUGAACUCCGAACCGAGUCCGAAUUCCAUGUCCAGGAA (((((.((((((((.....))))))))...)))))(((((.(((....(((((..(.(((.((((((((((...))))))..)))).))).)..)))))...(.....).))).))))). ( -49.30) >DroSec_CAF1 21746 120 - 1 GCGCGAGGCUGUCACAACCUGACCGCCCAAUCCGCUCUUGAACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCUCCGUGAACUCCGACCCCAGUCCGAAUUCCAUGUCCAGGAA (((.(((((.((((.....)))).)))...)))))(((((.(((......((((((((.(((((....)))))..)))))))).(((.((.(((....))).)).)))..))).))))). ( -40.20) >DroSim_CAF1 21934 120 - 1 GCGGGAGGCGGUCACAACCUGACCGCCCAAUCCGCUCUUGAACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCUCCGUGAACUCCGACCCCAGUCCGAAUUCCAUGUCCAGGAA (((((.((((((((.....))))))))...)))))(((((.(((......((((((((.(((((....)))))..)))))))).(((.((.(((....))).)).)))..))).))))). ( -49.50) >DroEre_CAF1 23837 120 - 1 GCGGGAGGCGGUCACAACCUGACCGCCCAGACCGCUCUUGAAUAGAUCCUCGGAGCUGAGCUUACAGCUCGGCACCAGCUCCGUGAACUCCGACCCCAGUCCGAACUCCAUGUCCAGGAA ((((..((((((((.....))))))))....))))(((((.(((..((..(((((((((((.....))))(....)))))))).))..((.(((....))).))......))).))))). ( -47.80) >DroYak_CAF1 24219 120 - 1 GCGGGAGGCGGUCACAACCUGACCGCCCAGUCCGCUCUUGAAGAGAUCCUCCGAGCUGAGCUUACAGCUCGGCACCAGCUCCGUGAACUCCGACCCCAGUCCGAACUCCAUGUCCAGGAA (((((.((((((((.....))))))))...)))))(((((..(((.....((((((((......))))))))......)))((((...((.(((....))).))....))))..))))). ( -49.00) >consensus GCGGGAGGCGGUCACAACCUGACCGCCCAAUCCGCUCUUGAACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCUCCGUGAACUCCGACCCCAGUCCGAAUUCCAUGUCCAGGAA (((((.((((((((.....))))))))...)))))(((((.(((......((((((((.(((((....)))))..)))))))).(((.((.(((....))).)).)))..))).))))). (-42.34 = -43.98 + 1.64)

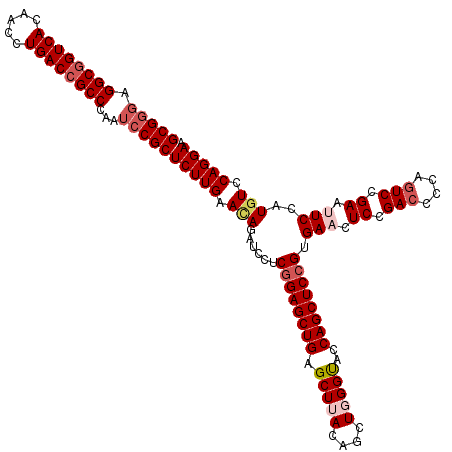
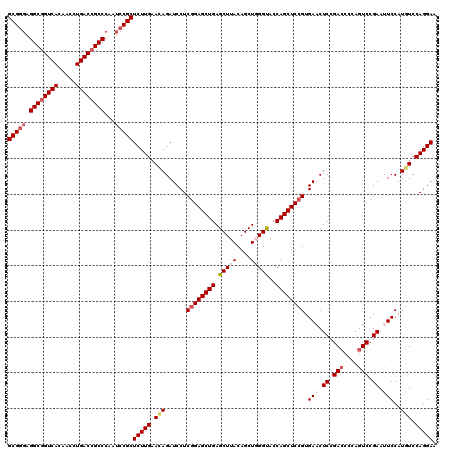
| Location | 12,085,095 – 12,085,215 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.75 |
| Mean single sequence MFE | -49.82 |
| Consensus MFE | -42.94 |
| Energy contribution | -43.98 |
| Covariance contribution | 1.04 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.28 |
| Structure conservation index | 0.86 |
| SVM decision value | 0.04 |
| SVM RNA-class probability | 0.552191 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 12085095 120 - 27905053 CCAUGAUGUGGGCCGGGUGCUCCAGUUCGUUGUCGUGCAGGCGGGAGGCGGUCACAACCUGACCGCCCAAUCCGCUCUUGAACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCU ......(((((((.....(((((.((((((......)).((((((.((((((((.....))))))))...))))))...)))).(.....))))))...)))))))(((((...))))). ( -50.20) >DroSec_CAF1 21786 120 - 1 CCAUGAUGUGGGCCGGGUGCUCCAGUUCGUUGUCAUGCAGGCGCGAGGCUGUCACAACCUGACCGCCCAAUCCGCUCUUGAACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCU ......(((((((.(((((...(((...(((((...((((.(....).)))).))))))))..)))))..((.(((((.((........))))))).)))))))))(((((...))))). ( -44.80) >DroSim_CAF1 21974 120 - 1 CCAUGAUGUGGGCCGGGUGCUCCAGUUCGUUGUCGUGCAGGCGGGAGGCGGUCACAACCUGACCGCCCAAUCCGCUCUUGAACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCU ......(((((((.....(((((.((((((......)).((((((.((((((((.....))))))))...))))))...)))).(.....))))))...)))))))(((((...))))). ( -50.20) >DroEre_CAF1 23877 120 - 1 CCAUGAUGUGGGCCGGCUGCUCCAGUUCGUUGUCGUGCAGGCGGGAGGCGGUCACAACCUGACCGCCCAGACCGCUCUUGAAUAGAUCCUCGGAGCUGAGCUUACAGCUCGGCACCAGCU .((.((.((((.(((.((((..(((....)))....)))).)))(.((((((((.....)))))))))...)))))).))...........((.(((((((.....))))))).)).... ( -50.30) >DroYak_CAF1 24259 120 - 1 CCAUGAUGUGGGCCGGCUGCUCCAGUUCGUUGUCGUGCAGGCGGGAGGCGGUCACAACCUGACCGCCCAGUCCGCUCUUGAAGAGAUCCUCCGAGCUGAGCUUACAGCUCGGCACCAGCU ........((((((((((((((.((((((..(((...((((((((.((((((((.....))))))))...))))))..))....)))....))))))))))....))).)))).)))... ( -53.60) >consensus CCAUGAUGUGGGCCGGGUGCUCCAGUUCGUUGUCGUGCAGGCGGGAGGCGGUCACAACCUGACCGCCCAAUCCGCUCUUGAACAGAUCCUCGGAGCUGAGCUUACAGCUGGGUACCAGCU ......(((((((.....(((((((.((....(((....((((((.((((((((.....))))))))...))))))..)))...))..)).)))))...)))))))(((((...))))). (-42.94 = -43.98 + 1.04)


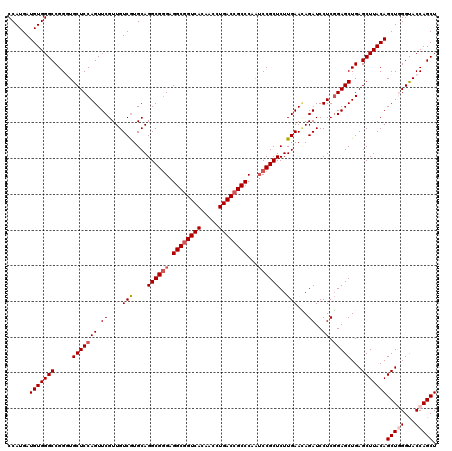
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:34:04 2006