
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 11,918,730 – 11,918,921 |
| Length | 191 |
| Max. P | 0.819260 |

| Location | 11,918,730 – 11,918,830 |
|---|---|
| Length | 100 |
| Sequences | 6 |
| Columns | 103 |
| Reading direction | forward |
| Mean pairwise identity | 82.64 |
| Mean single sequence MFE | -39.47 |
| Consensus MFE | -26.39 |
| Energy contribution | -29.58 |
| Covariance contribution | 3.20 |
| Combinations/Pair | 1.18 |
| Mean z-score | -1.80 |
| Structure conservation index | 0.67 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.589800 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
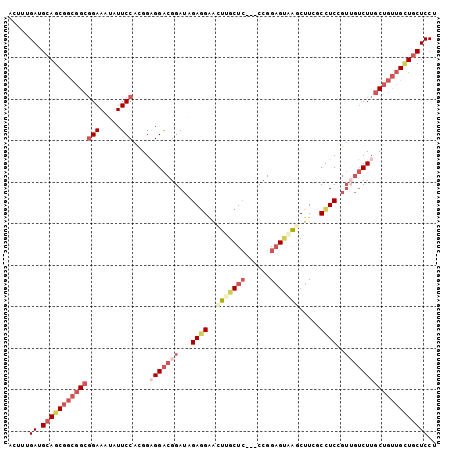
>3R_DroMel_CAF1 11918730 100 + 27905053 ACUUUGAUGCAGCGGCGGCGGAAAUAUUCCACGGAGGACGGAUAGAGGAACUGGCUC---CCGGAGUAAGCUUCGCCUCCGUUGUCUUGCUGUUGUUGCUCCU .....((.((((((((((((((.....)))..(((.((((((..((((.....((((---...))))...))))...)))))).))).))))))))))))).. ( -40.00) >DroSec_CAF1 23565 100 + 1 AUUUUGAUGCAGCGGCGGCGGAAAUAUUCCACGGAGGACGGAUGGAGGAACUUGCUC---CCGGUGUGAGCUUUGCCUCGGUUGUCUUGCUGUUGCUGCUCCU .....((.((((((((((((((.....)))....((((((.((.((((.....((((---.......))))....)))).))))))))))))))))))))).. ( -42.00) >DroSim_CAF1 23497 100 + 1 ACUUUGAUGCAGCGGCGGCGGAAAUAUUCCACGGAGGACGGAUGGAGGAACUUGCUC---CCGGAGUAAGCUUUGCCUCGGUUGUCUUACUGUUGCUGCUCCU .....((.((((((((((.(((.....)))....((((((.((.((((..(((((((---...))))))).....)))).)))))))).)))))))))))).. ( -38.80) >DroEre_CAF1 23199 100 + 1 ACUUUGAUGCAGCGGCGGCGGAAAUAUUCCAUGGCGGACGGAUAGAGGAACUUGCUC---CCGGAGUAACCUUCGCUUCCGUUGUCUUGCUGUUGCUACUCCU ..........((((((((((((.....)))..((((.(((((..((((...((((((---...)))))).))))...)))))))))..)))))))))...... ( -34.90) >DroYak_CAF1 23555 100 + 1 ACUUUGAUGCAGCGGCGGCGGAAAUAUUCCACGGAGGACGGAUUGAAGGACUUGCUA---CCGGAGCAACCUUGGCCUCCGUGGUCUUGCUGUUGCUGCUCCU .....((.((((((((((((((......(((((((((......(.((((..(((((.---....))))))))).)))))))))))).)))))))))))))).. ( -44.10) >DroAna_CAF1 41005 97 + 1 ACUUGGAGGCAGCUAUCGCCGAACUGUUCCAGGGCGGGCGGAUAGAGGCGUUGGCUCCAGCCGGAGAAGGCUUGCCCUC------CUUGCUUCCGGUGCUCCU ....(((((.(((((.((((...((((((......)))))).....)))).))))).).(((((((((((........)------))).).)))))).)))). ( -37.00) >consensus ACUUUGAUGCAGCGGCGGCGGAAAUAUUCCACGGAGGACGGAUAGAGGAACUUGCUC___CCGGAGUAAGCUUCGCCUCCGUUGUCUUGCUGUUGCUGCUCCU .....((.((((((((((((((.....)))....(((((((...((((..(((((((......))))))).....))))..)))))))))))))))))))).. (-26.39 = -29.58 + 3.20)


| Location | 11,918,830 – 11,918,921 |
|---|---|
| Length | 91 |
| Sequences | 6 |
| Columns | 91 |
| Reading direction | forward |
| Mean pairwise identity | 85.13 |
| Mean single sequence MFE | -36.25 |
| Consensus MFE | -28.44 |
| Energy contribution | -29.22 |
| Covariance contribution | 0.78 |
| Combinations/Pair | 1.19 |
| Mean z-score | -2.59 |
| Structure conservation index | 0.78 |
| SVM decision value | 0.67 |
| SVM RNA-class probability | 0.819260 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
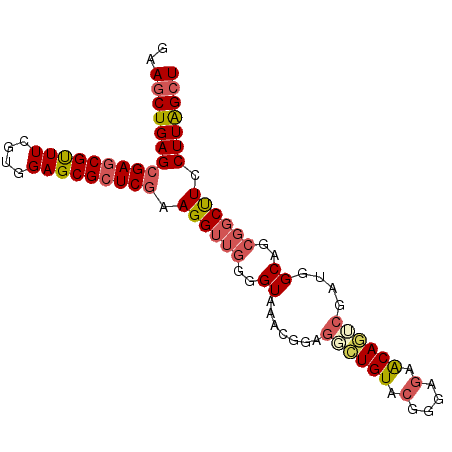
>3R_DroMel_CAF1 11918830 91 + 27905053 GAAGCUGAGCGAGCGUUUCGUGGAGCGCUCGAAGGUUGGGGUAAAAGGAGGCUGUACGGGAGAGCAGUCGAUGGCAGCGGCUUCCUUAGCU ..((((((((((((((((....))))))))).((((((..((....(..((((((.(....).))))))..).))..)))))).))))))) ( -37.30) >DroSec_CAF1 23665 91 + 1 GAAGCUGAGCGAGCGUUUCGUGGAGCGCUCGAAGGUUGGGGUAAACGGAGGCUGUACGGGAGAGCAGUCAAUGGCAGCGGCUUCCUUAGCU ..((((((((((((((((....))))))))).((((((..((...((..((((((.(....).))))))..))))..)))))).))))))) ( -36.50) >DroSim_CAF1 23597 91 + 1 GAAGCUGAGCGAGCGUUUCGUGGAGCGCUCGAAGGUUGGGGUAAACGGAGGCUGUACGGGAGAGCAGUCGAUGGCAGCGGCUUCCUUAGCU ..((((((((((((((((....))))))))).((((((..((...((..((((((.(....).))))))..))))..)))))).))))))) ( -37.50) >DroEre_CAF1 23299 91 + 1 GAAACUGAGCGAGCGUUUUGUGGAGCGCUCGAAGGUUGGGGUAAAUGGAGGCUGUACUGGCGAACAGGCGAUGGCAGCGGCUUCCUUAGCU ..((((...(((((((((....)))))))))..))))..(((.((.(((((((((.(((.....)))((....)).))))))))))).))) ( -36.60) >DroYak_CAF1 23655 91 + 1 GAAGCUGAGCGAGCGUUUUGUGGAGCGCUCGAAGGUUGGUGUAGACGGAGUCUGUACGGGAGAACAAGCGAUGGCAGCGGCUUCCUUAGCU ..((((((((((((((((....))))))))).((((((.(((...((..(..(((.(....).)))..)..))))).)))))).))))))) ( -34.30) >DroAna_CAF1 41102 88 + 1 GAAGCUGAGCGAUCGCUUCGUGGAGCGUUCAAAGGUGGGUGUUACGGGUAUGUGUGGAGCAACACAUCCUAAGGCAG---CCUCCUUGGCU ..((((....((.(((((....))))).)).((((.((.((((..(((.((((((......)))))))))..)))).---)).)))))))) ( -35.30) >consensus GAAGCUGAGCGAGCGUUUCGUGGAGCGCUCGAAGGUUGGGGUAAACGGAGGCUGUACGGGAGAACAGUCGAUGGCAGCGGCUUCCUUAGCU ..((((((((((((((((....))))))))).((((((..((.......((((((.(....).))))))....))..)))))).))))))) (-28.44 = -29.22 + 0.78)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:30:49 2006