
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
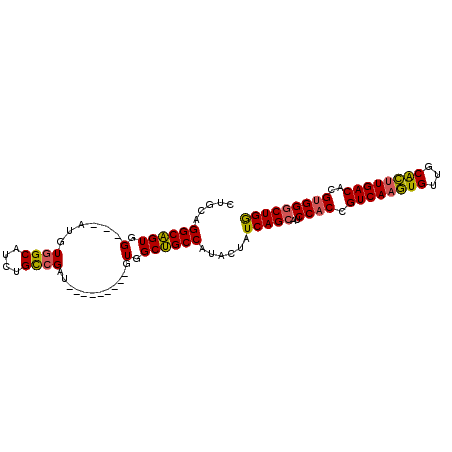
| Location | 11,738,790 – 11,738,917 |
| Length | 127 |
| Max. P | 0.820826 |

| Location | 11,738,790 – 11,738,880 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | forward |
| Mean pairwise identity | 84.20 |
| Mean single sequence MFE | -37.53 |
| Consensus MFE | -30.87 |
| Energy contribution | -30.57 |
| Covariance contribution | -0.30 |
| Combinations/Pair | 1.18 |
| Mean z-score | -2.11 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.68 |
| SVM RNA-class probability | 0.820826 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11738790 90 + 27905053 CUGCAGGCAGUGGAGGAUGUGGCAUCUGCCGAU---------GUGGCUGCCAUACUAUCAGCAUCCACCGUCAAGUGUUGCACGUGACACGUGGGCUGG .....((((((.(......((((....))))..---------.).)))))).......((((..((((.((((.(((...))).))))..)))))))). ( -32.70) >DroSec_CAF1 71561 90 + 1 CUGCAGGCAGUGGAUGAUGUGGCUUCUGCCGAU---------GUGGCUGCCAUACUAUCAGCUUCCACCGUCAAGUGUUGCACUUGACACGUGGGCUGG .....((((((.(......((((....))))..---------.).)))))).......((((..((((.((((((((...))))))))..)))))))). ( -37.00) >DroSim_CAF1 73328 90 + 1 CUGCAGGCAGUGGAGGAUGUGGCAUCUGCCGAU---------GUGGCUGCCAUACUAUCAGCAUCCACCGUCAAGUGUUGCACUUGACACGUGGGCUGG .....((((((.(......((((....))))..---------.).)))))).......((((..((((.((((((((...))))))))..)))))))). ( -36.80) >DroEre_CAF1 72038 87 + 1 CGGCAGGCAGUGG---AUGUGGCAUCUGCCGAU---------GUGGCUGCCACACCAUCAGCAUCCACCGUCAAGUGUGGCACUUGACACGUGGGCUGG ((((..((.((((---.(((((((.(..(....---------)..).)))))))))))..))..((((.((((((((...))))))))..)))))))). ( -41.50) >DroYak_CAF1 75579 87 + 1 CGGCAGGCAGUGG---AUGUGGCAUCUGCCGAU---------GUGGCUGCCAUGCUAUCAGCAUCCACCGUCAAGUGUUGCACUUGACACGUGGGCUGG .((((((((((.(---...((((....))))..---------.).)))))).))))..((((..((((.((((((((...))))))))..)))))))). ( -43.20) >DroPer_CAF1 75709 96 + 1 CUGCAGGCUGUUG---AUGUUGCCGCUGUUGAUGAGUCCGCAGUUGCGGCCAGAGUAUCAGUGUCCACCGUCAAAUGCUGCGUUUGACAUGGGGCCUGA ...((((((.(((---(((((((.(((((.((....)).))))).)))))......)))))...(((..((((((((...)))))))).))))))))). ( -34.00) >consensus CUGCAGGCAGUGG___AUGUGGCAUCUGCCGAU_________GUGGCUGCCAUACUAUCAGCAUCCACCGUCAAGUGUUGCACUUGACACGUGGGCUGG .....((((((.(......((((....))))............).))))))......(((((..((((.((((((((...))))))))..))))))))) (-30.87 = -30.57 + -0.30)


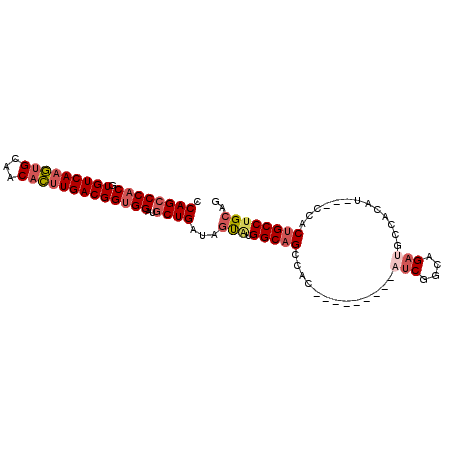
| Location | 11,738,790 – 11,738,880 |
|---|---|
| Length | 90 |
| Sequences | 6 |
| Columns | 99 |
| Reading direction | reverse |
| Mean pairwise identity | 84.20 |
| Mean single sequence MFE | -34.36 |
| Consensus MFE | -25.58 |
| Energy contribution | -26.72 |
| Covariance contribution | 1.14 |
| Combinations/Pair | 1.11 |
| Mean z-score | -2.62 |
| Structure conservation index | 0.74 |
| SVM decision value | 0.56 |
| SVM RNA-class probability | 0.782769 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11738790 90 - 27905053 CCAGCCCACGUGUCACGUGCAACACUUGACGGUGGAUGCUGAUAGUAUGGCAGCCAC---------AUCGGCAGAUGCCACAUCCUCCACUGCCUGCAG .((((((((.(((((.(((...))).)))))))))..))))...(((.(((((((..---------...))).((((...))))......))))))).. ( -31.40) >DroSec_CAF1 71561 90 - 1 CCAGCCCACGUGUCAAGUGCAACACUUGACGGUGGAAGCUGAUAGUAUGGCAGCCAC---------AUCGGCAGAAGCCACAUCAUCCACUGCCUGCAG .((((((((.(((((((((...)))))))))))))..))))...(((.(((((....---------...(((....)))..........)))))))).. ( -34.43) >DroSim_CAF1 73328 90 - 1 CCAGCCCACGUGUCAAGUGCAACACUUGACGGUGGAUGCUGAUAGUAUGGCAGCCAC---------AUCGGCAGAUGCCACAUCCUCCACUGCCUGCAG .((((((((.(((((((((...)))))))))))))..))))...(((.(((((((..---------...))).((((...))))......))))))).. ( -35.50) >DroEre_CAF1 72038 87 - 1 CCAGCCCACGUGUCAAGUGCCACACUUGACGGUGGAUGCUGAUGGUGUGGCAGCCAC---------AUCGGCAGAUGCCACAU---CCACUGCCUGCCG .((((((((.(((((((((...)))))))))))))..)))).(((((((((((((..---------...)))...))))))).---))).......... ( -40.00) >DroYak_CAF1 75579 87 - 1 CCAGCCCACGUGUCAAGUGCAACACUUGACGGUGGAUGCUGAUAGCAUGGCAGCCAC---------AUCGGCAGAUGCCACAU---CCACUGCCUGCCG .((((((((.(((((((((...)))))))))))))..))))...(((.(((((....---------...(((....)))....---...)))))))).. ( -37.54) >DroPer_CAF1 75709 96 - 1 UCAGGCCCCAUGUCAAACGCAGCAUUUGACGGUGGACACUGAUACUCUGGCCGCAACUGCGGACUCAUCAACAGCGGCAACAU---CAACAGCCUGCAG .(((((.((((((((((.......)))))).))))....((((......(((((...((..((....))..)))))))...))---))...)))))... ( -27.30) >consensus CCAGCCCACGUGUCAAGUGCAACACUUGACGGUGGAUGCUGAUAGUAUGGCAGCCAC_________AUCGGCAGAUGCCACAU___CCACUGCCUGCAG .((((((((.(((((((((...)))))))))))))..))))...(((.(((((.............(((....))).............)))))))).. (-25.58 = -26.72 + 1.14)

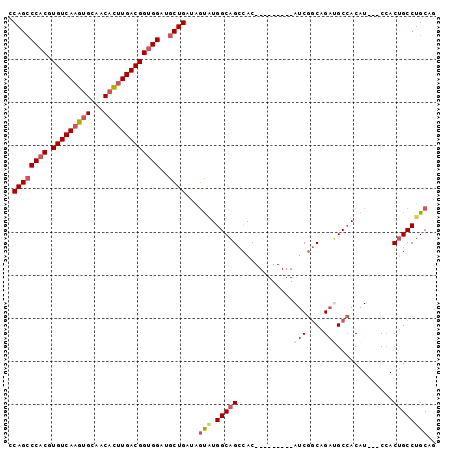
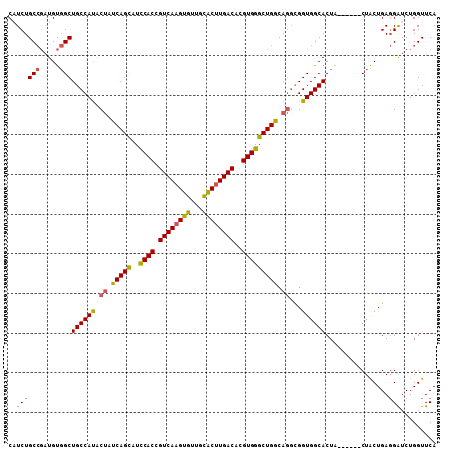
| Location | 11,738,812 – 11,738,917 |
|---|---|
| Length | 105 |
| Sequences | 6 |
| Columns | 108 |
| Reading direction | forward |
| Mean pairwise identity | 87.60 |
| Mean single sequence MFE | -38.90 |
| Consensus MFE | -31.92 |
| Energy contribution | -31.65 |
| Covariance contribution | -0.27 |
| Combinations/Pair | 1.23 |
| Mean z-score | -1.79 |
| Structure conservation index | 0.82 |
| SVM decision value | 0.06 |
| SVM RNA-class probability | 0.563931 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11738812 105 + 27905053 CAUCUGCCGAUGUGGCUGCCAUACUAUCAGCAUCCACCGUCAAGUGUUGCACGUGACACGUGGGCUGGCAGGCGGUGGCACUACU---ACUACUGAGGAUCUGGUUCA .....((((((((((.((((((.((..((((..((((.((((.(((...))).))))..))))))))..))...)))))))))).---.((....)).))).)))... ( -35.70) >DroSec_CAF1 71583 102 + 1 CUUCUGCCGAUGUGGCUGCCAUACUAUCAGCUUCCACCGUCAAGUGUUGCACUUGACACGUGGGCUGGCAGGCGGUGGCACUA------CUACUGAGGAUCUGGUUCA ((((.......((((.((((((.((..((((..((((.((((((((...))))))))..))))))))..))...)))))))))------)....)))).......... ( -39.50) >DroSim_CAF1 73350 102 + 1 CAUCUGCCGAUGUGGCUGCCAUACUAUCAGCAUCCACCGUCAAGUGUUGCACUUGACACGUGGGCUGGCAGGCGGUGGCACUA------CUACUGAGGAUCUGGUUCA .((((......((((.((((((.((..((((..((((.((((((((...))))))))..))))))))..))...)))))))))------)......))))........ ( -39.90) >DroEre_CAF1 72057 102 + 1 CAUCUGCCGAUGUGGCUGCCACACCAUCAGCAUCCACCGUCAAGUGUGGCACUUGACACGUGGGCUGGCAGGCGGUGGCACUU------CUACUGAGGAUCUGGUUCA .....((((((((.((((((......(((((..((((.((((((((...))))))))..)))))))))..)))))).)).(((------(....))))))).)))... ( -39.10) >DroYak_CAF1 75598 108 + 1 CAUCUGCCGAUGUGGCUGCCAUGCUAUCAGCAUCCACCGUCAAGUGUUGCACUUGACACGUGGGCUGGCAGGCGGUGGCACUACUCGUACUACUGAGGAUCUGGUUCA .....((((((((.((((((.(((...((((..((((.((((((((...))))))))..))))))))))))))))).))....((((......)))).))).)))... ( -42.70) >DroAna_CAF1 71415 102 + 1 CUUCUGC---UGCUGCUGCCAUACUUUCAGUAUUCACUGUCAAGUUCUGGGCUUGACAUGUGGGCUGACAAUUGGUGGCACUGCC---ACUGCUGAUGGUCUGGUUCA .....((---....)).((((.(((.(((((.(((((((((((((.....)))))))).))))).........((((((...)))---)))))))).))).))))... ( -36.50) >consensus CAUCUGCCGAUGUGGCUGCCAUACUAUCAGCAUCCACCGUCAAGUGUUGCACUUGACACGUGGGCUGGCAGGCGGUGGCACUA______CUACUGAGGAUCUGGUUCA ..((((((.....)))((((((.((.(((((..((((.((((((((...))))))))..))))))))).))...))))))................)))......... (-31.92 = -31.65 + -0.27)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:29:24 2006