
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 11,408,589 – 11,408,741 |
| Length | 152 |
| Max. P | 0.999991 |

| Location | 11,408,589 – 11,408,707 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.12 |
| Mean single sequence MFE | -32.54 |
| Consensus MFE | -26.91 |
| Energy contribution | -27.91 |
| Covariance contribution | 1.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.42 |
| Structure conservation index | 0.83 |
| SVM decision value | 0.11 |
| SVM RNA-class probability | 0.586326 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11408589 118 + 27905053 GCGGACGUGGACAUGGGAUC--GAGUUUGACUAAUAAAGGUAACCAGAAAUUAACAUAUUCCCAGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAGCAGCAUGAGGCCA ..((.(..(((..(((((((--..(((...((.....))..)))..))...........)))))..)))(((((.(((((((((((.........))))).))))))..))))).).)). ( -31.40) >DroSec_CAF1 156215 118 + 1 UCGGACGUGGACAUGGUAUC--GGGUUUGACUAAUAAAGGCAACCAGAAAUUAACAUAUUCCCUGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAGCAGCAUGAGGCCA ((((((.(((........))--).))))))........(((...(((.(((......)))..)))....(((((.(((((((((((.........))))).))))))..)))))..))). ( -30.10) >DroSim_CAF1 147687 118 + 1 UCGGACGUGGACAUGGGAUC--GGGUUUGACUAAUAAAGGUAACCAGAAAUUAACAUAUUCCCAGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAGCAGCAUGAGGCCA ..((.(..(((..(((((((--.((((...((.....))..)))).))...........)))))..)))(((((.(((((((((((.........))))).))))))..))))).).)). ( -34.50) >DroEre_CAF1 143921 118 + 1 UCGGACGUGGACAUGGGAUC--GGGUUCGACUAAUAAAGGUAACCAGAAAUUAACAUAUUCCCAGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAGCAGCAUGAGGCCA ..((.(..(((..(((((((--.((((..(((......))))))).))...........)))))..)))(((((.(((((((((((.........))))).))))))..))))).).)). ( -34.30) >DroYak_CAF1 147747 120 + 1 UCGGACGUGGACAUGGGAUCUUUGCUCCGACUAAUAAAGGUAACCAGAAAUUAACAUAUUCCCAGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAUCAGCAUGCGGCCG .(((.((.(((..((((((((.....((..........)).....)))...........)))))..)))(((((..((((((((((.........))))).)))))...))))))).))) ( -32.40) >consensus UCGGACGUGGACAUGGGAUC__GGGUUUGACUAAUAAAGGUAACCAGAAAUUAACAUAUUCCCAGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAGCAGCAUGAGGCCA ..((.(..(((..(((((.....((((...((.....))..))))..............)))))..)))(((((..((((((((((.........))))).)))))...))))).).)). (-26.91 = -27.91 + 1.00)

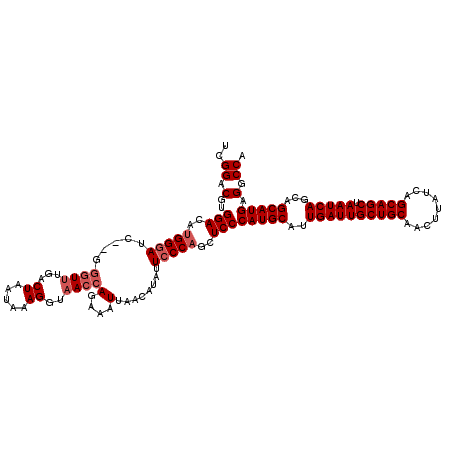

| Location | 11,408,589 – 11,408,707 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.12 |
| Mean single sequence MFE | -36.08 |
| Consensus MFE | -32.28 |
| Energy contribution | -32.08 |
| Covariance contribution | -0.20 |
| Combinations/Pair | 1.07 |
| Mean z-score | -1.71 |
| Structure conservation index | 0.89 |
| SVM decision value | 1.78 |
| SVM RNA-class probability | 0.977129 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11408589 118 - 27905053 UGGCCUCAUGCUGCUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCUGGGAAUAUGUUAAUUUCUGGUUACCUUUAUUAGUCAAACUC--GAUCCCAUGUCCACGUCCGC .((((((((((...(((((.(((((.........))))))))))..))))))).)))((((....(((.....(((((.......)))))...)))..--..)))).............. ( -34.90) >DroSec_CAF1 156215 118 - 1 UGGCCUCAUGCUGCUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCAGGGAAUAUGUUAAUUUCUGGUUGCCUUUAUUAGUCAAACCC--GAUACCAUGUCCACGUCCGA ..(((((((((...(((((.(((((.........))))))))))..))))))).))..(((.((((......((.((((..((.....))...)))).--))...)))))))........ ( -35.10) >DroSim_CAF1 147687 118 - 1 UGGCCUCAUGCUGCUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCUGGGAAUAUGUUAAUUUCUGGUUACCUUUAUUAGUCAAACCC--GAUCCCAUGUCCACGUCCGA .((((((((((...(((((.(((((.........))))))))))..))))))).))).(((.((((......((.((((..((.....))...)))).--))...)))))))........ ( -34.80) >DroEre_CAF1 143921 118 - 1 UGGCCUCAUGCUGCUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCUGGGAAUAUGUUAAUUUCUGGUUACCUUUAUUAGUCGAACCC--GAUCCCAUGUCCACGUCCGA .((((((((((...(((((.(((((.........))))))))))..))))))).))).(((.((((......((.((((..((.....))...)))).--))...)))))))........ ( -35.90) >DroYak_CAF1 147747 120 - 1 CGGCCGCAUGCUGAUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCUGGGAAUAUGUUAAUUUCUGGUUACCUUUAUUAGUCGGAGCAAAGAUCCCAUGUCCACGUCCGA (((.(((((((...(((((.(((((.........))))))))))..)))))(((..(((((...((((.....(((((.......)))))....))))....)))))..))).)).))). ( -39.70) >consensus UGGCCUCAUGCUGCUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCUGGGAAUAUGUUAAUUUCUGGUUACCUUUAUUAGUCAAACCC__GAUCCCAUGUCCACGUCCGA (((((((((((...(((((.(((((.........))))))))))..)))))))(((..(((((......)))))..)))..........))))........................... (-32.28 = -32.08 + -0.20)

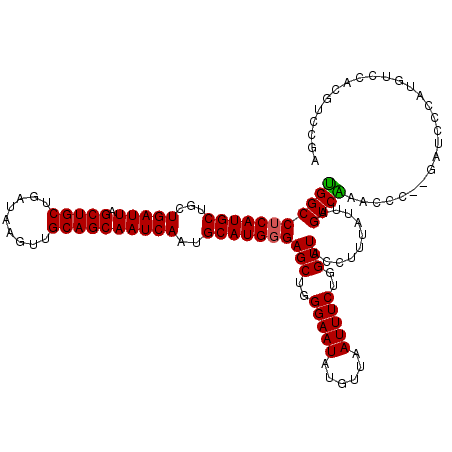
| Location | 11,408,627 – 11,408,741 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 94.85 |
| Mean single sequence MFE | -27.86 |
| Consensus MFE | -26.80 |
| Energy contribution | -26.80 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -3.50 |
| Structure conservation index | 0.96 |
| SVM decision value | 5.60 |
| SVM RNA-class probability | 0.999991 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11408627 114 + 27905053 UAACCAGAAAUUAACAUAUUCCCAGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAGCAGCAUGAGGCCAUAGAC------GAUCUGAAAUCUCAGCACCUAAUUUCCUC ......(((((((...........(((..(((((.(((((((((((.........))))).))))))..))))).))).......------...((((....))))....)))))))... ( -27.60) >DroSec_CAF1 156253 114 + 1 CAACCAGAAAUUAACAUAUUCCCUGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAGCAGCAUGAGGCCAUAGAC------GAUCUGAAAUCUCAGCACCUAAUUUCCUC ......(((((((.........(((..(((((((.(((((((((((.........))))).))))))..))))).))...)))..------...((((....))))....)))))))... ( -27.90) >DroSim_CAF1 147725 114 + 1 UAACCAGAAAUUAACAUAUUCCCAGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAGCAGCAUGAGGCCAUAGAC------UAUCUGAAAUCUCAGCACCUAAUUUCCUC ......(((((((...........(((..(((((.(((((((((((.........))))).))))))..))))).))).......------...((((....))))....)))))))... ( -27.60) >DroEre_CAF1 143959 114 + 1 UAACCAGAAAUUAACAUAUUCCCAGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAGCAGCAUGAGGCCACAGAC------GCUCUGAAAUCUCAGCACCUAAUUUCCUC ......(((((((...........(((..(((((.(((((((((((.........))))).))))))..))))).))).......------(((..(....)..)))...)))))))... ( -28.40) >DroYak_CAF1 147787 120 + 1 UAACCAGAAAUUAACAUAUUCCCAGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAUCAGCAUGCGGCCGUAGACCUGAUAGAUCUGAUAUCUCAGCACCUAAUUUCCUC ......(((((((.((..((..(.((.(.(((((..((((((((((.........))))).)))))...))))).))).)..))..))......((((....))))....)))))))... ( -27.80) >consensus UAACCAGAAAUUAACAUAUUCCCAGCUCCCAUGCAUUGAUUGCUGCAACUUAUCAGCAGCUAAUCAGCAGCAUGAGGCCAUAGAC______GAUCUGAAAUCUCAGCACCUAAUUUCCUC ......(((((((...........(((..(((((..((((((((((.........))))).)))))...))))).)))................((((....))))....)))))))... (-26.80 = -26.80 + 0.00)

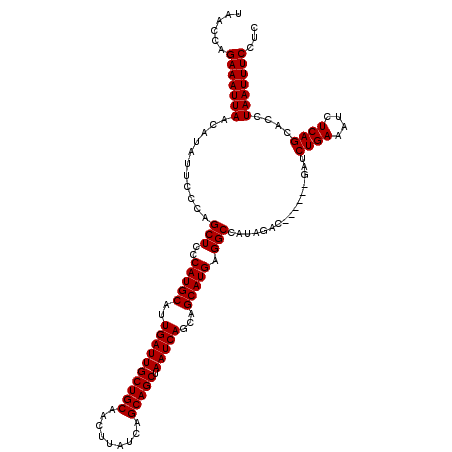

| Location | 11,408,627 – 11,408,741 |
|---|---|
| Length | 114 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.85 |
| Mean single sequence MFE | -38.62 |
| Consensus MFE | -35.98 |
| Energy contribution | -36.22 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.03 |
| Mean z-score | -3.17 |
| Structure conservation index | 0.93 |
| SVM decision value | 5.08 |
| SVM RNA-class probability | 0.999972 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11408627 114 - 27905053 GAGGAAAUUAGGUGCUGAGAUUUCAGAUC------GUCUAUGGCCUCAUGCUGCUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCUGGGAAUAUGUUAAUUUCUGGUUA ..(((((((((((.(((......))))))------.(((.(((((((((((...(((((.(((((.........))))))))))..))))))).)))))))......))))))))..... ( -38.10) >DroSec_CAF1 156253 114 - 1 GAGGAAAUUAGGUGCUGAGAUUUCAGAUC------GUCUAUGGCCUCAUGCUGCUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCAGGGAAUAUGUUAAUUUCUGGUUG ..(((((((((((.(((......))))))------.(((...(((((((((...(((((.(((((.........))))))))))..))))))).))..)))......))))))))..... ( -38.80) >DroSim_CAF1 147725 114 - 1 GAGGAAAUUAGGUGCUGAGAUUUCAGAUA------GUCUAUGGCCUCAUGCUGCUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCUGGGAAUAUGUUAAUUUCUGGUUA ..(((((((((...(((......)))(((------.(((.(((((((((((...(((((.(((((.........))))))))))..))))))).))))))).))).)))))))))..... ( -37.40) >DroEre_CAF1 143959 114 - 1 GAGGAAAUUAGGUGCUGAGAUUUCAGAGC------GUCUGUGGCCUCAUGCUGCUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCUGGGAAUAUGUUAAUUUCUGGUUA ..(((((((((((.(((......))).))------.(((.(((((((((((...(((((.(((((.........))))))))))..))))))).))))))).....)))))))))..... ( -38.40) >DroYak_CAF1 147787 120 - 1 GAGGAAAUUAGGUGCUGAGAUAUCAGAUCUAUCAGGUCUACGGCCGCAUGCUGAUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCUGGGAAUAUGUUAAUUUCUGGUUA ..(((((((((((.((((....)))))))...((..(((.(((((.(((((...(((((.(((((.........))))))))))..))))).).)))))))...)).))))))))..... ( -40.40) >consensus GAGGAAAUUAGGUGCUGAGAUUUCAGAUC______GUCUAUGGCCUCAUGCUGCUGAUUAGCUGCUGAUAAGUUGCAGCAAUCAAUGCAUGGGAGCUGGGAAUAUGUUAAUUUCUGGUUA ..(((((((((...((((....))))..........(((.(((((((((((...(((((.(((((.........))))))))))..))))))).))))))).....)))))))))..... (-35.98 = -36.22 + 0.24)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:24:40 2006