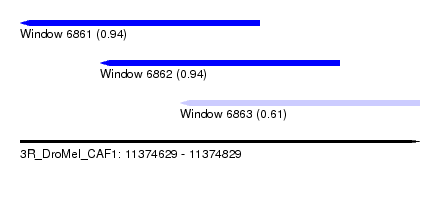
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 11,374,629 – 11,374,829 |
| Length | 200 |
| Max. P | 0.942877 |

| Location | 11,374,629 – 11,374,749 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.67 |
| Mean single sequence MFE | -50.14 |
| Consensus MFE | -47.04 |
| Energy contribution | -47.00 |
| Covariance contribution | -0.04 |
| Combinations/Pair | 1.12 |
| Mean z-score | -1.41 |
| Structure conservation index | 0.94 |
| SVM decision value | 1.24 |
| SVM RNA-class probability | 0.935111 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11374629 120 - 27905053 UCCCUGGAUAGAUGUCGGUCAGGUGGAGGGCGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGCGAACUGCUCAUCUACGAGGGCGACAAUGGUCGCCUGCUGACUAACCGCACAUGGAA (((.(((......(((((.(((((((...((((((((((.((((((((((((.....))))))......))))))..))))))))).).....))))))))))))......).)).))). ( -52.30) >DroSec_CAF1 122267 120 - 1 UCCCUGGAUAGAUGUCGGCCAGGUGGAGGGCGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGUGAACUGCUCAUCUACGAGGGCGACAAUGGUCGCCUGCUGACCAACCGCACAUGGAA (((.(((......((((((.((((((...((((((((((.((((((((((((.....))))))......))))))..))))))))).).....))))))))))))......).)).))). ( -51.60) >DroSim_CAF1 113271 120 - 1 UCCCUGGAUAGAUGUCGGCCAGGUGGAGGGCGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGCGAACUGCUCAUCUACGAGGGCGACAAUGGUCGCCUGCUAACCAACCGCACAUGGAA ..(((((...(....)..)))))(((.((((((((((((.((((((((((((.....))))))......))))))..))))))))(((....))))))).))).(((........))).. ( -48.80) >DroEre_CAF1 109822 120 - 1 UCCCUGGAUAGAUGUCGGCCAGGUGGAAGGUGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGCGAACUGCUCGUCUACGAGGGCGACAACGGUCGCCUCCUGACCAACCGCACCUGGAA ......(((....)))..(((((((...(((((((((((.((((((((((((.....))))))......))))))..))))))))......(((((.....))))).))).))))))).. ( -49.70) >DroYak_CAF1 112360 120 - 1 UCCCUGGAUAGAUGUCGGCCAGGUGGAGGGUGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGCGAACUGCUCAUCUACGAGGGCGACAAUGGUCGCCUUUUGACCAACCGCACGUGGAA (((((((...(....)..))))((((...((((((((((.((((((((((((.....))))))......))))))..)))))))).))..((((((.....)))))).))))....))). ( -48.30) >consensus UCCCUGGAUAGAUGUCGGCCAGGUGGAGGGCGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGCGAACUGCUCAUCUACGAGGGCGACAAUGGUCGCCUGCUGACCAACCGCACAUGGAA (((.(((......(((((.(((((((..(.(((((((((.((((((((((((.....))))))......))))))..)))))))))....)..))))))))))))......).)).))). (-47.04 = -47.00 + -0.04)


| Location | 11,374,669 – 11,374,789 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.00 |
| Mean single sequence MFE | -53.20 |
| Consensus MFE | -52.42 |
| Energy contribution | -51.54 |
| Covariance contribution | -0.88 |
| Combinations/Pair | 1.18 |
| Mean z-score | -1.32 |
| Structure conservation index | 0.99 |
| SVM decision value | 1.33 |
| SVM RNA-class probability | 0.942877 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11374669 120 - 27905053 GGCGGGUGGACAUCCUAGAGGACGCCGGUGAGAGCCUGAGUCCCUGGAUAGAUGUCGGUCAGGUGGAGGGCGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGCGAACUGCUCAUCUACG ....(((.((((((((((.((((.(.(((....))).).))))))))...)))))).)))..((((((((((..(((((..((((((.((....))))))))))))).))))).))))). ( -51.10) >DroSec_CAF1 122307 120 - 1 GGCGGGUGGACAUCCUAGAGGACGCCGGCGAAAGCUUGAGUCCCUGGAUAGAUGUCGGCCAGGUGGAGGGCGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGUGAACUGCUCAUCUACG ....(((.((((((((((.((((.(.(((....))).).))))))))...)))))).)))..(((((((((((((.......))))((((((.....))))))......)))).))))). ( -57.10) >DroSim_CAF1 113311 120 - 1 GACGGGUGGACAUCCUAGAAGACGCUGGCGAGAGUCUGAGUCCCUGGAUAGAUGUCGGCCAGGUGGAGGGCGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGCGAACUGCUCAUCUACG ....(((.((((((((((..(((...(((....)))...))).))))...)))))).)))..((((((((((..(((((..((((((.((....))))))))))))).))))).))))). ( -50.80) >DroEre_CAF1 109862 120 - 1 GGCGGGUGGACAUCCUGGAGGAUGCCGGCGAGAGCCUGAGUCCCUGGAUAGAUGUCGGCCAGGUGGAAGGUGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGCGAACUGCUCGUCUACG (((((((((.(((((....))))))).((..(((((..(((((...(((....)))((((..(((((((....))))..))).)))).)))))...))))).)).....))))))).... ( -52.50) >DroYak_CAF1 112400 120 - 1 GGCGGGUGGACAUCCUAGAGGAUGCCGGCGAGAGCCUGAGUCCCUGGAUAGAUGUCGGCCAGGUGGAGGGUGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGCGAACUGCUCAUCUACG ....(((.((((((((((.((((.(.(((....))).).))))))))...)))))).)))..(((((((((...(((((..((((((.((....)))))))))))))..)))).))))). ( -54.50) >consensus GGCGGGUGGACAUCCUAGAGGACGCCGGCGAGAGCCUGAGUCCCUGGAUAGAUGUCGGCCAGGUGGAGGGCGCCUUCGUCAUGGGCCUGGGCUACUGGCUCAGCGAACUGCUCAUCUACG ....(((.((((((((((.((((.(.(((....))).).))))))))...)))))).)))..(((((((((((((.......))))((((((.....))))))......)))).))))). (-52.42 = -51.54 + -0.88)

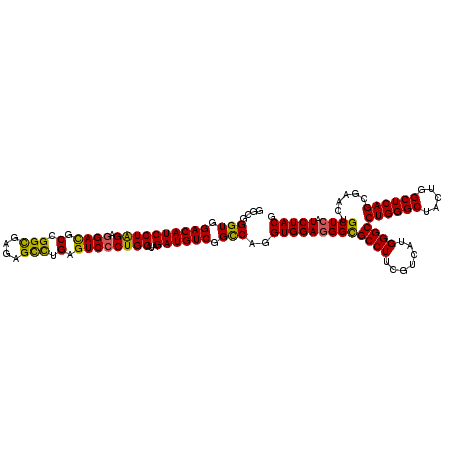
| Location | 11,374,709 – 11,374,829 |
|---|---|
| Length | 120 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 94.00 |
| Mean single sequence MFE | -46.92 |
| Consensus MFE | -44.68 |
| Energy contribution | -44.04 |
| Covariance contribution | -0.64 |
| Combinations/Pair | 1.20 |
| Mean z-score | -1.07 |
| Structure conservation index | 0.95 |
| SVM decision value | 0.16 |
| SVM RNA-class probability | 0.611432 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11374709 120 - 27905053 GAGGUGGAGAUCGAUGUGCUGACCGGCAACAUUCUGAUCAGGCGGGUGGACAUCCUAGAGGACGCCGGUGAGAGCCUGAGUCCCUGGAUAGAUGUCGGUCAGGUGGAGGGCGCCUUCGUC ((((((....((.((...((((((((((....((((.((((..((((....))))....((((.(.(((....))).).)))))))).)))))))))))))))).))...)))))).... ( -46.90) >DroSec_CAF1 122347 120 - 1 GAGGUGGAGAUGGAUGUGCUGACCGGCAACAUUCUGAUCAGGCGGGUGGACAUCCUAGAGGACGCCGGCGAAAGCUUGAGUCCCUGGAUAGAUGUCGGCCAGGUGGAGGGCGCCUUCGUC ......((...(((((((((....))).))))))...)).(((((((.((((((((((.((((.(.(((....))).).))))))))...)))))).)))(((((.....))))).)))) ( -51.40) >DroSim_CAF1 113351 120 - 1 GAAGUGGAGAUGGAUGUGCUGACCGGGAACAUUCUGAUCAGACGGGUGGACAUCCUAGAAGACGCUGGCGAGAGUCUGAGUCCCUGGAUAGAUGUCGGCCAGGUGGAGGGCGCCUUCGUC ......((...((((((.((....))..))))))...)).(((((((.((((((((((..(((...(((....)))...))).))))...)))))).)))(((((.....))))).)))) ( -41.90) >DroEre_CAF1 109902 120 - 1 GAGUUGGAGAUGGAUGUGCUGACCGGCAACAUUCUGAUCAGGCGGGUGGACAUCCUGGAGGAUGCCGGCGAGAGCCUGAGUCCCUGGAUAGAUGUCGGCCAGGUGGAAGGUGCCUUCGUC ..((..((...(((((((((....))).))))))...))..)).(((.(((((((..(.((((.(.(((....))).).)))))..)...)))))).))).....((((....))))... ( -48.00) >DroYak_CAF1 112440 120 - 1 GAGGUGGAGAUGGAUGUUCUGACCGGCAACAUUCUGAUCAGGCGGGUGGACAUCCUAGAGGAUGCCGGCGAGAGCCUGAGUCCCUGGAUAGAUGUCGGCCAGGUGGAGGGUGCCUUCGUC ..(..((((..(.((.((((.((((((.((((.(((.(((((.((.(((.(((((....)))))))(((....)))..).))))))).)))))))..))).))))))).)).)))))..) ( -46.40) >consensus GAGGUGGAGAUGGAUGUGCUGACCGGCAACAUUCUGAUCAGGCGGGUGGACAUCCUAGAGGACGCCGGCGAGAGCCUGAGUCCCUGGAUAGAUGUCGGCCAGGUGGAGGGCGCCUUCGUC ......((...(((((((((....))).))))))...)).(((((((.((((((((((.((((.(.(((....))).).))))))))...)))))).)))(((((.....))))).)))) (-44.68 = -44.04 + -0.64)

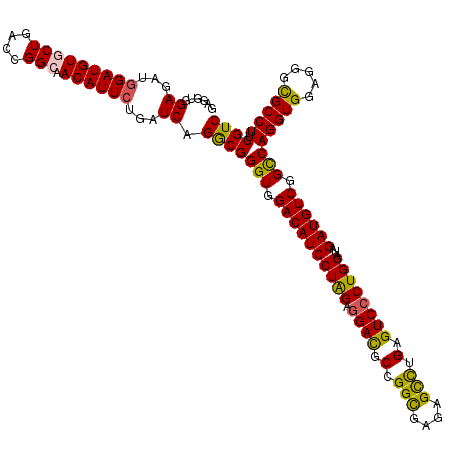
Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:22:55 2006