
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 11,354,255 – 11,354,481 |
| Length | 226 |
| Max. P | 0.963927 |

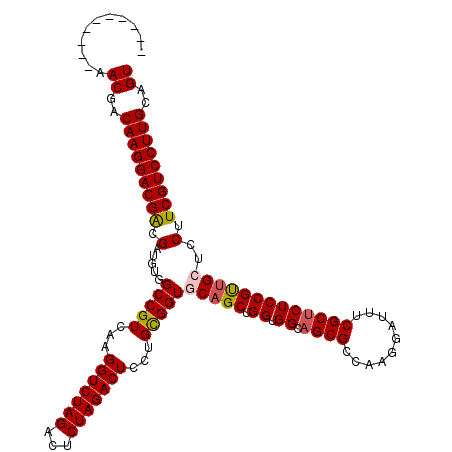
| Location | 11,354,255 – 11,354,365 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 92.28 |
| Mean single sequence MFE | -44.78 |
| Consensus MFE | -36.56 |
| Energy contribution | -36.72 |
| Covariance contribution | 0.16 |
| Combinations/Pair | 1.03 |
| Mean z-score | -2.00 |
| Structure conservation index | 0.82 |
| SVM decision value | 1.49 |
| SVM RNA-class probability | 0.958607 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11354255 110 + 27905053 ACUGCAAGGACGAAGGAGCAACGGAGAGCGAAAUCGUUGGCGCUGCCACCAGCUGCACCGCAGGAGUCUAGAGUCUAGACCUUGACGGCCACAUCGUCGUCCUUGUCGUU---------- ((.((((((((((..(((((...(..((((....))))..)((((....))))))).(((((((.((((((...)))))))))).))).....)).)))))))))).)).---------- ( -43.70) >DroSec_CAF1 101812 110 + 1 ACUGCAAGGACGAAGGAGCAACGGAGAGCGAAAUCCUGGGCGCUGCCACCAGCUGCACCACAGGAGUCUAGAGUCUAGACCUUGACGGCCACAUCGCCGUCCUUGUCGUU---------- ((.((((((((...........(..(((.....(((((((.((.((.....)).)).)).)))))((((((...)))))))))..)(((......))))))))))).)).---------- ( -41.10) >DroSim_CAF1 92588 110 + 1 ACUGCAAGGACGAAGCAGCGACGGAGAGCGAAAUCCUUGGCGCUGCCACCAGCUGCACCGCAGGAGUCUAGAGUCUAGACCUUGACGGCCACAUCGCCGUCCUUGUCGUU---------- ((.(((((((((..(((((...((..((((..(....)..)))).))....))))).(((((((.((((((...)))))))))).))).........))))))))).)).---------- ( -47.00) >DroEre_CAF1 89291 110 + 1 ACUGCAAGGACGAAGGACCAACGGAGUGCGAAUUCCUUGGCGCUGCCACCAGCUGCACCGCAGGAGUCUAGAGUCUAGACCUUGACGGCCACAUCGUCGUCCUUGUCGUU---------- ((.((((((((((..((((((.(((((....))))))))).((((....))))....(((((((.((((((...)))))))))).))).....)).)))))))))).)).---------- ( -45.20) >DroYak_CAF1 91793 120 + 1 ACUGCAAGGACGAAGGACCAGCGGAGAGCGAAAUCCUUGCCGCUGCCACCAGCUGCACCGCAGGAGUCUAGAGUCUAGACCUUGACGGCCACAUCGUCGUCCUUGUCGUUGUGGCGAUGG .(..(((.(((.((((((((((((..((.(....)))..))))))............(((((((.((((((...)))))))))).)))..........))))))))).)))..)...... ( -46.90) >consensus ACUGCAAGGACGAAGGAGCAACGGAGAGCGAAAUCCUUGGCGCUGCCACCAGCUGCACCGCAGGAGUCUAGAGUCUAGACCUUGACGGCCACAUCGUCGUCCUUGUCGUU__________ ((.(((((((((..(......).....((((........((((((....)))).)).(((((((.((((((...)))))))))).))).....))))))))))))).))........... (-36.56 = -36.72 + 0.16)


| Location | 11,354,255 – 11,354,365 |
|---|---|
| Length | 110 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 92.28 |
| Mean single sequence MFE | -44.60 |
| Consensus MFE | -37.86 |
| Energy contribution | -38.10 |
| Covariance contribution | 0.24 |
| Combinations/Pair | 1.08 |
| Mean z-score | -1.56 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.90 |
| SVM RNA-class probability | 0.877798 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11354255 110 - 27905053 ----------AACGACAAGGACGACGAUGUGGCCGUCAAGGUCUAGACUCUAGACUCCUGCGGUGCAGCUGGUGGCAGCGCCAACGAUUUCGCUCUCCGUUGCUCCUUCGUCCUUGCAGU ----------.((..(((((((((.(.....(((((...(((((((...)))))))...)))))(((((.((.((.((((..........)))))))))))))..).)))))))))..)) ( -42.30) >DroSec_CAF1 101812 110 - 1 ----------AACGACAAGGACGGCGAUGUGGCCGUCAAGGUCUAGACUCUAGACUCCUGUGGUGCAGCUGGUGGCAGCGCCCAGGAUUUCGCUCUCCGUUGCUCCUUCGUCCUUGCAGU ----------.((..((((((((((((((.(((.......((((((...))))))(((((.(((((.((.....)).))))))))))....)))...)))))).....))))))))..)) ( -45.60) >DroSim_CAF1 92588 110 - 1 ----------AACGACAAGGACGGCGAUGUGGCCGUCAAGGUCUAGACUCUAGACUCCUGCGGUGCAGCUGGUGGCAGCGCCAAGGAUUUCGCUCUCCGUCGCUGCUUCGUCCUUGCAGU ----------.((..(((((((((.......(((((...(((((((...)))))))...)))))((((((((((....))))).(((........)))...))))).)))))))))..)) ( -46.20) >DroEre_CAF1 89291 110 - 1 ----------AACGACAAGGACGACGAUGUGGCCGUCAAGGUCUAGACUCUAGACUCCUGCGGUGCAGCUGGUGGCAGCGCCAAGGAAUUCGCACUCCGUUGGUCCUUCGUCCUUGCAGU ----------.((..(((((((((.(.((((.((((...(((((((...)))))))...))))))))((((....))))((((((((........))).))))).).)))))))))..)) ( -43.30) >DroYak_CAF1 91793 120 - 1 CCAUCGCCACAACGACAAGGACGACGAUGUGGCCGUCAAGGUCUAGACUCUAGACUCCUGCGGUGCAGCUGGUGGCAGCGGCAAGGAUUUCGCUCUCCGCUGGUCCUUCGUCCUUGCAGU ...........((..(((((((((.(.((((.((((...(((((((...)))))))...))))))))((..((((.(((((........)))))..))))..)).).)))))))))..)) ( -45.60) >consensus __________AACGACAAGGACGACGAUGUGGCCGUCAAGGUCUAGACUCUAGACUCCUGCGGUGCAGCUGGUGGCAGCGCCAAGGAUUUCGCUCUCCGUUGCUCCUUCGUCCUUGCAGU ...........((..(((((((((.(.....(((((...(((((((...)))))))...)))))(((((.((.((.((((..........)))))))))))))..).)))))))))..)) (-37.86 = -38.10 + 0.24)

| Location | 11,354,365 – 11,354,481 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 98.30 |
| Mean single sequence MFE | -42.30 |
| Consensus MFE | -42.10 |
| Energy contribution | -42.10 |
| Covariance contribution | 0.00 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.24 |
| Structure conservation index | 1.00 |
| SVM decision value | 0.49 |
| SVM RNA-class probability | 0.755778 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11354365 116 + 27905053 ----GGCGGUGUCGUCUCCGAUAAGCAUACGCCCUGUGGGCGCGCCAGAUAUUAUAAUACGUGGCCAUUAUCCCGGCUCUGCCGGCGAUAAAUCACACCGGGACAUCUUUGUGUGCCAGC ----((((.(((((....)))))......(((((...)))))))))...............((((..((((((((((...))))).)))))...((((..((....))..)))))))).. ( -41.70) >DroSec_CAF1 101922 116 + 1 ----GGCGGUGUCGACUCCGAUAAGCAUACGCCCUGUGGGCGCGCCAGAUAUUAUAAUACGUGGCCAUUAUCCCGGCUCUGCCGGCGAUAAAUCACACCGGGACAUCUUUGUGUGCCAGC ----((((.(((((....)))))......(((((...)))))))))...............((((..((((((((((...))))).)))))...((((..((....))..)))))))).. ( -42.20) >DroSim_CAF1 92698 116 + 1 ----GGCGGUGUCGACUCCGAUAAGCAUACGCCCUGUGGGCGCGCCAGAUAUUAUAAUACGUGGCCAUUAUCCCGGCUCUGCCGGCGAUAAAUCACACCGGGACAUCUUUGUGUGCCAGC ----((((.(((((....)))))......(((((...)))))))))...............((((..((((((((((...))))).)))))...((((..((....))..)))))))).. ( -42.20) >DroEre_CAF1 89401 116 + 1 ----GGCGGUGUCGACUCCGAUAAGCAUACGCCCUGUGGGCGCGCCAGAUAUUAUAAUACGUGGCCAUUAUCCCGGCUCUGCCGGCGAUAAAUCACACCGGGACAUCUUUGUGUGCCAGC ----((((.(((((....)))))......(((((...)))))))))...............((((..((((((((((...))))).)))))...((((..((....))..)))))))).. ( -42.20) >DroYak_CAF1 91913 120 + 1 CGAUGGCGGUGUCGACUCCGAUAAGCAUACGCCCUGUGGGCGCGCCAGAUAUUAUAAUACGUGGCCAUUAUCCCGGCUCUGCCGGCGAUAAAUCACACCGGGACAUCUUUGUGUGCCAGC ...(((((.(((((....)))))......(((((...))))))))))..............((((..((((((((((...))))).)))))...((((..((....))..)))))))).. ( -43.20) >consensus ____GGCGGUGUCGACUCCGAUAAGCAUACGCCCUGUGGGCGCGCCAGAUAUUAUAAUACGUGGCCAUUAUCCCGGCUCUGCCGGCGAUAAAUCACACCGGGACAUCUUUGUGUGCCAGC ....((((.(((((....)))))......(((((...)))))))))...............((((..((((((((((...))))).)))))...((((..((....))..)))))))).. (-42.10 = -42.10 + 0.00)

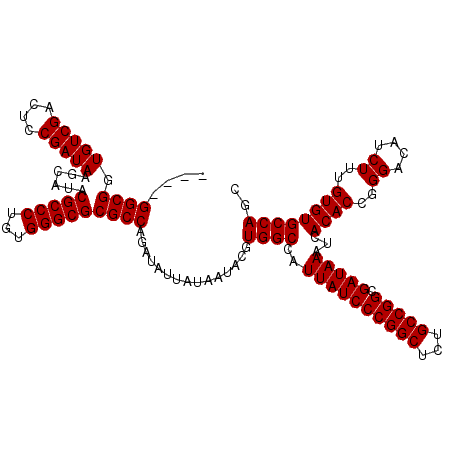
| Location | 11,354,365 – 11,354,481 |
|---|---|
| Length | 116 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 98.30 |
| Mean single sequence MFE | -44.26 |
| Consensus MFE | -43.04 |
| Energy contribution | -43.24 |
| Covariance contribution | 0.20 |
| Combinations/Pair | 1.00 |
| Mean z-score | -1.58 |
| Structure conservation index | 0.97 |
| SVM decision value | 1.56 |
| SVM RNA-class probability | 0.963927 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11354365 116 - 27905053 GCUGGCACACAAAGAUGUCCCGGUGUGAUUUAUCGCCGGCAGAGCCGGGAUAAUGGCCACGUAUUAUAAUAUCUGGCGCGCCCACAGGGCGUAUGCUUAUCGGAGACGACACCGCC---- ((.((.(((......))).))(((((...(((((.(((((...))))))))))..((((.((((....)))).))))((((((...))))))........((....))))))))).---- ( -43.60) >DroSec_CAF1 101922 116 - 1 GCUGGCACACAAAGAUGUCCCGGUGUGAUUUAUCGCCGGCAGAGCCGGGAUAAUGGCCACGUAUUAUAAUAUCUGGCGCGCCCACAGGGCGUAUGCUUAUCGGAGUCGACACCGCC---- ((.((.(((......))).))((((((((((....(((((...)))))(((((..((((.((((....)))).))))((((((...))))))....))))).))))).))))))).---- ( -44.00) >DroSim_CAF1 92698 116 - 1 GCUGGCACACAAAGAUGUCCCGGUGUGAUUUAUCGCCGGCAGAGCCGGGAUAAUGGCCACGUAUUAUAAUAUCUGGCGCGCCCACAGGGCGUAUGCUUAUCGGAGUCGACACCGCC---- ((.((.(((......))).))((((((((((....(((((...)))))(((((..((((.((((....)))).))))((((((...))))))....))))).))))).))))))).---- ( -44.00) >DroEre_CAF1 89401 116 - 1 GCUGGCACACAAAGAUGUCCCGGUGUGAUUUAUCGCCGGCAGAGCCGGGAUAAUGGCCACGUAUUAUAAUAUCUGGCGCGCCCACAGGGCGUAUGCUUAUCGGAGUCGACACCGCC---- ((.((.(((......))).))((((((((((....(((((...)))))(((((..((((.((((....)))).))))((((((...))))))....))))).))))).))))))).---- ( -44.00) >DroYak_CAF1 91913 120 - 1 GCUGGCACACAAAGAUGUCCCGGUGUGAUUUAUCGCCGGCAGAGCCGGGAUAAUGGCCACGUAUUAUAAUAUCUGGCGCGCCCACAGGGCGUAUGCUUAUCGGAGUCGACACCGCCAUCG (.((((..(((....)))...((((((((((....(((((...)))))(((((..((((.((((....)))).))))((((((...))))))....))))).))))).))))))))).). ( -45.70) >consensus GCUGGCACACAAAGAUGUCCCGGUGUGAUUUAUCGCCGGCAGAGCCGGGAUAAUGGCCACGUAUUAUAAUAUCUGGCGCGCCCACAGGGCGUAUGCUUAUCGGAGUCGACACCGCC____ ((.((.(((......))).))((((((((((....(((((...)))))(((((..((((.((((....)))).))))((((((...))))))....))))).))))).)))))))..... (-43.04 = -43.24 + 0.20)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:21:45 2006