
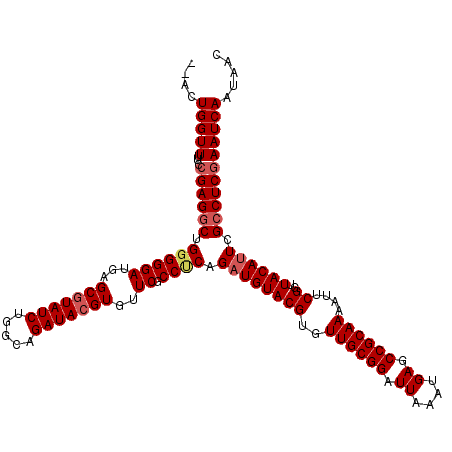
| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
| Location | 11,348,753 – 11,348,871 |
| Length | 118 |
| Max. P | 0.997923 |

| Location | 11,348,753 – 11,348,871 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 95.45 |
| Mean single sequence MFE | -41.18 |
| Consensus MFE | -37.40 |
| Energy contribution | -38.28 |
| Covariance contribution | 0.88 |
| Combinations/Pair | 1.02 |
| Mean z-score | -2.09 |
| Structure conservation index | 0.91 |
| SVM decision value | 2.96 |
| SVM RNA-class probability | 0.997923 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11348753 118 + 27905053 --ACUGGUUUUCCGAGUCUGGGGGAUGAGCAUAUCUGGCAGAUACGUGUUCGCCUCAGAUGUACGUGUUGCGGAUUAAAUGAGCCGCAAAAUUCGGUUACAUUCGCCUCAAAUCAAUAAC --....((...((((((((((((..((((((((((.....)))..))))))))))))))).......((((((.((....)).))))))...))))..)).................... ( -32.30) >DroSec_CAF1 87630 118 + 1 --ACUGGUUUUCCGAGGCUGGGGGAUGAGCGUAUCAGGCAGAUACGUGUUCGCCUCAGAUGUACGUGUUGCGGAUUAAAUGAGCCGCAAAAUUCGGUUACACUCGCCUCGAAUCAAUAAC --..(((((...(((((((((((((...(((((((.....)))))))..)).)))))((((((((..((((((.((....)).))))))....))..)))).)))))))))))))..... ( -41.90) >DroSim_CAF1 86204 118 + 1 --ACUGGUUUUCCGAGGCUGGGGGAUGAGCGUAUCUGGCAGAUACGUGUUCGCCUCAGAUGUACGUGUUGCGGAUUAAAUGAGCCGCAAAAUUCGGUUACAUUCGCCUCGAAUCAAUAAC --..(((((...(((((((((((((...(((((((.....)))))))..)).)))))((((((((..((((((.((....)).))))))....))..)))))).)))))))))))..... ( -42.80) >DroEre_CAF1 83935 119 + 1 GCACUGGUUUUUCGAGGCUG-GGGAUGGGCGUAUCUGGCAGAUACGUGUUCGCCUCAGAUGUACGUGUUGCGGAUUAAAUGAGCCGCAAAAUUCGGUUACAUUCGCCUCGAAUCAAUAAC ..........((((((((((-(((..(.(((((((.....))))))).)...)))))((((((((..((((((.((....)).))))))....))..)))))).))))))))........ ( -43.00) >DroYak_CAF1 86191 118 + 1 --ACUGGUUUGUCGAGGCCGGGGGAUGAGCGUAUCUGGCAGAUACGUGUUCGCCCCAGAUGUACGUGUUGCGGAUUAAAUGAGCCGCAAAAUUCGGUUACAUUCGCCUCGAAUCAAUAAC --..((((...(((((((.((((((...(((((((.....)))))))..)).)))).((((((((..((((((.((....)).))))))....))..)))))).)))))))))))..... ( -45.90) >consensus __ACUGGUUUUCCGAGGCUGGGGGAUGAGCGUAUCUGGCAGAUACGUGUUCGCCUCAGAUGUACGUGUUGCGGAUUAAAUGAGCCGCAAAAUUCGGUUACAUUCGCCUCGAAUCAAUAAC ....(((((...((((((.((((((...(((((((.....)))))))..)).)))).((((((((..((((((.((....)).))))))....))..)))))).)))))))))))..... (-37.40 = -38.28 + 0.88)


| Location | 11,348,753 – 11,348,871 |
|---|---|
| Length | 118 |
| Sequences | 5 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 95.45 |
| Mean single sequence MFE | -32.04 |
| Consensus MFE | -29.86 |
| Energy contribution | -29.38 |
| Covariance contribution | -0.48 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.07 |
| Structure conservation index | 0.93 |
| SVM decision value | -0.04 |
| SVM RNA-class probability | 0.513923 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11348753 118 - 27905053 GUUAUUGAUUUGAGGCGAAUGUAACCGAAUUUUGCGGCUCAUUUAAUCCGCAACACGUACAUCUGAGGCGAACACGUAUCUGCCAGAUAUGCUCAUCCCCCAGACUCGGAAAACCAGU-- ........((((((.....((((..((....((((((..........))))))..))))))((((.((.((...((((((.....))))))....)))).))))))))))........-- ( -25.10) >DroSec_CAF1 87630 118 - 1 GUUAUUGAUUCGAGGCGAGUGUAACCGAAUUUUGCGGCUCAUUUAAUCCGCAACACGUACAUCUGAGGCGAACACGUAUCUGCCUGAUACGCUCAUCCCCCAGCCUCGGAAAACCAGU-- ........((((((((..(((((..((....((((((..........))))))..))))))).((.((.((...((((((.....))))))....)))).))))))))))........-- ( -32.50) >DroSim_CAF1 86204 118 - 1 GUUAUUGAUUCGAGGCGAAUGUAACCGAAUUUUGCGGCUCAUUUAAUCCGCAACACGUACAUCUGAGGCGAACACGUAUCUGCCAGAUACGCUCAUCCCCCAGCCUCGGAAAACCAGU-- ........((((((((((.((((..((....((((((..........))))))..))))))))((.((.((...((((((.....))))))....)))).))))))))))........-- ( -32.10) >DroEre_CAF1 83935 119 - 1 GUUAUUGAUUCGAGGCGAAUGUAACCGAAUUUUGCGGCUCAUUUAAUCCGCAACACGUACAUCUGAGGCGAACACGUAUCUGCCAGAUACGCCCAUCCC-CAGCCUCGAAAAACCAGUGC ..(((((.((((((((((.((((..((....((((((..........))))))..))))))))((.((.((...((((((.....))))))....))))-))))))))))....))))). ( -33.10) >DroYak_CAF1 86191 118 - 1 GUUAUUGAUUCGAGGCGAAUGUAACCGAAUUUUGCGGCUCAUUUAAUCCGCAACACGUACAUCUGGGGCGAACACGUAUCUGCCAGAUACGCUCAUCCCCCGGCCUCGACAAACCAGU-- ....(((..(((((((((.((((..((....((((((..........))))))..))))))))(((((.((...((((((.....))))))....)))))))))))))))))......-- ( -37.40) >consensus GUUAUUGAUUCGAGGCGAAUGUAACCGAAUUUUGCGGCUCAUUUAAUCCGCAACACGUACAUCUGAGGCGAACACGUAUCUGCCAGAUACGCUCAUCCCCCAGCCUCGGAAAACCAGU__ ........(((((((((.(((((..((....((((((..........))))))..))))))))((.((.((...((((((.....))))))....)))).)))))))))).......... (-29.86 = -29.38 + -0.48)


Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:21:17 2006