| Sequence ID | 3R_DroMel_CAF1 |
|---|---|
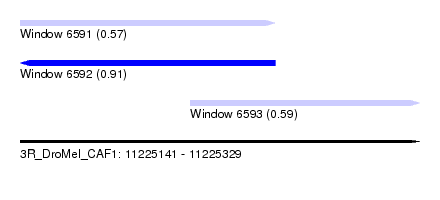
| Location | 11,225,141 – 11,225,329 |
| Length | 188 |
| Max. P | 0.907386 |

| Location | 11,225,141 – 11,225,261 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 93.94 |
| Mean single sequence MFE | -43.80 |
| Consensus MFE | -37.31 |
| Energy contribution | -37.23 |
| Covariance contribution | -0.08 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.54 |
| Structure conservation index | 0.85 |
| SVM decision value | 0.07 |
| SVM RNA-class probability | 0.571000 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
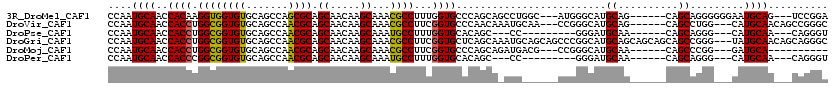
>3R_DroMel_CAF1 11225141 120 + 27905053 GCGCGACUAGCCUGCUCUCUGGUGCUACCUCGGCUGCCACCGGUGGUCUAUUUGGGUCGAAGCAACCGCCCAAGACGGAUCCAAUGCAACCACAAGGUGGUGUGCAGCCAGCGCAGCAAC ((((((.((((((.......)).))))..))((((((((((..((((((.((((((.((.......))))))))..))).))).....(((....))))))).)))))).))))...... ( -47.10) >DroVir_CAF1 26231 120 + 1 GCGCAACUAGCCUGCUCUCUGGUGCUACUUCGGCUGCCACCGGUGGUCUAUUUGGUUCAAAGCAACCACCCAAAACGGAUCCAAUGCAACCACCUGGCGGUGUGCAGCCAACGCAGCAAC (((((.((((........)))))))......((((((((((((((((.....(((((......))))).((.....))..........))))))....)))).))))))...))...... ( -40.20) >DroGri_CAF1 28624 120 + 1 GCGCGACUAGCCUGCUUUCUGGUGCUACUUCGGCUGCCACCGGUGGUCUAUUUGGUUCAAAGCAACCGCCCAAAACGGAUCCAAUGCAACCACCUGGCGGUGUGCAGCCAACGCAGCAAC (((((..((((..(((....)))))))....((((((((((((((((....((((......((....))((.....))..))))....))))))....)))).))))))..))).))... ( -43.10) >DroSec_CAF1 26993 120 + 1 GCGCGACUAGCCUGCUCUCUGGUGCUACCUCGGCUGCCACCGGUGGUCUAUUUGGGUCGAAGCAACCGCCUAAGACGGAUCCAAUGCAACCACAAGUUGGUGUGCAGCCAGCGCAGCAAC ((((((.((((((.......)).))))..))((((((((((((((((....(((((((...((....))(......))))))))....)))))....))))).)))))).))))...... ( -45.90) >DroWil_CAF1 31906 120 + 1 GCGCGACUAGCCUGCUCUCUGGUGCUACUUCGGCUGCCACCGGUGGUCUAUUUGGUUCGAAACAACCACCCAAAACGGAUCCAAUGCAACCACCCGGUGGUGUGCAGCCAACGCAGCAAC (((((..((((((.......)).))))....((((((((((((((((....((((((((................)))).))))....))))))....)))).))))))..))).))... ( -43.69) >DroMoj_CAF1 26929 120 + 1 GGGCAACUAGCCUGCUCUCUGGUGCUACUUCGGCUGCCACCGGUGGUCUAUUUGGUUCGAAGCAACCGCCCAAAACGGAUCCAAUGCAACCACCUGGCGGUGUGCAGCCAACGCAGCAAC ((((.....))))......((.(((......((((((((((((((((....((((((((..((....))......)))).))))....))))))....)))).))))))...))).)).. ( -42.80) >consensus GCGCGACUAGCCUGCUCUCUGGUGCUACUUCGGCUGCCACCGGUGGUCUAUUUGGUUCGAAGCAACCGCCCAAAACGGAUCCAAUGCAACCACCUGGCGGUGUGCAGCCAACGCAGCAAC ((((.(((((........)))))))......((((((((((((((((....((((.((...................)).))))....)))))....))))).))))))...))...... (-37.31 = -37.23 + -0.08)



| Location | 11,225,141 – 11,225,261 |
|---|---|
| Length | 120 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | reverse |
| Mean pairwise identity | 93.94 |
| Mean single sequence MFE | -48.60 |
| Consensus MFE | -45.27 |
| Energy contribution | -45.72 |
| Covariance contribution | 0.44 |
| Combinations/Pair | 1.03 |
| Mean z-score | -1.30 |
| Structure conservation index | 0.93 |
| SVM decision value | 1.05 |
| SVM RNA-class probability | 0.907386 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11225141 120 - 27905053 GUUGCUGCGCUGGCUGCACACCACCUUGUGGUUGCAUUGGAUCCGUCUUGGGCGGUUGCUUCGACCCAAAUAGACCACCGGUGGCAGCCGAGGUAGCACCAGAGAGCAGGCUAGUCGCGC (.((((((.((((((((.(((((((....))).....(((.((..(.((((((((.....))).))))))..)))))..)))))))))).)))))))).).....((.((....)))).. ( -46.10) >DroVir_CAF1 26231 120 - 1 GUUGCUGCGUUGGCUGCACACCGCCAGGUGGUUGCAUUGGAUCCGUUUUGGGUGGUUGCUUUGAACCAAAUAGACCACCGGUGGCAGCCGAAGUAGCACCAGAGAGCAGGCUAGUUGCGC (.((((((.((((((((.((((.((((((....)).))))..........(((((((..((((...))))..))))))))))))))))))).)))))).).....((((.....)))).. ( -49.90) >DroGri_CAF1 28624 120 - 1 GUUGCUGCGUUGGCUGCACACCGCCAGGUGGUUGCAUUGGAUCCGUUUUGGGCGGUUGCUUUGAACCAAAUAGACCACCGGUGGCAGCCGAAGUAGCACCAGAAAGCAGGCUAGUCGCGC (.((((((.((((((((.((((....(((((((((.(..((.....))..)))((((......)))).....))))))))))))))))))).)))))).).....((.((....)))).. ( -49.50) >DroSec_CAF1 26993 120 - 1 GUUGCUGCGCUGGCUGCACACCAACUUGUGGUUGCAUUGGAUCCGUCUUAGGCGGUUGCUUCGACCCAAAUAGACCACCGGUGGCAGCCGAGGUAGCACCAGAGAGCAGGCUAGUCGCGC (.((((((.((((((((.((((.....((((((...((((....(((..((((....)))).)))))))...)))))).)))))))))).)))))))).).....((.((....)))).. ( -45.00) >DroWil_CAF1 31906 120 - 1 GUUGCUGCGUUGGCUGCACACCACCGGGUGGUUGCAUUGGAUCCGUUUUGGGUGGUUGUUUCGAACCAAAUAGACCACCGGUGGCAGCCGAAGUAGCACCAGAGAGCAGGCUAGUCGCGC (.((((((.((((((((.((((....(((((((...((((((((.....))))((.....))...))))...))))))))))))))))))).)))))).).....((.((....)))).. ( -48.80) >DroMoj_CAF1 26929 120 - 1 GUUGCUGCGUUGGCUGCACACCGCCAGGUGGUUGCAUUGGAUCCGUUUUGGGCGGUUGCUUCGAACCAAAUAGACCACCGGUGGCAGCCGAAGUAGCACCAGAGAGCAGGCUAGUUGCCC (.((((((.((((((((.((((....(((((((((.(..((.....))..)))((((......)))).....))))))))))))))))))).)))))).)........(((.....))). ( -52.30) >consensus GUUGCUGCGUUGGCUGCACACCACCAGGUGGUUGCAUUGGAUCCGUUUUGGGCGGUUGCUUCGAACCAAAUAGACCACCGGUGGCAGCCGAAGUAGCACCAGAGAGCAGGCUAGUCGCGC (.((((((.((((((((.((((....(((((((...((((..((((.....))))..........))))...))))))))))))))))))).)))))).).........((.....)).. (-45.27 = -45.72 + 0.44)



| Location | 11,225,221 – 11,225,329 |
|---|---|
| Length | 108 |
| Sequences | 6 |
| Columns | 120 |
| Reading direction | forward |
| Mean pairwise identity | 77.50 |
| Mean single sequence MFE | -38.90 |
| Consensus MFE | -19.42 |
| Energy contribution | -19.08 |
| Covariance contribution | -0.33 |
| Combinations/Pair | 1.15 |
| Mean z-score | -1.89 |
| Structure conservation index | 0.50 |
| SVM decision value | 0.12 |
| SVM RNA-class probability | 0.593553 |
| Prediction | RNA |
Download alignment: ClustalW | MAF
>3R_DroMel_CAF1 11225221 108 + 27905053 CCAAUGCAACCACAAGGUGGUGUGCAGCCAGCGCAGCAACAAGCAAACGCCUUUGGUGCCCAGCAGCCUGGC---AUGGGCAUGCAG------CAGCAGGGGGGAAUGCAG---UCCGGA ((..(((.(((((...)))))((((.....)))).)))....(((..(.((((((.(((...(((((((...---..)))).))).)------)).)))))).)..)))..---...)). ( -41.80) >DroVir_CAF1 26311 108 + 1 CCAAUGCAACCACCUGGCGGUGUGCAGCCAACGCAGCAACAAGCAAACGCCUUCGGUGCCCAACAAAUGCAA---CCGGGCAUGCAG------CAGCCUGG---CAUGCAACAGCCGGGC ....((((...(((.((((((((.......)))).((.....))...))))...)))((((...........---..)))).)))).------..((((((---(........))))))) ( -38.42) >DroPse_CAF1 36913 96 + 1 CCAAUGCAACCACCUGGCGGUGUGCAGCCAACGCAGCAACAAGCAAAUGCCUUUGGUGCACAGC---CC---------GGGAUGCAA------CAGCAGGG---CAUGCAA---CAGGGU ((..((((.((.(((((.(.((((((.((((.(((((.....))...)))..)))))))))).)---))---------))).(((..------..))))).---..)))).---..)).. ( -36.40) >DroGri_CAF1 28704 117 + 1 CCAAUGCAACCACCUGGCGGUGUGCAGCCAACGCAGCAACAAGCAAACGCCUUCGGUGCUCAGCAAAUGCAGCAGCCCGGCAUGCAGCAGCAGCAGCCGGG---UAUGCAACAGCAGGGC ((..(((...((((.((((((((.......)))).((.....))...))))...))))....)))..((((...(((((((.(((.......)))))))))---).))))......)).. ( -44.50) >DroMoj_CAF1 27009 98 + 1 CCAAUGCAACCACCUGGCGGUGUGCAGCCAACGCAGCAACAAGCAAACGCCUUCGGUGCCCAGCAGAUGACG---CCGGGCAUGCAA------CAGCCCGG---GAUGCA---------- ....((((..((((.((((((((.......)))).((.....))...))))...))))(((.((.......)---).((((......------..))))))---).))))---------- ( -33.70) >DroPer_CAF1 37614 96 + 1 CCAAUGCAACCACCCGGCGGUGUGCAGCCAACGCAGCAACAAGCAAAUGCCUUUGGUGCACAGC---CC---------GGGAUGCAA------CAGCAGGG---CAUGCAA---CAGGGU ((..((((.((.(((((.(.((((((.((((.(((((.....))...)))..)))))))))).)---))---------))).(((..------..))))).---..)))).---..)).. ( -38.60) >consensus CCAAUGCAACCACCUGGCGGUGUGCAGCCAACGCAGCAACAAGCAAACGCCUUCGGUGCCCAGCA_ACG______CCGGGCAUGCAA______CAGCAGGG___CAUGCAA___CAGGGC ....((((..((((.((((((((.......)))).((.....))...))))...)))).........................((..........)).........)))).......... (-19.42 = -19.08 + -0.33)

Generated by rnazCluster.pl (part of RNAz 1.0) on Mon Dec 4 11:18:33 2006